摘要:本文以Merck的选择性CDK8抑制剂MSC2530818的衍生物为起点分子,用SPARK对连接臂部分进行了生物等排体替换,重现了Li等人CDK8抑制剂23设计过程中8种连接臂中的7种。比起文献报道的基于结构深度学习结构生成与药化专家设计的两步优化过程,Spark可以从起点分子设计出发,不仅一步直接设计出包括先导化合物1与优选化合物23在内的连接臂化合物,而且还生成许多文献未报到的高分化合物,表现出更高效的生物等排体替换性能。
肖高铿/2023-05-04
肖高铿/2023-05-06:补充了两个计算实验,包括1)扩大数据库搜索范围;2)分子对接打分。
肖高铿/2023-05-08:根据扩大数据库搜索结果进行了修改。
前言
细胞周期蛋白依赖性激酶8(Cyclin-dependent kinase 8,CDK8)参与RNA聚合酶II介导的转录的调节,从而调节参与致癌控制的多种信号通路和多种转录因子。多种证据表明,CDK8失调与人类疾病有关,CDK8可能在癌症的发生发展中起致癌基因的作用。最近Li等人1报道了从Merck的选择性CDK8抑制剂MSC25308182开始,如图1所示,经过衍生物,采用基于结构的AI分子生成方法对连接臂(Linker)进行生物等排体替换得到先导化合物1,然后药化专家对1进一步优化得到化合物23。
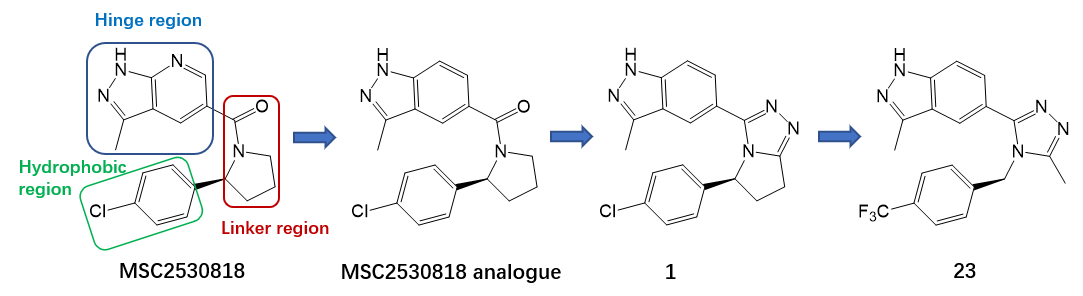
图1. 从MSC2530818到23的骨架跃迁与基团替换
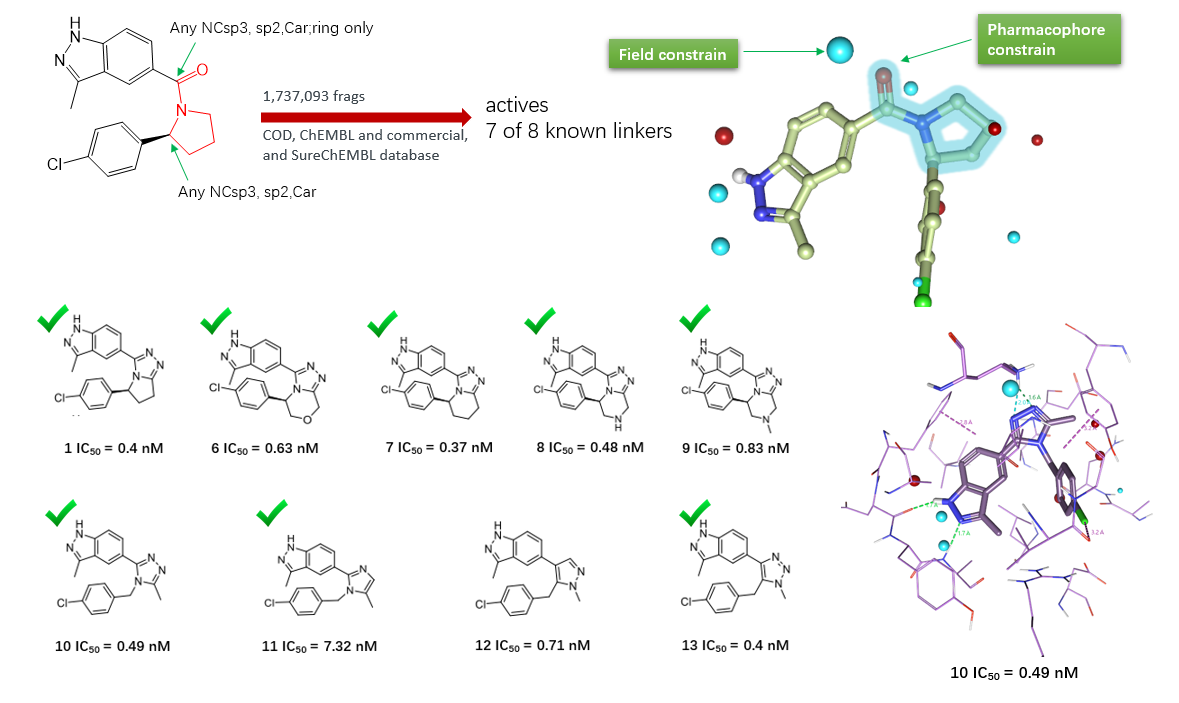
Spark是一款经过充分验证的生物等排体替换、大环化设计、水分子替换、分子连接与分子生长工具3,4,本文的主要目的是从MSC2530818衍生物(图1)开始,用SPARK对连接臂进行生物等排体替换,重现Li等人1化合物23的设计过程,并用Li等人1报道的8种活性化学类型化合物(图2)来验证Spark的设计。
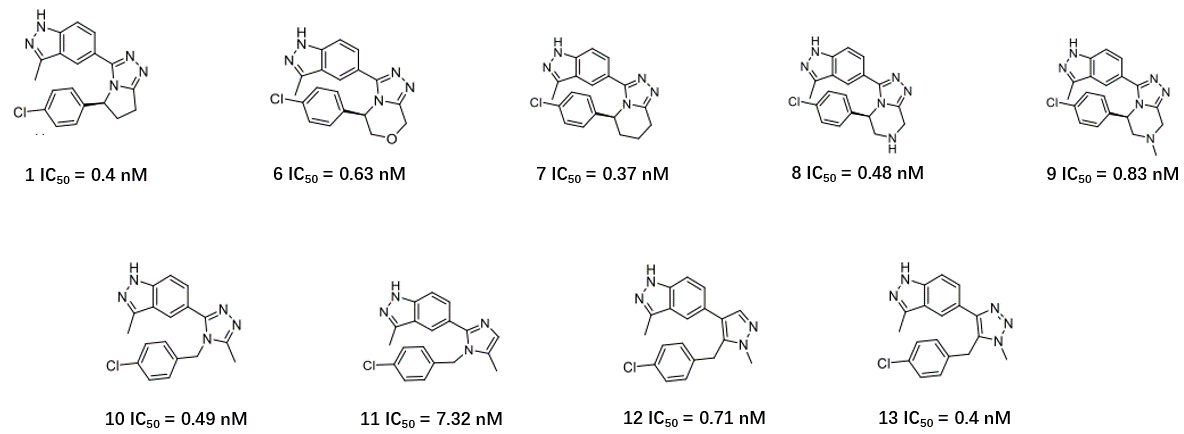
图2. Li等人1报道的8种典型活性化合物及其CDK8抑制活性
起点化合物与CDK8的复合物结构准备
MSC2530818与CDK8的共晶结构已经被Czodrowski等人2报道(PDB 5IDN),在Flare5里下载PDB 5IDN,然后用Protein preparation进行精心准备;最后用配体编辑器修改铰链结合片段(1H-吡唑并[3,4-b]吡啶)吡啶环上氮原子为碳,保存为MSC2530818衍生物。
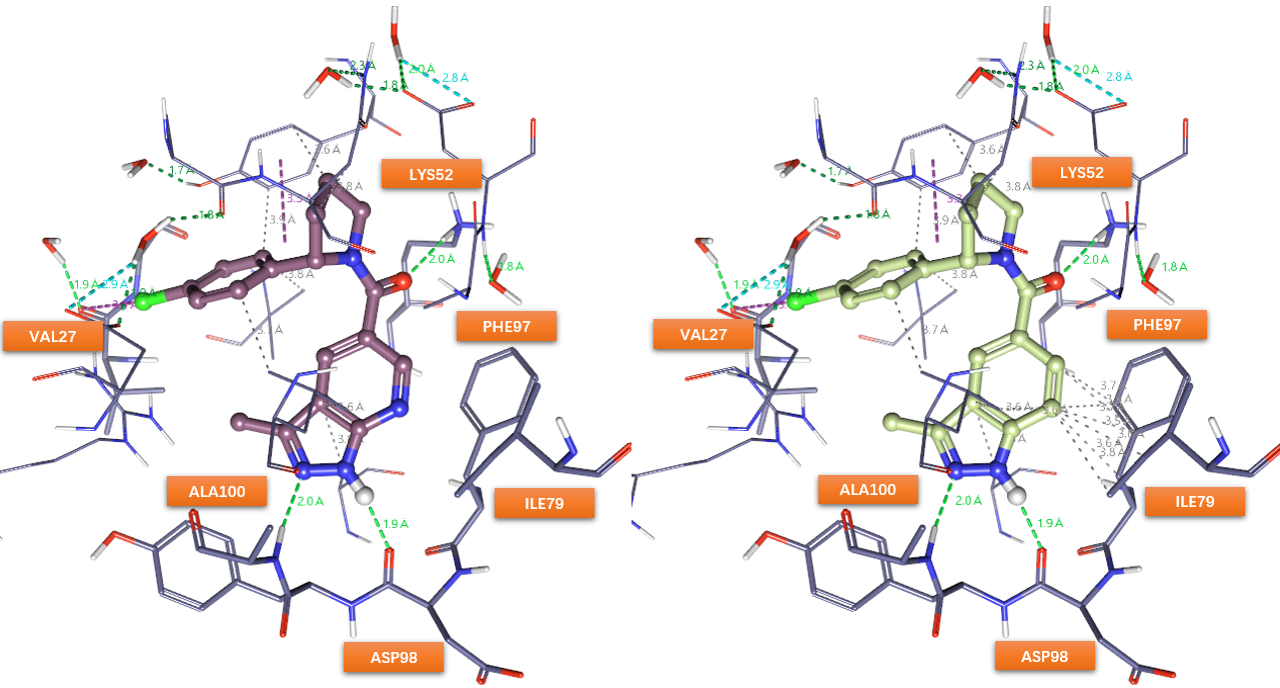
图3. MSC2530818(左)及其衍生物(右)与CDK8的相互作用比较(PDB 5IDN)
比较MSC2530818(左)及其衍生物(右)与CDK8的相互作用,如图3所示,可以发现比之MSC2530818,MSC2530818衍生物(图3-右)与CDK8多出了与PHE97、ILE79侧链的疏水接触,这一点可以从图4的相互作用2D图上可以更加清晰地看出来。从静电互补性(Electrostatic Complementarity,EC)5上讲,MSC2530818铰链区结合片段吡啶从N修改为C之后衍生物的EC打分值略有升高,说明这个修改改善了与靶标的静电相互作用。
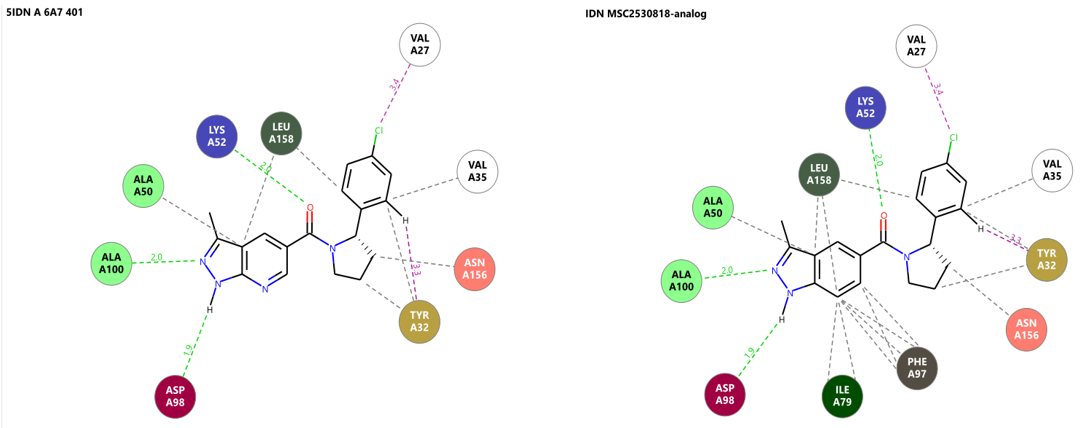
图4. MSC2530818(左)及其衍生物(右)与CDK8的相互作用2D图
连接臂的生物等排体替换实验
MSC2530818衍生物的连接臂生物等排体替换实验通过SPARK3来实现。Spark实验的query如图5所示,高亮区域为将被替换的部分,具有两个连接点1(与苯环连接的四氢吡咯C原子)与2(羰基C原子)。
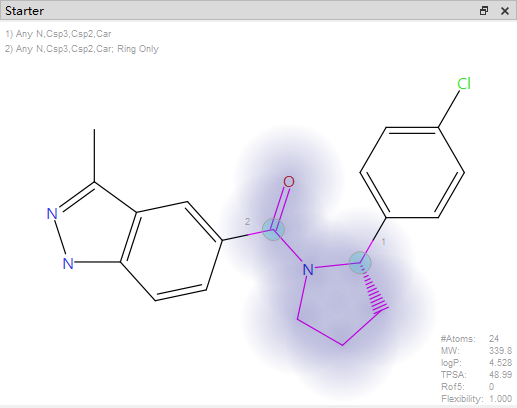
图5. SPARK生物等排体替换实验的Query
其中连接点1与2的设置如下:
- 连接点1:可以是C.sp3,C.sp2,C.ar,N.sp3,N.sp2,no ring constrains。
- 连接点2:可以是C.sp3,C.sp2,C.ar,N.sp3,N.sp2,ring atom only。
连接臂的生物等排体替换实验还使用了如图6所示的约束条件,包括:1)在羰基氧位置上添加氢键受体的药效团约束条件;2)在羰基氧下方的蓝色负场点处添加场约束条件。
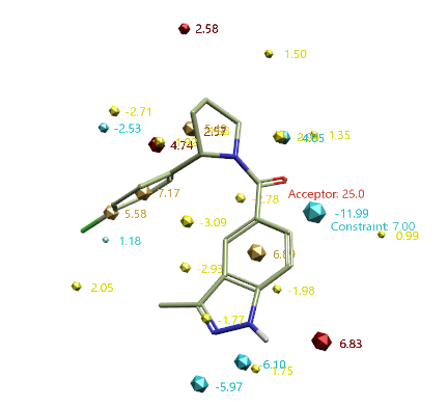
图6. SPARK生物等排体替换实验的约束条件
本次生物等排体替换实验对内置的COD、ChEMBL、Commercial与SureChEMBL等12个数据库6,7共1737093个片段进行虚拟筛选,如图7所示。
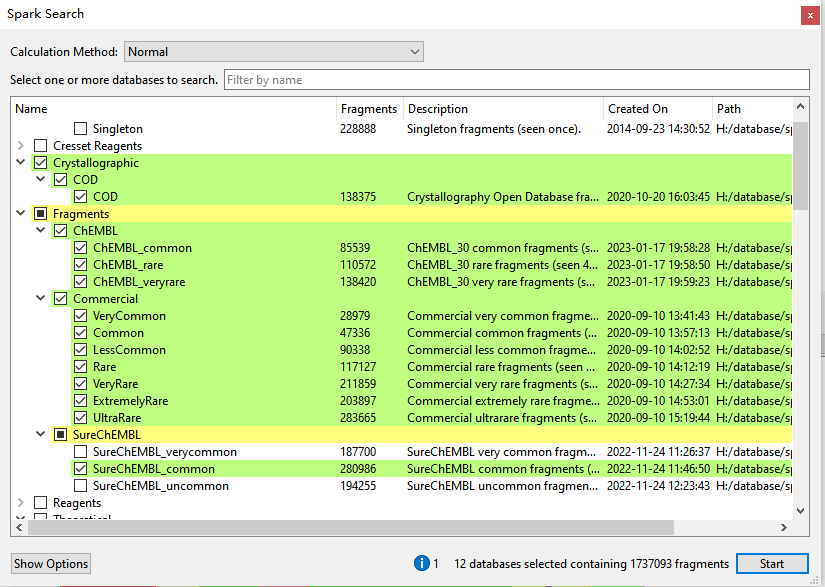
图7. SPARK生物等排体替换实验的数据库6,7
生物等排体替换实验使用了下面的设置:
- Calculation method: Normal
- Score method: Ligand Similarity
- Max number of results to keep: 500
其它参数均采用默认值。
结果
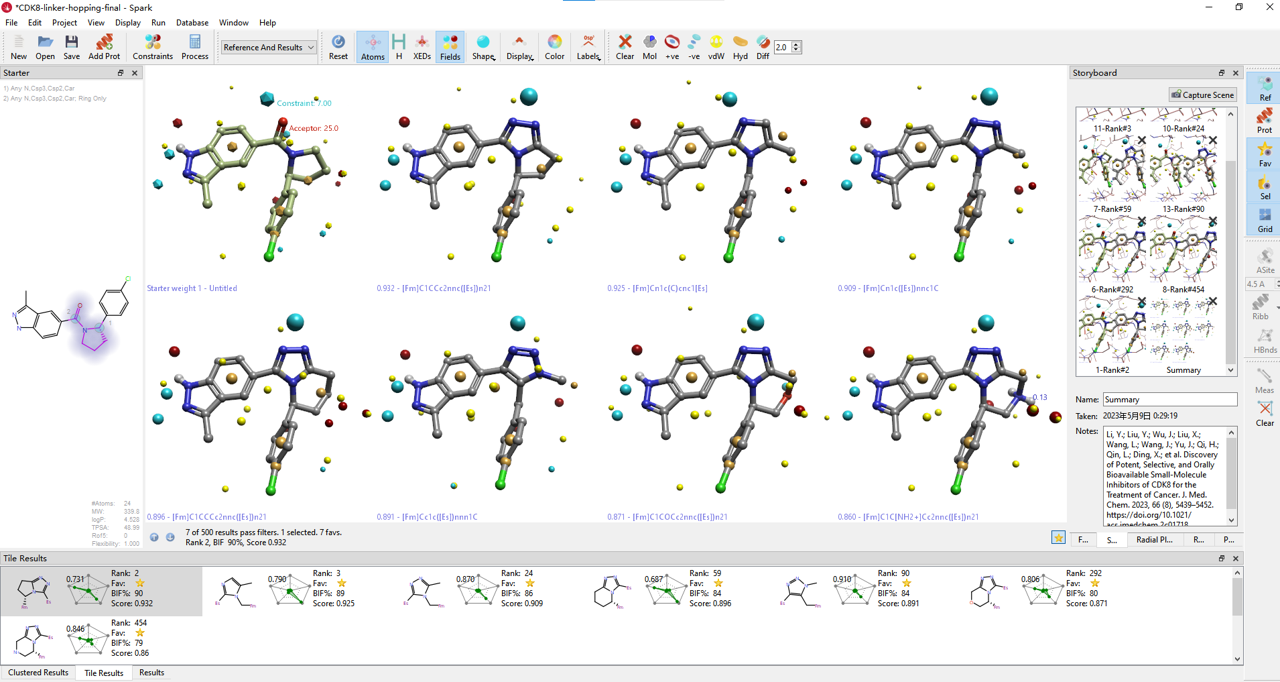
本次生物等排体替换实验共生成500个分子,其总体打分(score)、场相似性打分(Field score)、形状相似性打分(Shape score)以及生物等排体指数(BIF%)分布如表1所示。显而易见,这500个分子的打分都很高。单从打分值看,所有这些化合物都值得关注。
表1. SPARK生物等排体替换实验的500个结果主要打分值分布
| Items | Min | Max |
|---|---|---|
| Score | 0.857 | 0.938 |
| Field score | 0.773 | 0.922 |
| Shape score | 0.849 | 0.962 |
| BIF% | 78 | 91 |
对连接臂(linker)用Bemis-Murcko clustering方法进行聚类分析,总共包含96种不同化学类型。在这些Spark生成的结果中总共有7种连接臂出现在Li等人1报道的8个连接臂骨架中,如图8所示,其中Linker列给出骨架2D结构;Cluster Rank表示连接臂骨架在全部96个骨架中的排名;NC表示在该类别下命中的化合物数;Rank表示在文章中出现的化合物在500个结果中的打分排序;Compound in paper表示命中化合物在文章中的ID(见图2);Score与BIF%分别表示被文章报道的化合物总体打分与连接臂的生物等排体指数。
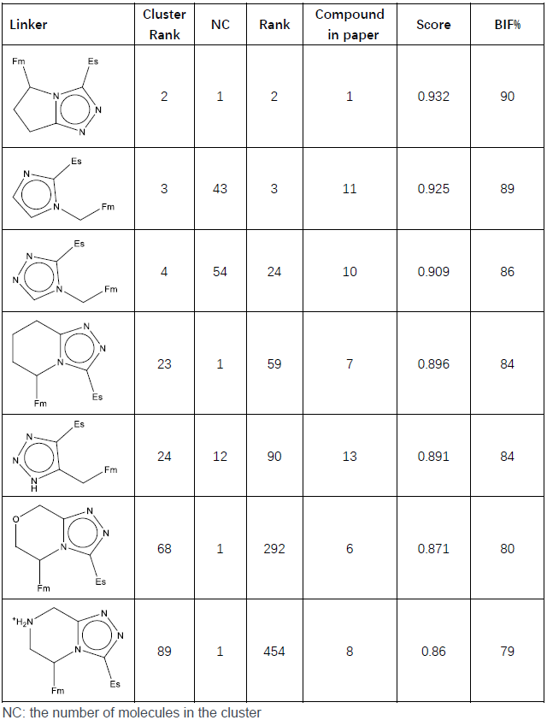
图8. SPARK生物等排体替换实验的重现了7种典型的连接臂
在Spark实验结果中没有出现Li等人1报道的化合物12。经过检索发现,虽然化合物12的骨架已经包含在所用的数据库里,但是12的骨架不满足Spark实验的场约束与羰基氧的氢键受体约束而不能被命中。如图9所示,一方面,起始化合物(左)羰基附近有一个大的、蓝色的负静电场点,而在化合物12却对应一个小的正静电势场点,因此化合物12不能满足场约束的要求;另一方面,羰基氧与吡唑CH重合(未呈现),因此不能满足药效团约束。这说明基于配体相似性的方法对化合物12不利,在补充部分第1小节里我们用Docking作为打分函数,从Commercial VeryCommon数据库里搜索到化合物12连接臂。
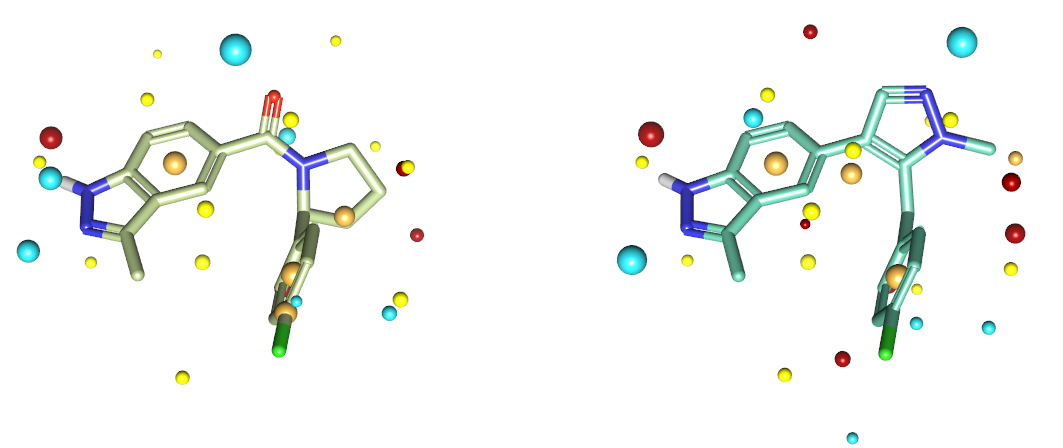
图9. 起点化合物MSC2530818衍生物(左)与化合物12(右)的场点比较
在Spark生成的96个连接臂中,其中7个被Li等人报道1有活性。具体的代表性化合物如图8与10所示,包含了文章中已知活性的化合物1、6、7、8、10、11与13。如图9所示,它们具有非常相似的场点,它们的连接臂骨架排序与化合物打分排序(Cluster Rank)均比较靠前,总体打分score大于0.8,生物等排体指数(BIF%)也均在70以上。
图10. 在Spark生物等排体替换实验结果中包含了7种已知连接臂的代表性化合物
在Li等人1的研究中选择了化合物10的连接臂进行了进一步研究,并对疏水区(图1)优化之后得到的化合物23进入下一轮研究。比较起始分子与化合物10的CDK8结合模式,如图11所示,可以发现起始分子的连接臂羰基氧与化合物10的三氮唑C=N重合,作为氢键受体与LYS52末端质子化的氨基发生氢键相互作用;起始分子连接臂与化合物10连接臂重合地相当良好,形状相当一致。同时Spark给出的整体分子之间的相似性定量打分也相当高,其中场相似性打分Field score = 0.883,形状相似性打Shape score = 0.9355,总体打分score = 0.909;连接臂局部相似性也相当高,体现在生物等排体指数BIF% = 86。
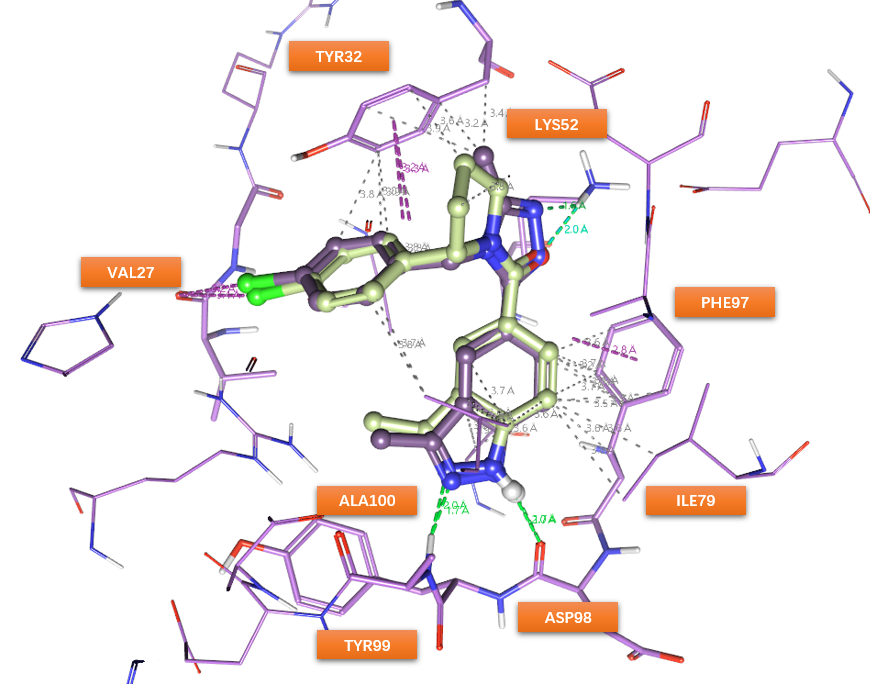
图11. 起始分子(淡黄色)与化合物10(淡紫色)的CDK8结合模式比较
在本次计算中,给出的500个分子里,整体打分score最小值为0.857,BIF%最小值为78,也就是说所有的分子打分都非常高。鉴于Li等人1报道的大部分连接臂骨架可被Spark以高的总体打分与生物等排体指数(BIF%)打分命中,那么其它总体打分高且BIF%高的未见文献报道的连接臂骨架也是有很大的概率具有良好的活性,值得进一步研究。
在Li等人1的研究中,如图1所示,连接臂部分两步优化获得:1)从起始分子出发,先用基于结构的深度学习结构生成获得双环连接臂的先导化合物1,2)再通过药化专家开环设计得到三氮唑连接臂。而在Spark的连接臂生物等排体替换实验中,直接从起始分子出发,直接生成了包括双环连接臂与三氮唑连接臂在内的7种经过验证的连接臂,因此Spark比基于结构的AI结构生成表现出更高的效率。
结论
本文以Merck的选择性CDK8抑制剂MSC2530818的衍生物为起点分子,用SPARK对连接臂部分进行了生物等排体替换,重现了Li等人1CDK8抑制剂23设计过程中8种连接臂中的7种。比起基于结构的深度学习结构生成与药化专家设计的两步优化过程,Spark可以从起点分子设计出发,一步直接设计出包括先导化合物1与优选化合物23在内的连接臂化合物,表现出更高效的生物等排体替换性能。
补充
1. 基于结构的打分———化合物12的设计
本部分计算实验的目的是验证用基于结构的打分是否适用于化合物12的设计。选择了一个比较小的数据库Commercial|VeryCommon(2020年9月),事先知道化合物12的连接臂出现在这个数据库里,我们感兴趣的是采用Docking做为打分函数能否将化合物12的吡唑连接臂重现出来。出于这个目的,在进行生物等排体替换的时候,使用了如下参数:
- Calculation method: Normal
- Score method: Docking
- Max number of results to keep: 500
- Option | filter: contains a ring, H-bond acceptor
- Option | Advanced filters: Cn1cccn1 is required
选项filter设置含有一个环与一个氢键受体是为了缩小搜索范围,Advanced filters设置包含吡唑环的结构也是为了进一步缩小搜索范围,但不影响我们的实验目的。
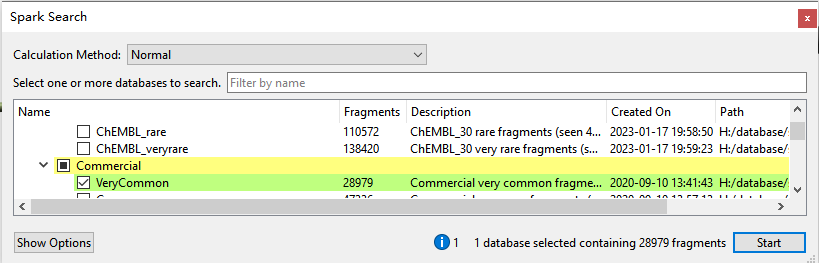
图12. 搜索Commercial VeryCommon数据库(2020年9月份版本)进行生物等排体替换实验
如图12所示,本次搜索用的Very Common数据库来创建于2020年9月10日,包含了28979个不重复的片段。
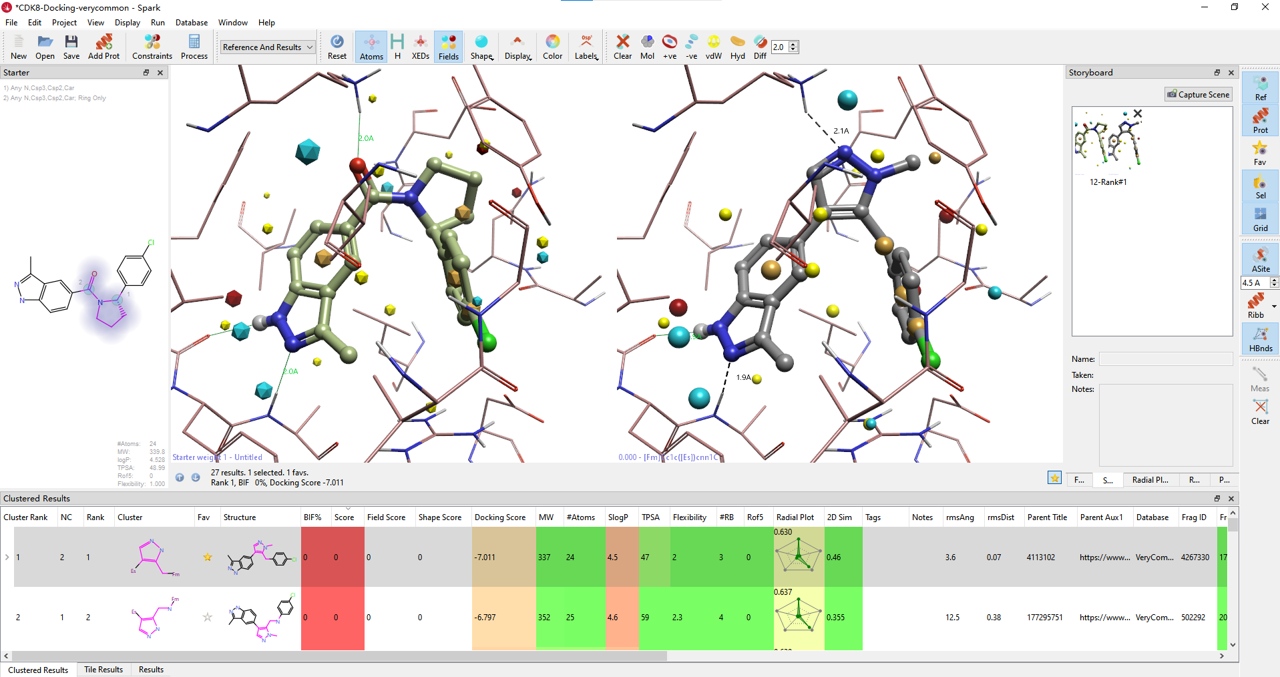
图13. 搜索Very Common数据库的结果Rank #1(总共500个结果)为化合物12连接臂
总共生成了500个结果,结果如图13所示,Docking score排序第1的结果(Rank #1)即为Li等人1报道的化合物12。这证明了我们之前的猜测,化合物12不适合用基于配体的打分而需要用蛋白的信息进行docking打分才能被重现出来。
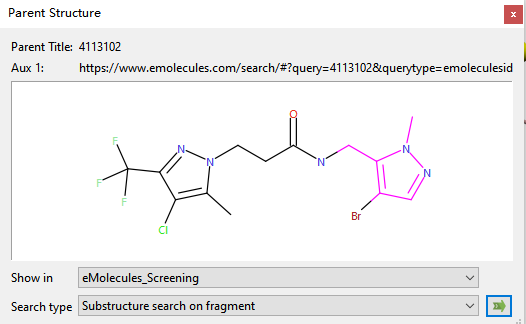
图14. Spark结果Rank #1(化合物12连接臂)的母体结构
Spark结果还给出了Rank #1的母体结构,如图14所示,连接臂裁剪自Emolecules的化合物4113102,该连接臂在Emolecules化合物库里不同的1790个分子里出现过,是个非常常见的片段。
总的来说,这部分计算实验说明基于结构与基于配体的打分结果是互补的,基于结构方法可以发现基于配体方法不能发现的新结果。
文献
- Li, Y.; Liu, Y.; Wu, J.; Liu, X.; Wang, L.; Wang, J.; Yu, J.; Qi, H.; Qin, L.; Ding, X.; et al. Discovery of Potent, Selective, and Orally Bioavailable Small-Molecule Inhibitors of CDK8 for the Treatment of Cancer. J. Med. Chem. 2023, 66 (8), 5439–5452. https://doi.org/10.1021/acs.jmedchem.2c01718.
- Czodrowski, P.; Mallinger, A.; Wienke, D.; Esdar, C.; Pöschke, O.; Busch, M.; Rohdich, F.; Eccles, S. A.; Ortiz-Ruiz, M.-J.; Schneider, R.; et al. Structure-Based Optimization of Potent, Selective, and Orally Bioavailable CDK8 Inhibitors Discovered by High-Throughput Screening. J. Med. Chem. 2016, 59 (20), 9337–9349. https://doi.org/10.1021/acs.jmedchem.6b00597.
- Spark™, Cresset®, Litlington, Cambridgeshire, UK; https://www.cresset-group.com/spark; Cheeseright T., Mackey M., Rose S., Vinter, A.; Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation. J. Chem. Inf. Model. 2006, 46 (2), 665-676
- Matthew P. Baumgartner and David A. Evans, Side chain virtual screening of matched molecular pairs: a PDB-wide and ChEMBL-wide analysis, Journal of Computer-Aided Molecular Design 2020, 34, 953–963. https://doi.org/10.1007/s10822-020-00313-1
- Flare™, Cresset®, Litlington, Cambridgeshire, UK; https://www.cresset-group.com/flare/; Cheeseright T., Mackey M., Rose S., Vinter, A.; Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation J. Chem. Inf. Model. 2006, 46 (2), 665-676; Bauer M. R., Mackey M. D.; Electrostatic Complementarity as a Fast and Effective Tool to Optimize Binding and Selectivity of Protein–Ligand Complexes J. Med. Chem. 2019, 62, 6, 3036-3050; Maximilian Kuhn, Stuart Firth-Clark, Paolo Tosco, Antonia S. J. S. Mey, Mark Mackey and Julien Michel Assessment of Binding Affinity via Alchemical Free-Energy Calculations J. Chem. Inf. Model. 2020, 60, 6, 3120–3130
- Spark数据库. http://www.molcalx.com.cn/cresset/spark-databases
- Spark™新增SureChEMBL片段数据库,交付比以往更大的化学空间. 墨灵格的博客. http://blog.molcalx.com.cn/2023/04/02/surechembl-fragment-databases.html