摘要:靶向蛋白降解是一种新的治疗方法,为以前认为难以成药靶标的药物发现提供了新方法,目前已有多个蛋白降解剂项目进入临床开发阶段。Cresset Discovery咨询团队在靶向蛋白质降解方面拥有丰富的经验,并在全球范围内为这一领域的公司提供支持。本文介绍了我们的CADD专家团队如何用专业的计算方法和专业知识助力您的蛋白降解剂项目。
药物发现中靶向蛋白降解(targeted protein degradation)的出现
近年来,靶向蛋白降解已成为一种新的治疗方式。这项新技术现在是制药和生物技术公司众多研发项目的一部分,这些项目现已进入临床前和临床开发阶段。为诸如转录因子、骨架蛋白、没有结合口袋或结合口袋太宽或太浅的蛋白等等以前被人们认为不具成药性的靶标提供了新方法。
Cresset Discovery在靶向蛋白质降解方面拥有丰富的经验,并在全球范围内为这一领域的公司提供支持。我们的CADD专家团队可以通过应用专业的计算方法和专业知识,深入了解您的降解剂体系。
什么是蛋白降解剂(protein degraders)
靶向蛋白降解广义上指的是利用细胞自身的机制来消除或降解感兴趣的蛋白,而不是抑制它的策略。目前有许多不同的策略被用于开发蛋白质降解剂。
靶向嵌合体蛋白分解(Proteolysis targeting chimeras,PROTACs)和分子胶(molecular glues)是促进靶向蛋白酶体蛋白降解的两种主要方法,是细胞废物处理系统1,2。这两种方法都是通过将靶蛋白与诸如Cereblon(CRBN)或von Hippel-Lindau蛋白(VHL)之类的E3泛素连接酶或与600多种已知E3泛素连接酶中的任何一种紧密靠近形成三元复合物(如图1所示)结合成的泛素-蛋白酶体系统(ubiquitin-proteasome system ,UPS)来进行操作。
然后靶标蛋白被连接蛋白泛素化,对它进行标识以便被蛋白酶体识别并降解。之后,降解剂分子解离并被回收,以靶向靶标蛋白的新副本。这种催化型的作用机制比传统的小分子抑制剂具有优势,因为大多数降解剂如PROTACs需要较低的浓度才能产生相同的效应,这就减少了脱靶和潜在的毒性作用的发生。
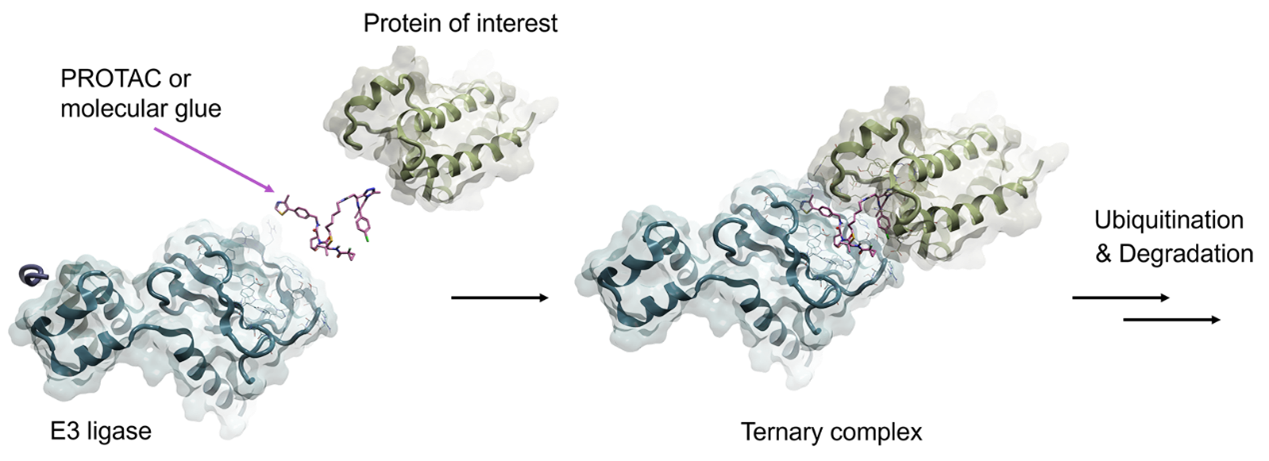
图1. 一个PROTAC分子(粉红色)与它的蛋白靶标(绿色)和E3连接酶(蓝色)结合成一个三元复合体,以触发蛋白体降解。
PROTAC分子是由两个配体通过柔性或刚性连接臂连接组成的双功能小分子,其中一个配体与靶标蛋白结合,另一个配体与E3泛素连接酶结合。而分子胶(molecular glue)降解剂通过直接参与E3泛素连接酶和感兴趣蛋白之间的蛋白-蛋白相互作用(protein-protein interaction,PPI)来稳定这两种蛋白之间的相互作用。
用计算化学专业知识优化蛋白质降解剂的设计
你可以通过聘请我们的建模专家来加强你降解剂的设计和开发的每一步。我们不仅在识别致病蛋白的选择性降解剂以及新的E3连接酶弹头方面经验丰富,而且我们将对你的降解剂分子进行勤奋的结构指导设计,包括评估其三元复合物和连接臂的影响。我们可以帮助选择E3泛素连接酶,并考虑降解剂的物理化学特性,这些特性可能超出Lipinski的 “五项规则 “范围。
许多三元复合物的实验结构可用于降解剂结合的计算研究2。我们的团队利用这些结构来帮助你可视化和理解PPI以及与降解剂分子的相互作用,并为优化新的降解剂设计提供宝贵的见解。在没有实验结构的情况下,可以采用适当的相关蛋白结构来构建一个叠合模型或同源模型。
一个例子是设计分子胶降解剂将GSPT1与CRBN结合成复合物。表型筛选和化合物库设计确定了早期的GSPT1降解剂CC-885(图2a)和随后的CC-90009,也被称为Eragidomide,目前正处于治疗急性骨髓性白血病的一期临床试验中(图2b)3。 然后,一个独立小组应用分子对接和基于结构的库设计报告了改进的降解剂SJ6986(图2c)4。

图2. 让CRBN(米色)与GSPT1(紫色)形成复合物的分子胶降解剂
通过对GSPT1降解剂配体的相互作用进行建模5,我们对实验和对接计算的复合物分子表面进行可视化分析。如图2所示,绿色编码的区域是降解剂的静电场与CRBN的静电场互补的地方。这些区域随着GSPT1降解剂的逐步优化而变得更加广泛。相应地,不利EC的红色区域也相应减少。
Cresset Discovery的科学家使用专有的方法将EC预测结果纳入许多蛋白降解建模工作流程中。当蛋白受体的结构可用时,我们在用对接打分的同时也EC进行打分,以准确和快速地评估数以千计的苗头化合物6。在化合物库设计中,我们对枚举的结构排序产生入围名单用于视觉检查,并识别出那些与受体发生有利的相互作用或与已知配体相似的结构。在虚拟筛选中,我们一直在识别新的活性配体,并取得了显著的成功7。我们建立的设计范式同样适用于E3泛素连接酶和降解的靶标受体。这也延伸到对特定的降解剂成分,比如连接酶弹头或PROTAC连接臂的化学替代物进行建模。
让Cresset Discovery加速您的靶向蛋白质降解项目
Cresset Discovery是药物发现中计算模型的一个真正的合作伙伴。我们的专家团队在药物化学、计算化学以及生物学方面有广泛的经验,可以与您一起工作,实现您的项目目标。
想要了解更多关于我们如何在蛋白降解项目或任何其他项目中支持你,请联系我们或要求与我们的团队进行无风险的免费保密讨论。
相关文章
- 了解Cresset Discovery都提供什么样的服务:http://blog.molcalx.com.cn/2020/06/27/cresset-discovery-service.html
- 了解Cresset Discovery服务流程:http://blog.molcalx.com.cn/2022/10/26/engaging.html
文献
- Miklos Bekes, David R. Langley and Craig M. Crews, PROTAC targeted protein degraders: the past is prologue, Nature Reviews Drug Discovery 2022, 21, 181-200. https://www.nature.com/articles/s41573-021-00371-6.
- Zuzanna Kozicka and Nicolas Holger Thoma, Haven’t got a glue: Protein surface variation for the design of molecular glue degraders, Cell Chemical Biology 28, 1032-1046. https://doi.org/10.1016/j.chembiol.2021.04.009.
- Surka, C., Jin, L., Mbong, N., Lu, C.-C., Jang, I.S., Rychak, E., Mendy, D., Clayton, T., Tindall, E.A., Hsu, C., et al. CC-90009, a novel cereblon E3 ligase modulator targets acute myeloid leukemia blasts and leukemia stem cells, Blood 2020,137, 661–677. https://doi.org/10.1182/blood.2020008676.
- Nishiguchi, G., Keramatnia, F., Min, J., Chang, Y., Jonchere, B., Das, S., Actis, M., Price, J., Chepyala, D., Young, B., McGowan, K., Slavish, P.J., Mayasundari, A., Jarusiewicz, J.A., Yang, L., Li, Y., Fu, X., Garrett, S.H., Papizan, J.B., Kodali, K., Peng, J., Pruett Miller, S.M., Roussel, M.F., Mullighan, C., Fischer, M., Rankovic, Z., Identification of potent, selective, and orally bioavailable small-molecule GSPT1/2 degraders from a focused library of cereblon modulators J. Med. Chem. 2021, 64, 7296–7311. https://doi.org/10.1021/acs.jmedchem.0c01313.
- Electrostatic Complementarity™ (EC)
- Lead Finder™, BioMolTech®, Toronto, Ontario, Canada; https://www.cresset-group.com/lead-finder/
- Blaze™, Cresset®, Litlington, Cambridgeshire, UK; https://www.cresset-group.com/blaze/; Cheeseright, T.J.; Mackey, M.D., Melville, J.L.; Vinter, J.G. FieldScreen: Virtual Screening Using Molecular Fields. Application to the DUD Data Set. J. Chem. Inf. Model. 2008, 48 (11), 2108-2117; Cheeseright T., Mackey M., Rose S., Vinter, A.; Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation. J. Chem. Inf. Model. 2006, 46 (2), 665-676
Flare™, Cresset®, Litlington, Cambridgeshire, UK; https://www.cresset-group.com/flare/; Cheeseright T., Mackey M., Rose S., Vinter, A.; Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation J. Chem. Inf. Model. 2006, 46 (2), 665-676; Bauer M. R., Mackey M. D.; Electrostatic Complementarity as a Fast and Effective Tool to Optimize Binding and Selectivity of Protein–Ligand Complexes J. Med. Chem. 2019, 62, 6, 3036-3050; Maximilian Kuhn, Stuart Firth-Clark, Paolo Tosco, Antonia S. J. S. Mey, Mark Mackey and Julien Michel Assessment of Binding Affinity via Alchemical Free-Energy Calculations J. Chem. Inf. Model. 2020, 60, 6, 3120–3130



