摘要:晶状体上皮衍生生长因子(LEDGF)或转录共活化因子p75(LEDGF/p75)在HIV整合中起到至关重要的作用。HIV-1整合酶(Integrase,IN)与它的细胞因子LEDGF/75之间的蛋白-蛋白相互作用或许可以作为开发全新抗HIV药物的靶点。在本研究中,用Ligandscout基于结构的策略开发了一个HIV-1的IN-LEDGF/p75相互作用的基于结构的药效团模型。并用该模型虚拟筛选CHIME数据库、发现了一个苗头化合物CHIBA-3002。对苗头化合物进行进一步优化:采用理性设计、合成了4个化合物、并进行了生物活性评价。最后发现一个小分子化合物CHIBA-3003可以在uM水平干扰HIV-1的IN-LEDGF/p75相互作用。这是首个专一作用于HIV-1的IN-LEDGF/p75蛋白-蛋白相互作用并有活性的化合物。本研究的靶点不同于现有抗病毒药物,为抗HIV药物开发开辟了全新方向。
原文:De Luca, L.; Barreca, M. L.; Ferro, S.; Christ, F.; Iraci, N.; Gitto, R.; Monforte, A. M.; Debyser, Z.; Chimirri, A. Pharmacophore-Based Discovery of Small-Molecule Inhibitors of Protein-Protein Interactions Between HIV-1 Integrase and Cellular Cofactor LEDGF/P75. ChemMedChem 2009, 4 (8), 1311–1316. DOI:10.1002/cmdc.200900070
一. 背景介绍
晶状体上皮衍生生长因子(lens epithelium-derived growth factor,LEDGF)或转录共活化因子(transcriptional coactivator)p75(LEDGF/p75)在HIV整合中起到至关重要的作用。HIV-1整合酶(Integrase,IN)与它的细胞因子LEDGF/75之间的蛋白-蛋白相互作用(protein–protein interactions,PPI)因此或许可以作为开发全新抗HIV药物的靶点。在本研究中,用Ligandscout开发了一个HIV-1的IN-LEDGF/p75相互作用的基于结构的药效团模型。并用该模型对自用的数据库CHIME进行虚拟筛选发现一个苗头化合物CHIBA-3002。对苗头化合物进行进一步优化:采用理性设计、合成了4个化合物、并进行了生物活性评价。最后发现一个小分子化合物CHIBA-3003可以在μM水平干扰HIV-1的IN-LEDGF/p75相互作用。这是首个专一作用于HIV-1的IN-LEDGF/p75蛋白-蛋白相互作用并有活性的化合物。本研究的靶点不同于现有抗病毒药物,为抗HIV药物开发开辟了全新方向。
二. 方法与结果
LEDGF/p75与HIV-1 整合酶(HIV-1 IN)的结合是通过一个小的IN结合域(IN-binding domain, IBD)来实现。IBD是与HIV-1 IN发生相互作用的充分必要条件,LEDGF/p75 IBD与HIV0-1 IN二聚体复合物催化核心部分已有X-Ray晶体结构发布(PDB code:2B4J)。De Luca等人的目的是从这些结构生物学数据出发生成药效团模型、并用模型识别能够干扰HIV-1 IN-LEDGF/p75相互作用的全新化合物。该计算方法也适用于其它的PPI抑制剂研究。
IBD最关键的相互作用残基为Ile365、Asp366、Phe406, 已经有突变实验证实对LEDGF/p75上的这些残基突变会破坏其与HIV-1 IN之间的相互作用。作者假设:如果一个小分子化合物可以模拟LEDGF/p75 IBD上的二肽Ile365、Asp366,那么这个化合物应该可以抑制LEDGF/p75与HIV-1 IN之间的识别。
以C链LEDGF/p75的二肽Ile365-Asp366为配体,以A、B链为受体(见图1)用ligandscout识别药效团(图2为2D模型);再用该药效团对化合物进行虚拟筛选,发现化合物CHIBA-3002(见图3),在100μM时对HIV-1 IN-LEDGF/p75相互作用具有46%抑制作用。
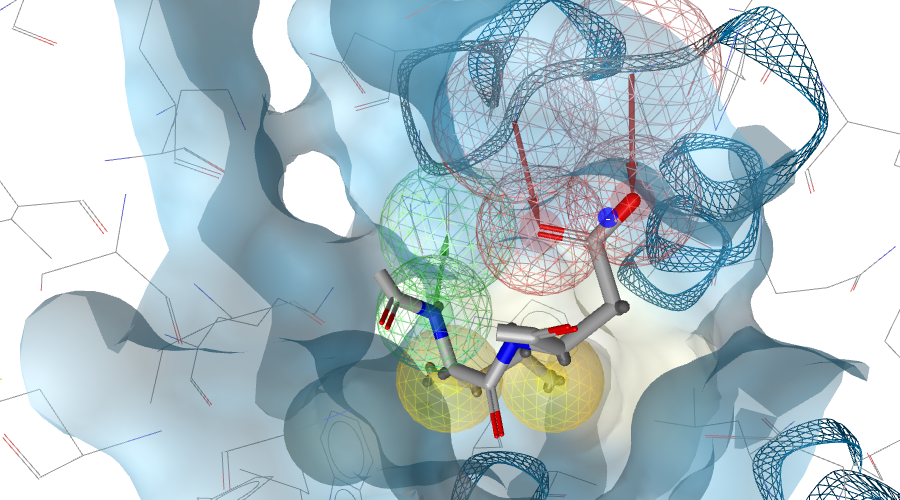
图1. PDB 2B4J: LEDGF/p75 IBD上的二肽Ile365、Asp366(棒状模型)与HIV-I IN(分子表面)之间的相互作用
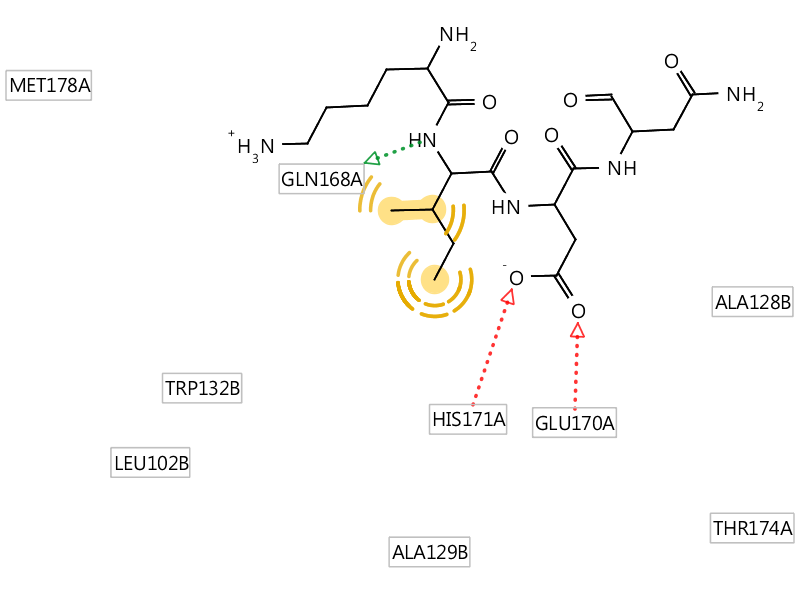
图2. 以LEDGF/p75 IBD上的二肽Ile365、Asp366作为配体,用Ligandscout生成LEDGF/p75-IN药效团模型
图3. 虚拟筛选命中化合物CHIBA-3002,在100μM时对HIV-1 IN-LEDGF/p75相互作用具有46%抑制作用.
三. 操作步骤
- 下载2B4J
- 删除无关的残基或杂原子
- 选择C链的二肽Ile365-Asp366及其两侧的残基,并设为配体
- 将C链其它的残基删除
- 定义结合位点
- 生成药效团模型
- 将Ile365-Asp366残基两侧之外残基生成的药效团特征删除,就很容易得到文章的药效团了(见图1)。
图4. 按Shift键,用鼠标右键选择合适的分子,再点删除按钮
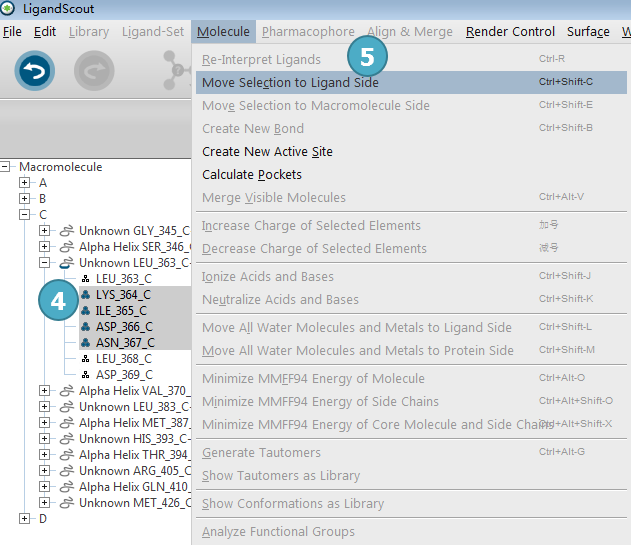
图5. 将选中的残基设为配体
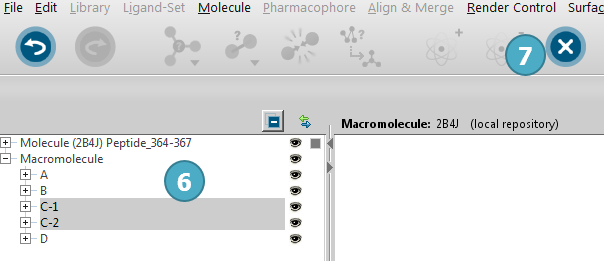
图6. 将C链其它残基删除
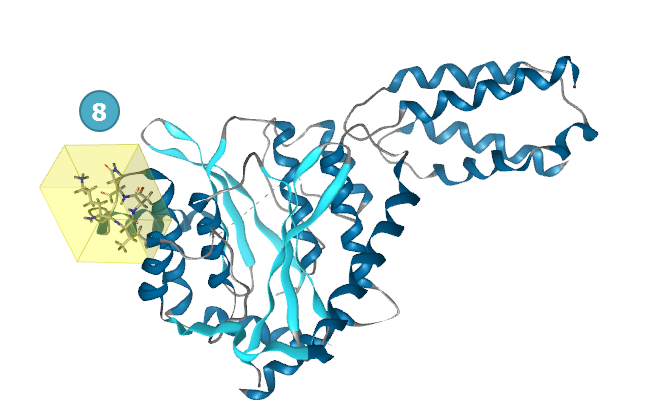
图7. 点击黄色框框,定义结合位点
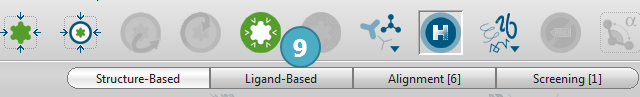
图8. 点击药效团生成按钮,生成药效团
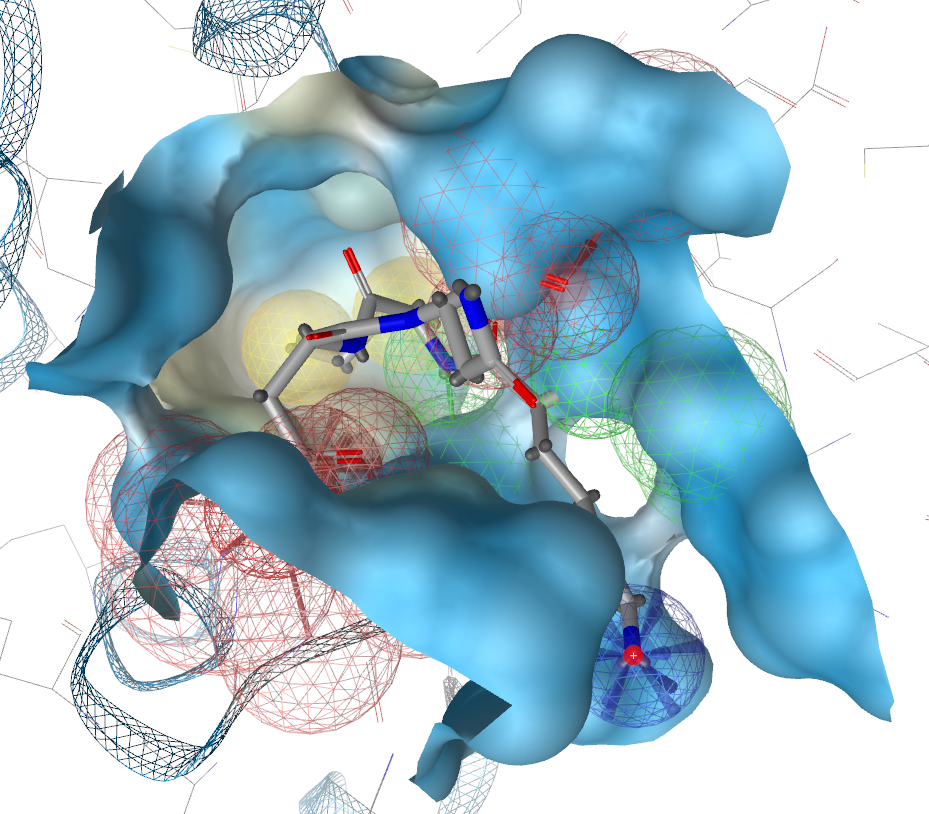
图9. 初始的药效团包含了Ile365-Asp366两侧之外的残基,是个信息冗余、复杂的模型,因此需要将与两侧残基有关的Feature删除,最后得到图1所示的药效团模型。
四. 相关文章
Liu, J.; Tian, Z.; Zhou, N.; Liu, X.; Liao, C.; Lei, B.; Li, J.; Zhang, S.; Chen, H. Targeting the Apoptotic Mcl-1-PUMA Interface with a Dual-Acting Compound. Oncotarget 2017, Vol 5. DOI:10.18632/oncotarget.17294
Ligandscout案例 | 第四军医大学刘吉元等人首次发现Mcl-1-PUMA调节小分子: http://blog.molcalx.com.cn/2017/06/02/ligandscout-ppi-mcl-1-puma.html



