摘要:在本文中,我们利用计算方法重现了新型合成致死药物MRTX1719的基于片段的发现过程。研究始于片段苗头化合物F1与PRMT5•MTA复合物的共晶结构。通过运用Flare软件中的蛋白质相互作用场以及GIST水分子分析技术,我们深入分析了F1所在结合口袋的特性,成功识别出两个具有高能量且潜在可被替换的结合水——HOH894和HOH886。随后,借助Spark的水分子替换模块,针对上述两个水分子位点进行了两轮水分子替换处理,在F1基础上引入了新的基团,从而获得了一系列与目标候选药物MRTX1719高度相似的先导化合物。鉴于这些计算所得的先导化合物与最终的候选药物极为接近,本研究证明了计算方法能够显著加速药物发现过程,为新型药物的研发提供了强有力的支持。
肖高铿/2024-11-26
前言
PRMT5-MEP50•MTA(PRMT5•MTA)复合物是最近发现的治疗MTAP缺失型癌症的合成致死药物靶标。假设一个能够与催化失活的PRMT5•MTA复合物结合、并使其稳定的化合物会抑制在MTAP缺失型肿瘤细胞中的PRMT5活性,并同时保留在MTAP野生型(WT)细胞中的PRMT5活性,那么这个化合物可能是一种潜在的精准医疗药物,用于治疗MTAP缺失型癌症。基于这个假设,Smith C.R.等人1发现了候选化合物MRTX1719,过程如图1所示:首先用片段筛选方法发现了片段苗头化合物F1;然后用基于结构的设计方法,采用分子生长策略得到结合亲和力提高的片段F9;接着进行第二轮的分子生长、SAR探索,最终发现了候选化合物MRTX1719。
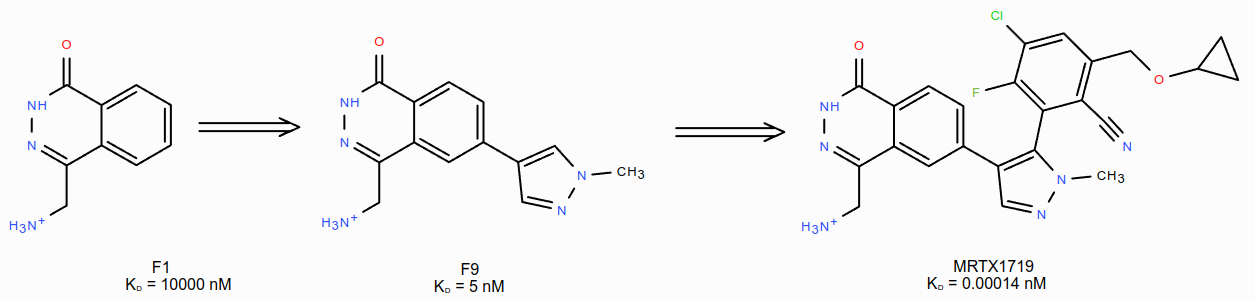
图1. MRTX1719基于片段的发现过程
本文从Smith等人1解释的PRMT5•MTA-F1共晶结构(PDB 7S0U)出发,用Flare™(Version 9)2对蛋白结合位点进行静电分析与GIST水分析,并依此制定水分子替换设计的策略,然后用Spark3的水分子替换实验重现图1所示的分子生长设计,并用Smith等人1报道的数据回溯性地验证该水分子替换设计的正确性与高效性。
结果
PRMT5•MTA复合物与片段苗头化合物F1的相互作用分析
将Smith C.R.等人1解释的F1共晶结构PDB 7S0U下载到Flare,并用Protein Prep进行标准的蛋白结构准备,然后用FPocket对准备好的蛋白结构进行分析,结果如下图2所示。
图2. PRMT5•MTA-F1共晶结构(PDB 7S0U)的结合位点。飘带:RMT5•MTA复合物;黄色球棍:F1;紫色表面:Fpocket计算的结合口袋。
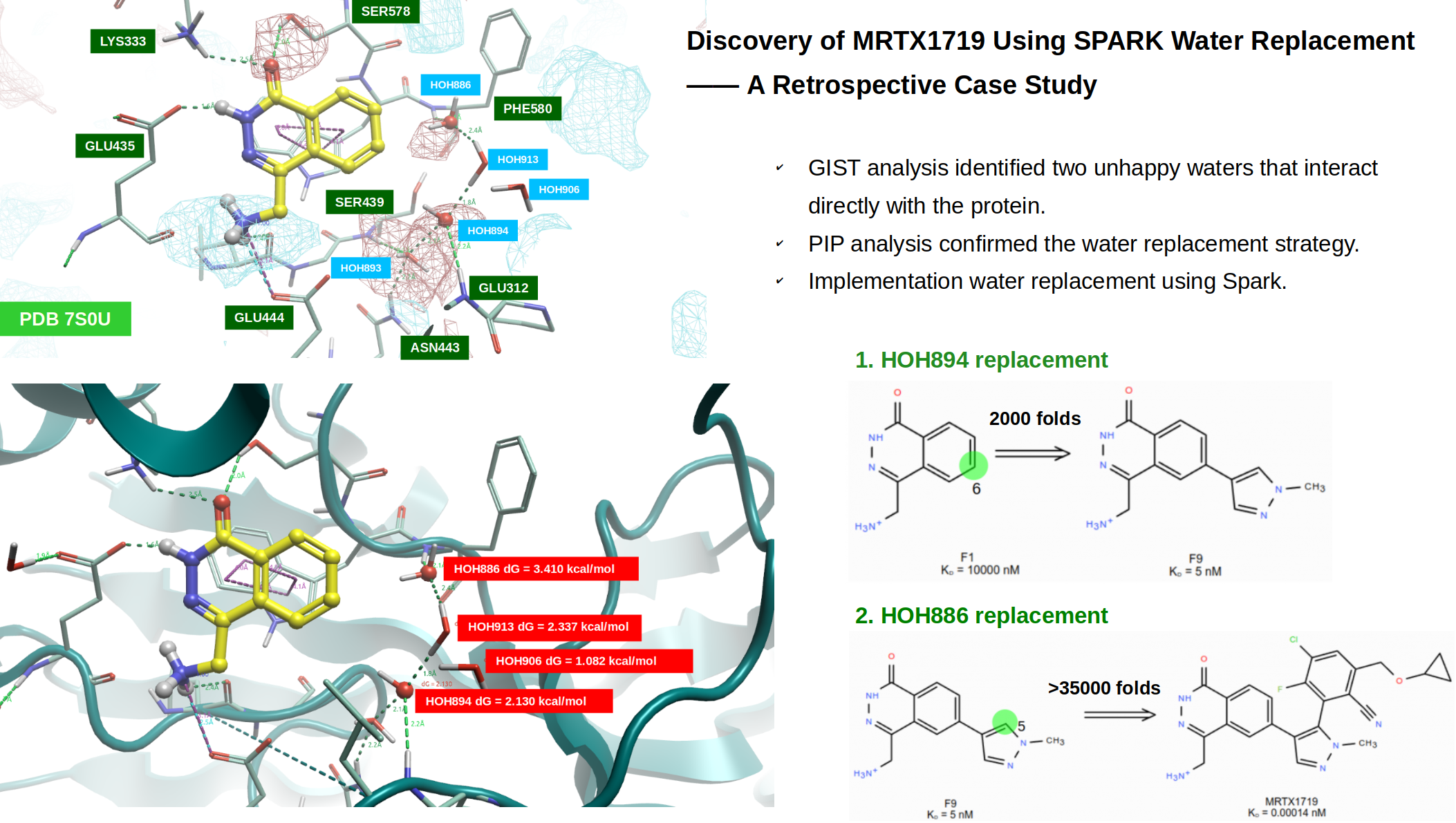
从图2可以看到黄色球棍显示的片段F1与蛋白残基发生了多种强的极性相互作用,比如羰基氧作为氢键受体与LYS333侧链末端质子化的胺基、以及SER576侧链末端的羟基发生氢键相互作用;酰胺-NH作为氢键供体与GLU435的羧基发生氢键相互作用;碱性胺甲基作为正电中心与GLU444侧链末端的羧基发生盐键相互作用;胺甲基作为氢键供体与LEU437的羰基氧发生氢键相互作用。此外F1的共轭芳香环与PHE327的侧链苯环、TRP579的侧链吲哚发生Face-to-Face的芳香相互作用。在F1的苯环那一侧的结合口袋(紫色区域)里有5个水分子,它们形成了氢键网络,其中3个水与蛋白的骨架酰胺NH发生氢键相互作用,这3个水中的2个(球型显示的氧)靠近苯环,这使得这两个水有可能带来较多设计的机会。
图3. PRMT5-MEP50•MTA与F1(PDB 7S0U)以及肽类底物H4(PDB 4GQB)的结合模式比较。飘带:PRMT5•MTA复合物(PDB 7S0U);黄色棍棒:F1(PDB 7S0U);绿色棍棒:H4(PDB 4GQB),为了清晰起见被截短,仅显示在紫色结合口袋的那部分结构。
此前,Antonysamy等人4解释了PRMT5-MEP50•DXU与底物组蛋白H4(histone H4)的共晶结构(PDB 4GQB)。将4GQB下载到Flare里,进行标准的蛋白结构准备、序列比对,然后按Cα原子将4GQB叠合到7S0U,结果发现两个共晶结构的Cα重合非常好,RMSD=0.472。然后,在PDB 7S0U的结合口袋里,观察F1与肽类H4的结合模式,如图3所示。可以发现,F1的碱性胺甲基以及环上酰胺NH片段与H4的胍基片段以相似的模式与PRMT5-MEP50相互作用;此外,而H4的胍基比起F1缺少了的酰胺羰基氧与蛋白的氢键相互作用。在7S0U结合口袋里的5个水分子,除了深处的HOH893之外,其它4个水在与H4结合的过程中被替换了。如图3所示,两个高亮显示的水(氧显示为球型,HOH886与894)直接与蛋白骨架酰胺-NH直接发生氢键相互作用,它们被H4的两个羰基氧替换;另外两个水为HOH906与913,它们不直接与蛋白发生相互作用。HOH906被H4的酰胺NH替换,介导了分子内氢键相互作用;而HOH913被H4疏水片段置换。
图4. PRMT5•MTA与F1(PDB 7S0U)以及肽类(PDB 4GQB)共晶结构的模式比较。飘带:PRMT5•MTA复合物(PDB 7S0U);黄色棍棒:F1(PDB 7S0U);绿色棍棒:肽底物H4(PDB 4GQB)
图4以叠合的方式在PDB 7S0U的口袋里比较了F1与H4的结合模式,可以更清楚地观察到H4羰基氧把两个高亮水HOH886、894替换之后,分别直接与蛋白的PHE580、LEU312酰胺NH发生氢键相互作用。这提示,如果以F1为起点进行地分子生长,可以引入新的基团来模仿H4与蛋白发生这两个氢键相互作用。
蛋白相互作用场(PIP)分析
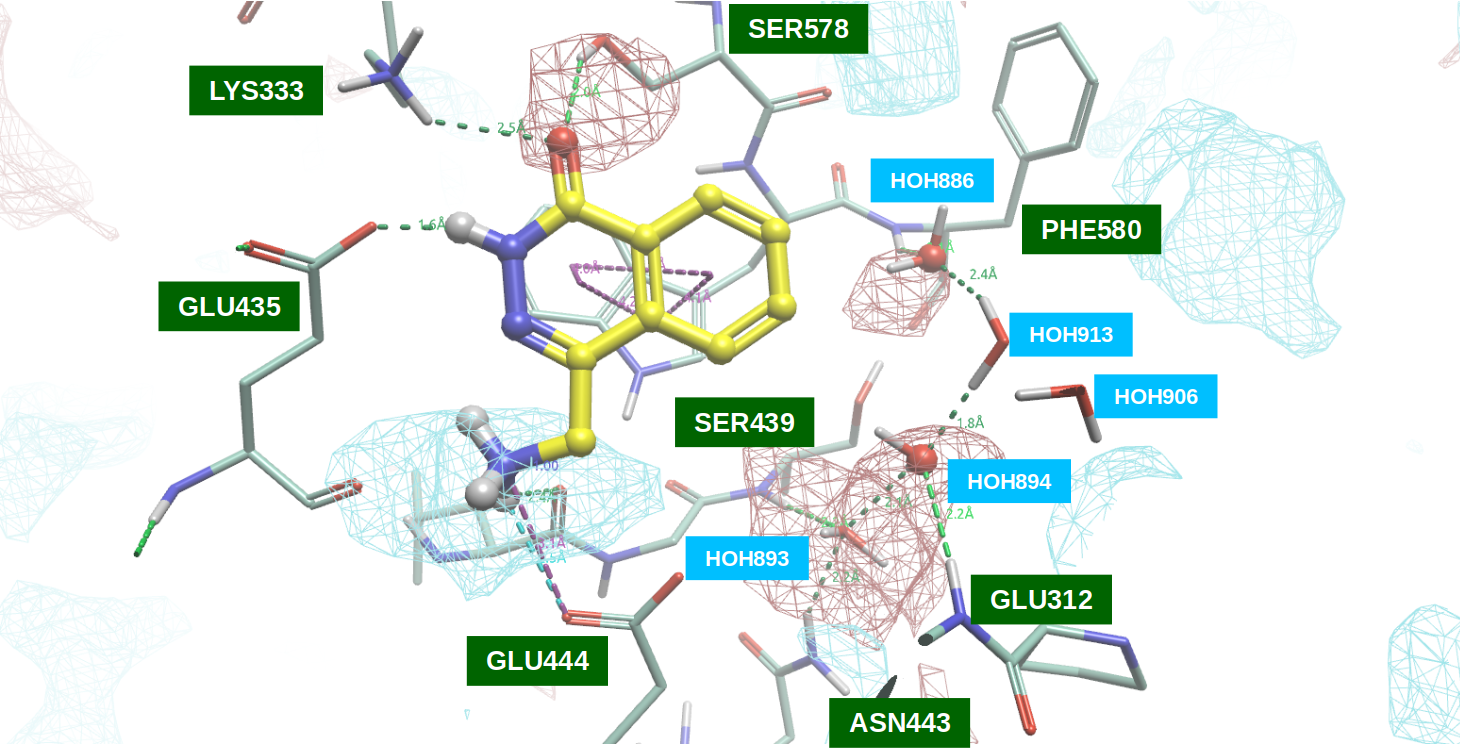
图5. PRMT5•MTA与F1(PDB 7S0U)共晶结构的结合位点的PIP分析。黄色棍棒:F1(PDB 7S0U);蓝色网格:-ev=8.0等值面;红色网格:+ev=7.0等值面。
如图5所示,PDB 7S0U的“干”(不包括结晶水分子或MTA)活性口袋的蛋白质相互作用静电势以令人满意的方式与F1配体场匹配,尤其是:
- 酰胺羰基氧位于活性口袋的正静电相互作用势区域,与LYS333的侧链末端质子化氨基以及SER578的羟基发生氢键相互作用;
- 碱性氮正电中心位于活性口袋的负静电相互作用势区域,与GLU444侧链末端羧基发生盐键相互作用,同时还与GLY438的酰胺羰基氧发生氢键相互作用。
如图5所示,在前一节的分析中,被PDB 4GQB配体H4羰基氧所取代的水分子HOH886和HOH894,位于活性口袋内具有正静电相互作用势的区域。这一发现进一步证实了采用水分子替换策略以在F1上引入新的相互作用基团的有效性和合理性。
GIST水分析
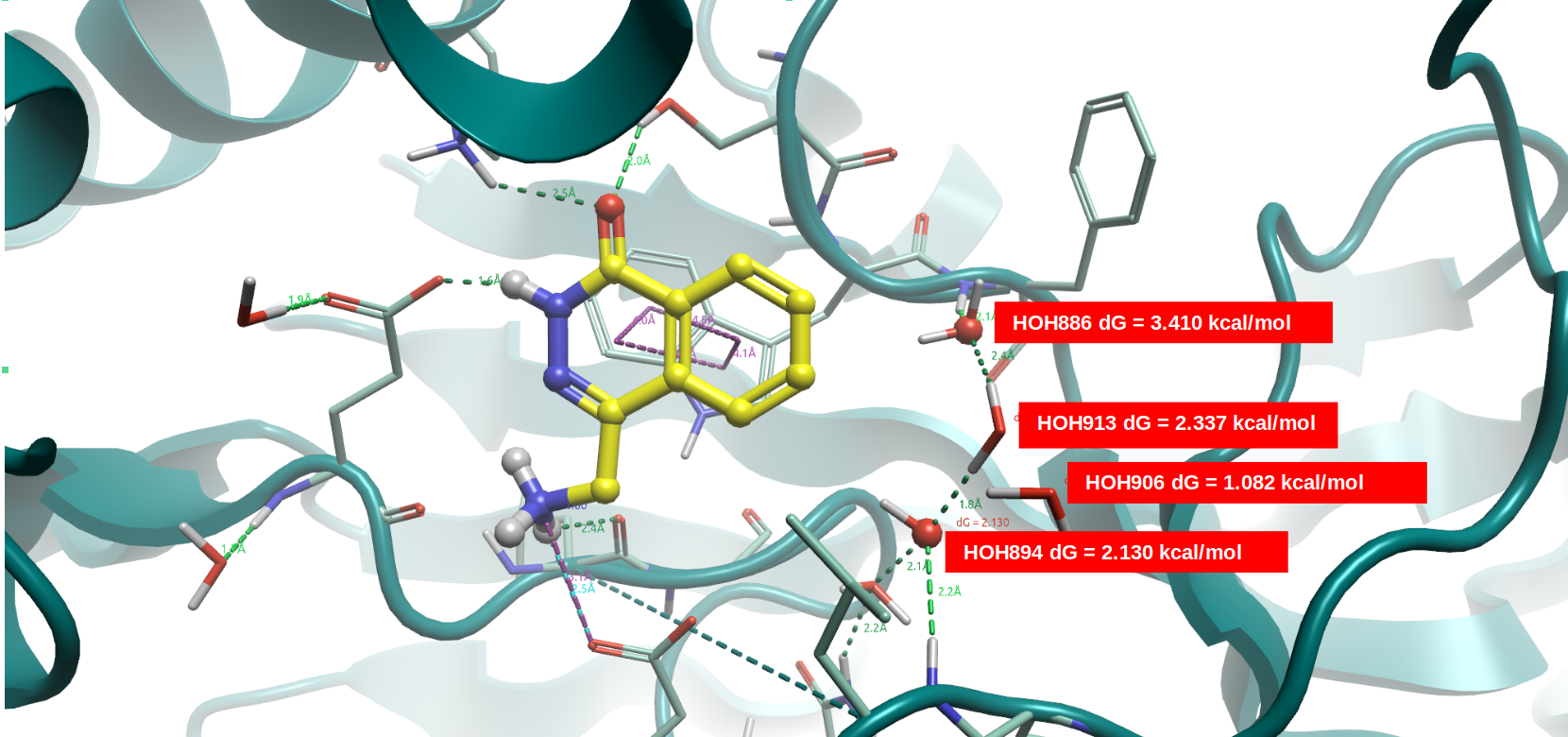
图6. 在PRMT5•MTA与F1(PDB 7S0U)共晶结构结合位点里GIST分析。黄色棍棒:F1(PDB 7S0U);飘带:PRMT5•MTA复合物。
在Flare里对PDB 7S0U的结合位点进行apo-GIST分析,然后计算被PDB 4GQB配体H4替/置换掉的结合水的GIST自由能(见方法部分),结果如图6所示。四个结合水的GIST自由能均为较大的正值,因此可以被新引入的配体置换或替换,这使得这四个水占据的子口袋具有较好的成药性,在与此前的Fpocket成药性打分(drugability=0.77)结果是一致的。鉴于蛋白相互作用场分析表明HOH886与894还位于正静电区域,因此这里引入氢键受体或具有-ev的配体基团将对提高结合亲和力是有利的,或者说对HOH886与894进行水分子替换有机会提高配体的结合亲和力。
HOH894的水分子替换实验
首先对HOH894进行水分子替换实验,计算过程参见方法部分。如图7所示,这个实验通过在F1的6位进行分子生长,引入新的基团,其目的是获得F9的类似物。
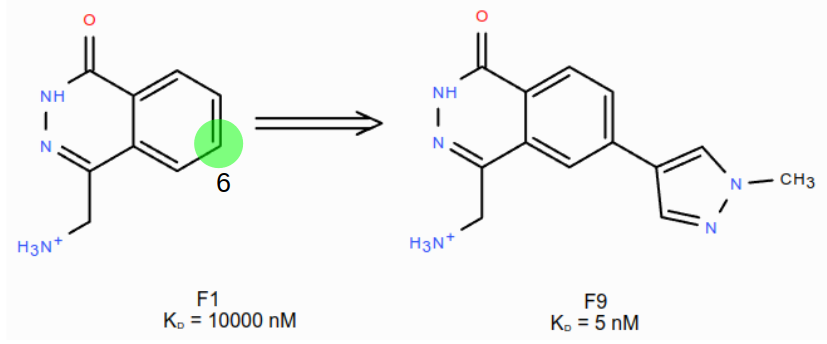
图7. 从F1到F9的分子生长设计
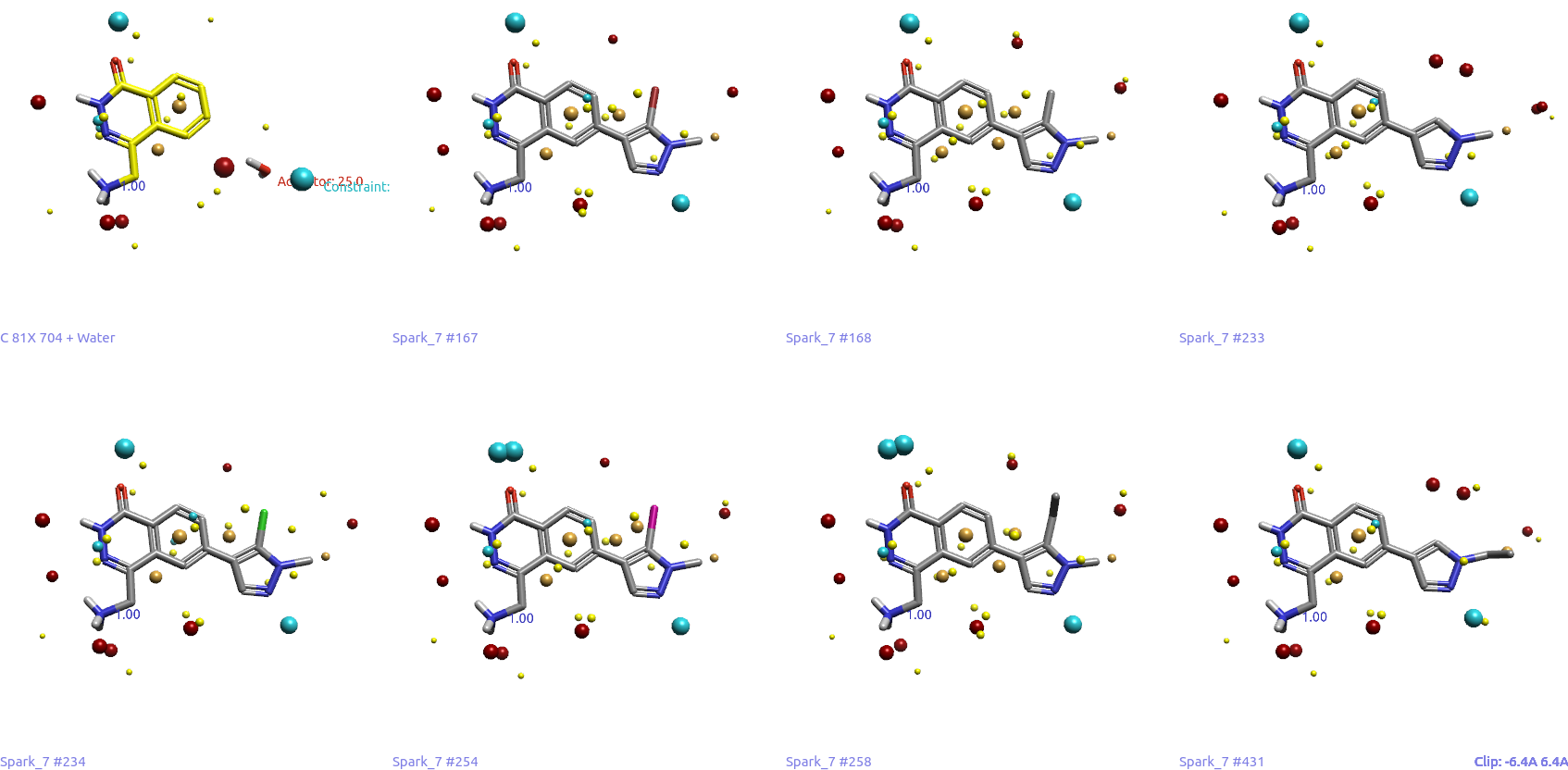
Spark总共生成了500个结果。出于考察Spark能否重现F9设计的目的,用Flare的Filter工具过滤出二氮唑子结构,发现63个结果包含有该子结构,图8给出了其中7个与F9最相似的结构,其中包含了F9本身(Result #233)。
图8. 7个含有二氮唑环的F9类似物,其中#233(右上角)就是F9本身。
所有这7个结构的配体场与起点化合物相似具有相似形状与场点,Sim打分值在0.7-0.75之间,生物等排体指数(BIF)在69-75之间。注意到,二氮唑氮原子附近的蓝色负静电场点模拟了起点分子水氧原子的蓝色场点。
HOH886的水分子替换实验
这个实验的目的是在F9的二氮唑5位碳原子上引入一个2-腈基取代的苯环衍生物,如图9所示。
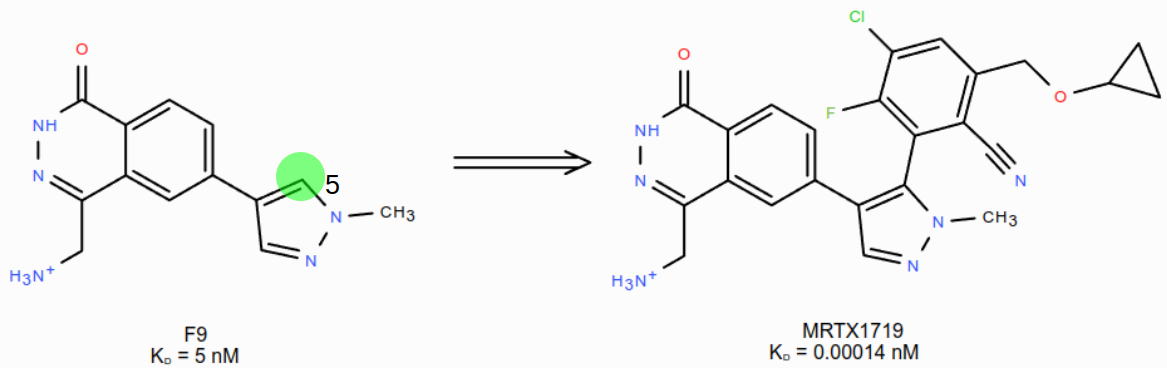
图9. 从F9到MRTX1719的分子生长设计
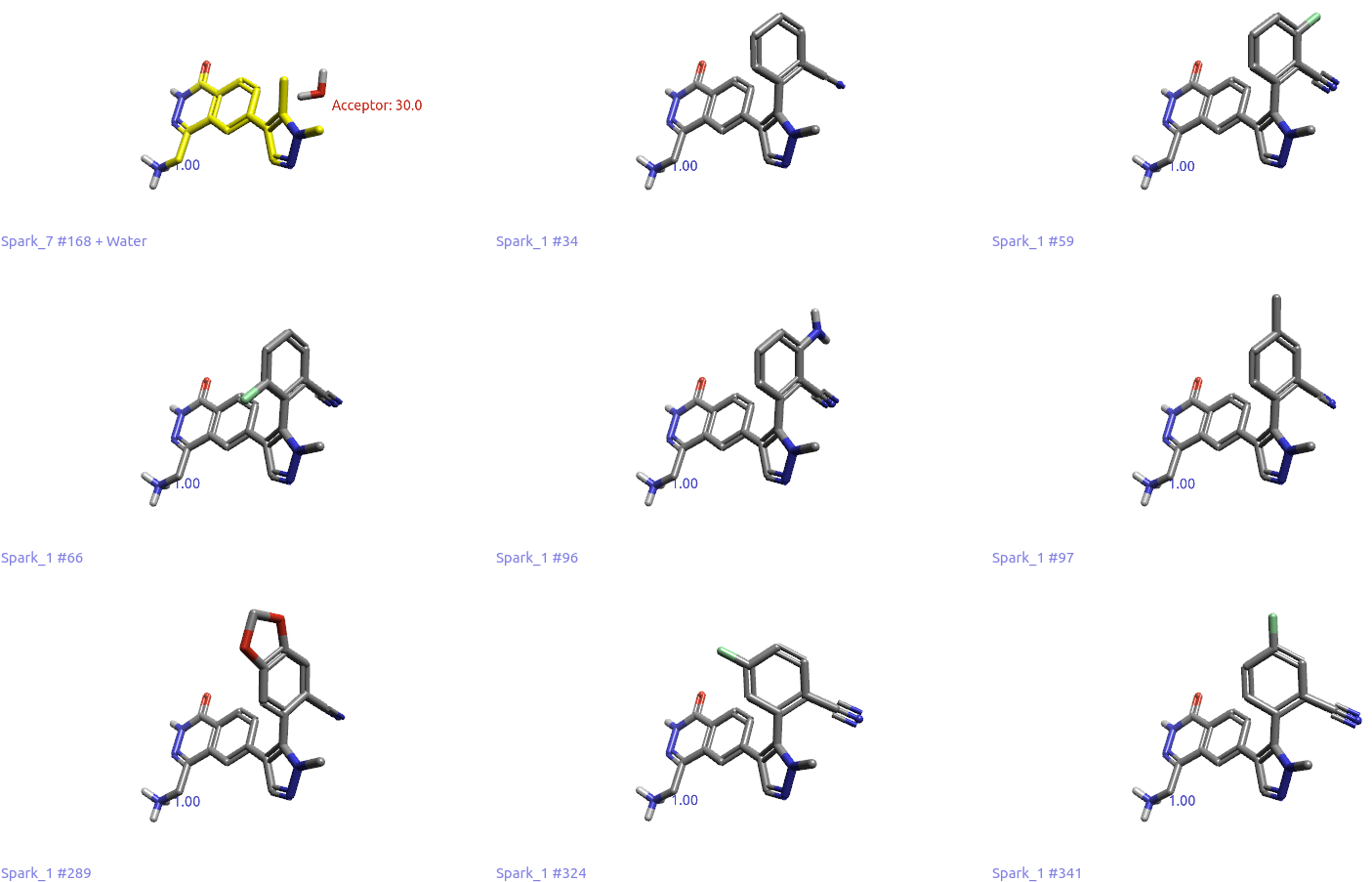
根据我们前面的实验设计,这个“分子生长”是通过对HOH886的水分子替换实验来实现的,计算过程参见方法部分。因此,接下来重点考察Spark的结果能否起到如图9所示的分子生长效果。为此,用Flare的Filter工具过滤出8个含有2-腈基苯的结果,如图10所示。
图10. 8个含有2-腈基苯片段的结果
这8个化合物与起点分子(包含水,图10黄色分子)的形状与静电总相似性在0.84~0.87之间,更重要的是,它们的腈基都模拟了HOH886氧原子作为氢键受体与PHE580主链酰胺NH发生氢键相互作用(没有显示)。其中,Result#34与66是候选药物MRTX1719的子结构(图10),仅在苯环上的取代基有所差异。在这两个结果的提示下,相信药化专家经过进一步的SAR探索,可以发现最终优选的化合物。
方法
蛋白结构准备
根据PDB代码将晶体结构从蛋白质数据库下载到Flare V9中,并使用来自Protein Prep工具小心地准备,以添加氢原子、优化氢键、消除原子冲突并将最佳质子化状态分配给蛋白质结构。任何截短的蛋白质链被封端作为蛋白质准备的一部分。
在Flare中Sequence/align多重比对工具比对蛋白质序列,然后通过Cα的最小二乘拟合进行叠合,结合口袋的比较都是基于这个叠合来实现。
蛋白相互作用场(PIP)分析
蛋白相互作用势是Cresset 分子相互作用势对蛋白质的延伸。两者都是使用XED力场计算的。该方法在原理上类似于配体场的计算:蛋白质的活性位点被探针原子充满,并且计算每个格点上的相互作用势。该方法利用了距离依赖的介电函数来更好地处理蛋白质结构中的大量带电基团。
GIST水分析
用Flare V9的GIST模块对“湿”结合位点Apo结构(不包含配体与金属离子)进行GIST分析,共晶结构的A链水包含在GIST分析里面,具体的GIST分析条件如下:
- Calculation method: Normal
- Ligand: None
- Grid spacing: 0.5 Å
- Grid Definition:Ligand
- Chains: A Chain, A Water
- Simulation length: 20ns
- Solvent Model: explicit TIP4Pew Water
在这个计算中,在分子动力学模拟之前将共晶结构的水链包含在GIST分析里。结合水的GIST自由能通过pyflare的python脚本计算得到5。
HOH894的水分子替换实验
HOH894的水分子替换实验在Flare V9的Spark里实现。Flare V9的一个特性是整合了Spark模块,只需在Flare 3D视窗选中HOH894,然后在Spark菜单里打开水分子替换工作流按钮,就可以设置Spark水分子替换实验,如图11所示。在这个计算里,将F1的6位氢作为作为要被替换的部分,参数如下:
- 连接点的原子类型:Car,Csp2,Nsp3,Nsp2
- Ring constrain:Ring atom only
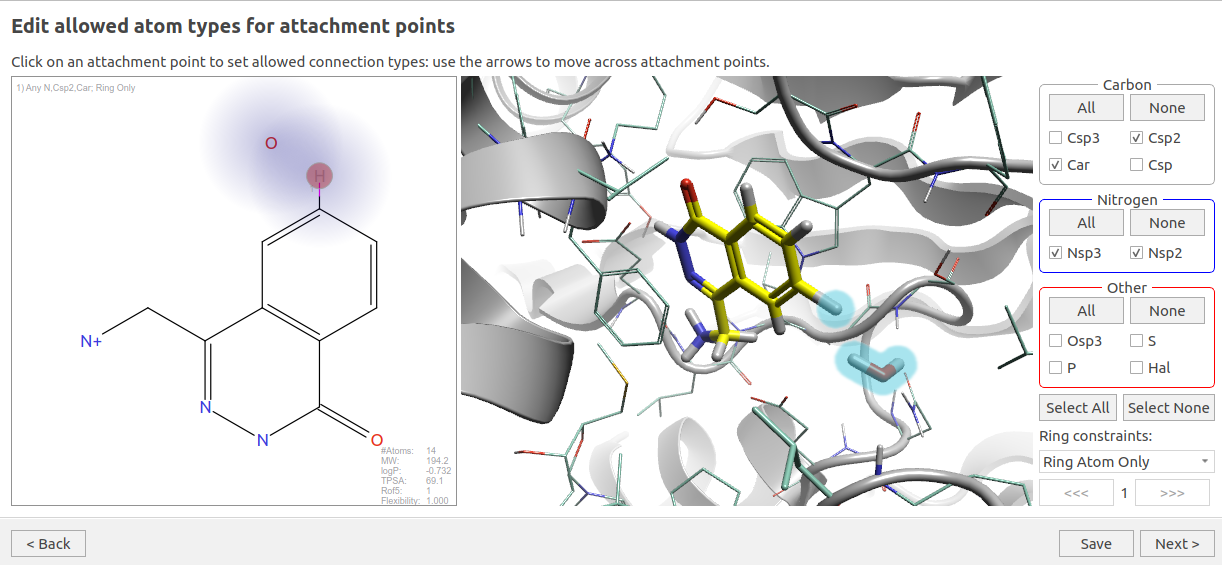
图11. HOH894水分子替换实验
此外,水分子替换实验还设置如图12所示的约束条件,包括:
- HOH894氧原子:氢键受体
- 场点约束:HOH894氧原子附近的-ev场点
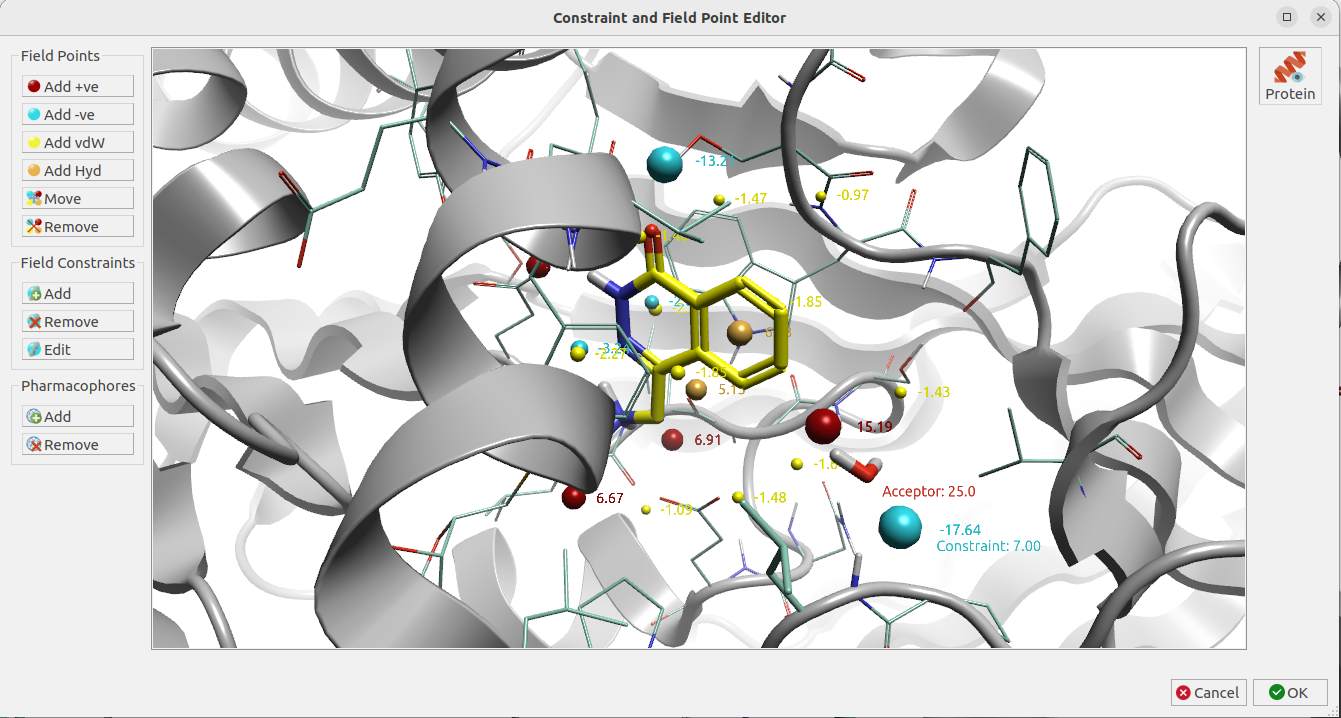
图12. HOH894水分子替换实验的约束条件
本次实验对CHEMBL_common、Commercial_Common与Commercial_VeryCommon等三个数据库共95568个片段进行搜索,如图13所示。
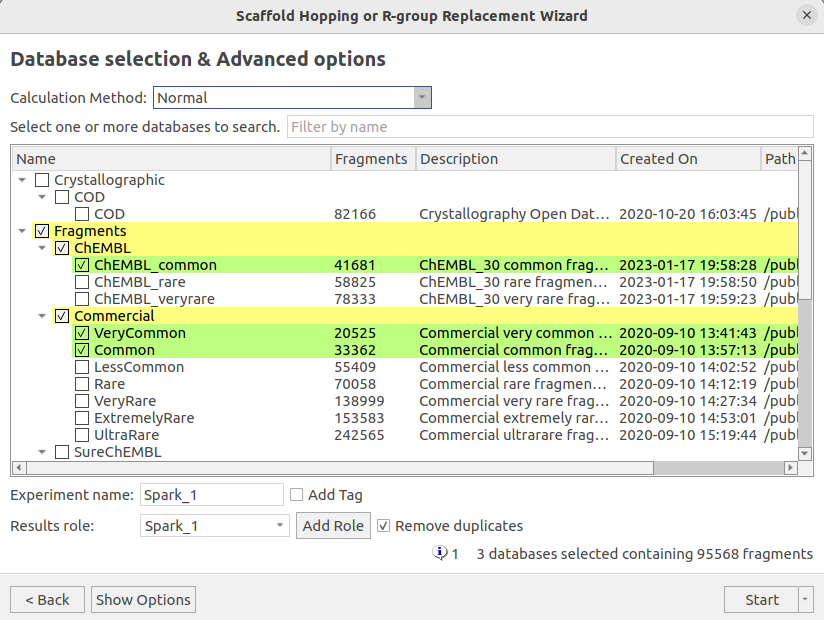
图13. HOH894水分子替换实验的数据库
此外,为了让计算更快地向目标靠拢,在进行数据库搜索时还设置了如下高级参数:
- Fragment Heavy Count:4~10
- Max number of rotatable bound: 3
- Contains a ring: Require
- Contains a hydrogen bond donor: Exclude
- Contains a hydrogen bond acceptor: Require
- Contains Toxophores etc: Exclude
其它参数均为默认值,打分函数采用基于配体的方法,保留打分最高的500个不重复结构。
HOH886的水分子替换实验
在HOH886水分子替换实验中,起点分子不选择图8的结果#233(即F9本身),而是结果#168(图8#223左边那个)。这么作目的是强调F9或是其衍生物是个提示(idea),为负责分子设计的药化专家起到足够的提示作用。在Flare的3D pose里,将#168在准备好的PDB 7S0U口袋里用XED进行配体几何优化,然后以优化过pose与HOH886一起为起点,开始进行水分子替换实验。
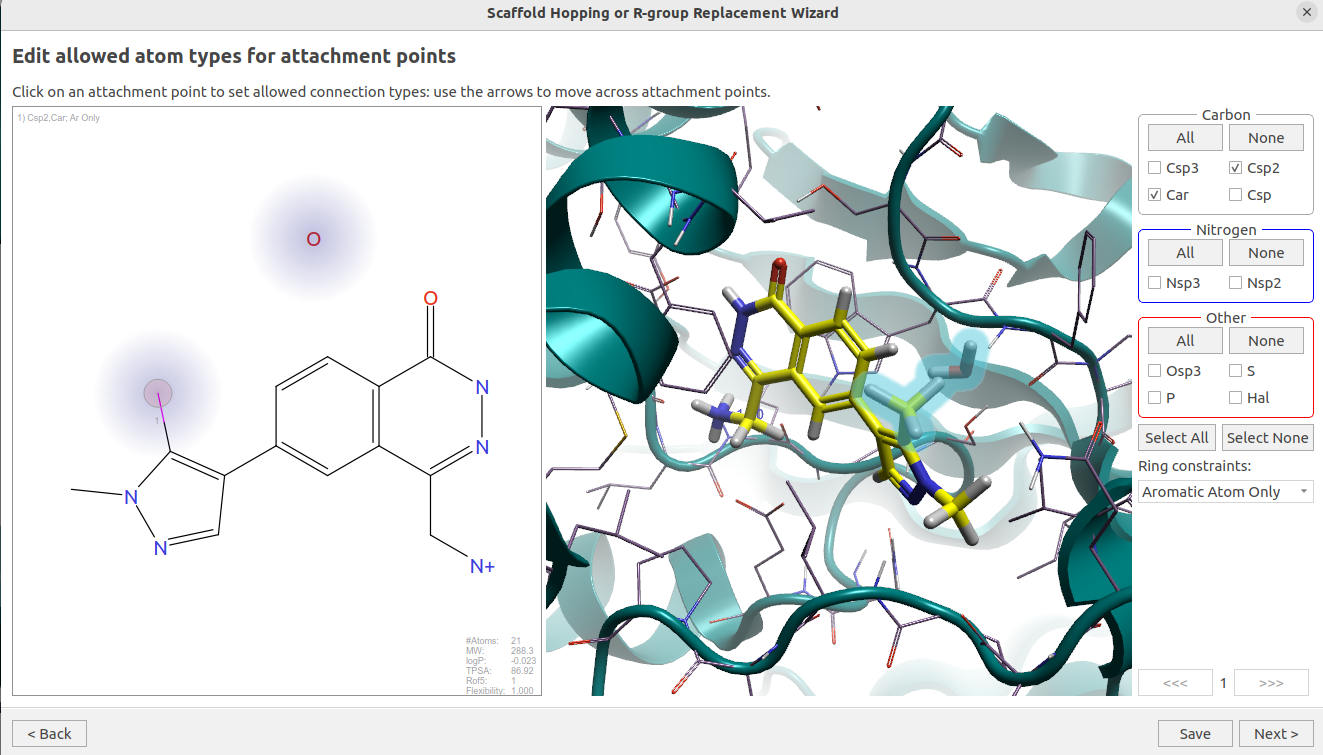
图14. HOH886水分子替换实验
如图14所示,在这个计算里,将#168的二氮唑片段上5位的甲基作为配体要被替换的部分,参数如下:
- 连接点的原子类型:Car,Csp2
- Ring constrain:Aromatic Atom Only
此外,水分子替换实验还在HOH886的氧原子上设置氢键受体约束条件,如图15所示。
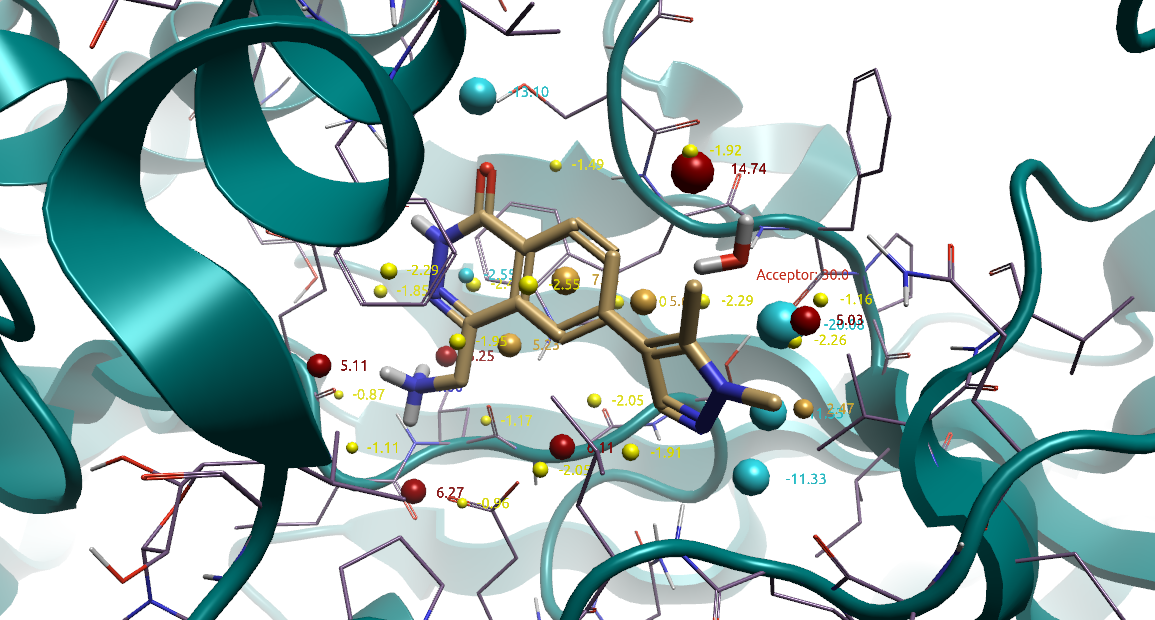
图15. HOH886水分子替换实验的约束条件
本次实验对CHEMBL_common、Commercial_Common与Commercial_VeryCommon等三个数据库共95568个片段进行搜索,如上一节的图13所示。
结论
在本文中,我们利用计算方法重现了新型合成致死药物MRTX1719的基于片段的发现过程。研究始于片段苗头化合物F1与PRMT5•MTA复合物的共晶结构。通过运用Flare软件中的蛋白质相互作用场以及GIST水分子分析技术,我们深入分析了F1所在结合口袋的特性,成功识别出两个具有高能量且潜在可被替换的结合水——HOH894和HOH886。随后,借助Spark的水分子替换模块,针对上述两个水分子位点进行了两轮水分子替换处理,在F1基础上引入了新的基团,从而获得了一系列与目标候选药物MRTX1719高度相似的先导化合物。
鉴于这些计算所得的先导化合物与最终的候选药物极为接近,本研究证明了计算方法能够显著加速药物发现过程,为新型药物的研发提供了强有力的支持。
文献
- Smith, C.R. et al. (2022) “Fragment-Based Discovery of MRTX1719, a Synthetic Lethal Inhibitor of the PRMT5•MTA Complex for the Treatment of MTAP-Deleted Cancers,” Journal of Medicinal Chemistry, 65(3). Available at: https://doi.org/10.1021/acs.jmedchem.1c01900.
- Flare V9. https://www.cresset-group.com/software/flare
- Spark. https://www.cresset-group.com/software/spark
- Antonysamy, S. et al. (2012) “Crystal structure of the human PRMT5:MEP50 complex,” Proceedings of the National Academy of Sciences, 109(44), pp. 17960–17965. Available at: https://doi.org/10.1073/pnas.1209814109.
- 肖高铿. 基于GIST的水合位点分析及其在基于结构设计中的应用. 墨灵格的博客. http://blog.molcalx.com.cn/2024/10/04/gist-based-hydration-site-analysis.html
联系我们
想在自己的项目中使用Flare与Spark,请联系我们获取免费的试用版;或者联系我在线演示Flare与Spark的功能;你还可以采购软件或委托我们进行项目合作:info@molcalx.com。