摘要:Gaussian 09作业文件:本文主要讲解了Gaussian 09的作业(输入)文件格式、组成、内存控制、CPU控制、输出保存、多节点并行的控制等等。
1. Gaussian 09作业文件的准备方法
绝大部分的Gaussian 09作业文件可以通过GaussView来方便的生成,当然也可以通过文本编辑器来建立。
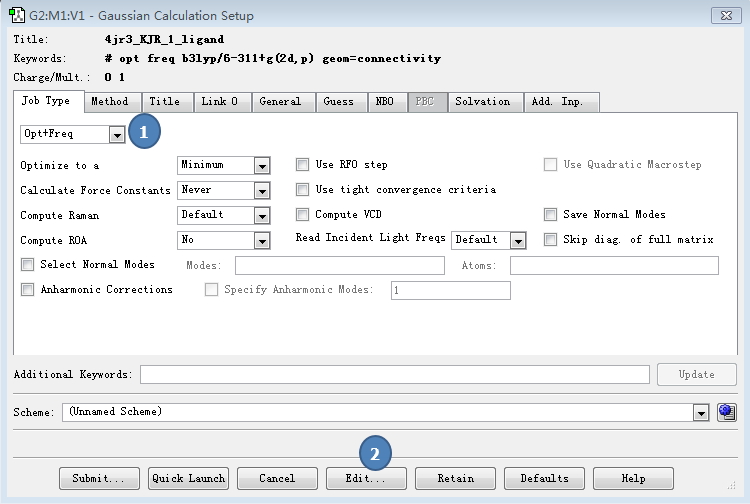
图1. GaussView可以很方便的生成Gaussian的作业文件
图2是Windows版Gaussian 09界面下显示的作业编辑器界面,可以看到它可分为多个不同部分的内容,包括:link0, Route, Title与molecular Specification等等。这些内容与GaussView的编辑器参数相对应。
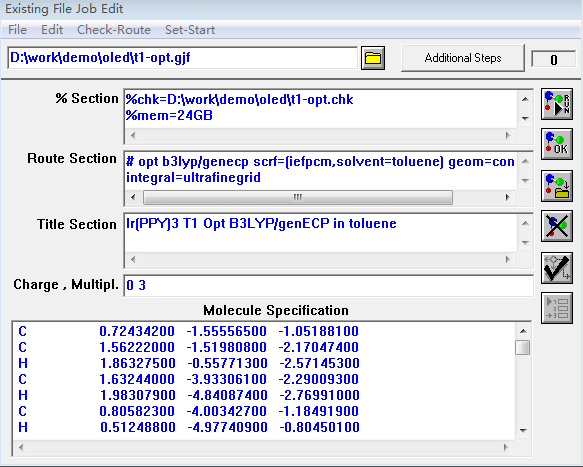
图2. 一个典型的Gaussian作业(截图,并不完整),可以看到它包含了:Link0,Route,Title与molecular Specification等几个部分
Gaussian的作业输入文件通常保存为.com或.gjf后缀的纯本文件,可以用文本编辑器进行修改。
2. Gaussian 09作业文件的特点
Gaussian 09作业文件是纯文本文件,具有如下特点:
- 自由格式、大小写不敏感的
- 空格、Tab键,逗号或反斜杠(/)可以任意组合为一个隔断将一行内的各项分隔,多个空白符号被视为一个隔断
- 感叹号(!)表示注释,可以出现与一行内的任意地方
- 引用一个外部文件的语法为:@filename
比如,Gaussian 09的测试用算例test0000.gjf:
1 2 3 4 5 6 7 8 9 10 11 | # SP, RHF/STO-3G punch=archive trakio scf=conventional Gaussian Test Job 00 Water with archiving 0 1 O H 1 0.96 H 1 0.96 2 109.471221 !上面一行为空白行 |
再比如Gaussian 09的测试用算例test0397.gjf却用了完全不同的风格(注意:节选,只展示部分代码):
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 | !%nproc=4 #p rb3lyp/3-21g force test scf=novaracc Gaussian Test Job 397: Valinomycin force 0,1 O,-1.3754834437,-2.5956821046,3.7664927822 O,-0.3728418073,-0.530460483,3.8840401686 O,2.3301890394,0.5231526187,1.7996834334 O,0.2842272248,2.5136416005,-0.2483875054 O,2.3870396194,3.3004808604,0.2860546915 O,3.927241841,1.9677029583,-2.7261655162 O,2.2191878407,-1.0673859692,-2.0338343532 O,4.209257915,-1.2459024314,-3.1616013359 O,1.7622263124,-1.2194205092,-5.8516071343 O,1.0363626567,3.1095923678,-3.7084594295 O,0.2486786219,0.966590651,-3.8828785542 O,-2.5586288262,-0.0906355161,-1.9360823389 O,-0.3281133832,-2.3890727819,0.2344263336 O,-2.5477902348,-2.7229175024,-0.3031393926 !此处省略了很多行 !上面一行为空白行 |
3. Gaussian 09作业文件的结构
Gaussian作业文件由5个空白行隔断的部分组成:
- Link 0命令:以%开头,可以设置计算资源、进行文件控制以及其它的通用作业控制处理
- Route 部分:以#开头,通过关键词/选项的组合来选用模型化学、计算类型以及其它作业选项
- Title 部分:可以用来简述计算目的,Gaussian计算并不理会这个部分,只是将它在输出里打印出来.这个部分禁用下列字符:@ # ! – _ \ 等控制字符
- 分子描述部分(Molecular Specifications):定义所研究的的分子体系
- 其它输入部分: 某些作业需要,并非全部作业都要该部分
典型的一个作业文件,如下所示:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 | %chk=water.chk %mem=2GB %nprocshared=2 #P HF/6-31G(d) opt=ModRedundant Water geometry Optimization with NBO 0 1 O 0.0 0.0 0.0 H 0.0 0.86 0.65 H 0.0 -0.86 0.65 B 2 3 A !上面空白行不可删除 |
其中:
- 第1-3行: 以%开头,为Link0命令
- 第4行: 以#开头,为Route部分
- 第6-7行: 与Route间有一个空白行分隔,为Title部分
- 第9-12行:为Molecular Specifications部分,与Titile部分有一个空白行分隔;该部分的第一行即第9行指定了体系的电荷与总的自旋多态性。
- 第14行为额外输入部分(Additional Input Section)
- 第15行为空白行,不可缺少
更多的例子,可以从Gaussian 09自带的测试文件里获取。
3.1 Link0 命令部分:作业控制
3.1.1 CPU控制: %NProcShared, %LindaWorkers, %UseSSH
请求双核共享内存并行:
1 | %NProcShared=2 |
分布式内存处理,通过Linda跨节点并行,分别在节点node1,node2,node3请求1、2、4个处理器:
1 | %LindaWorkers=node1,node2:2,node3:4 |
该%LindaWorkers将获得1+2+4=7个处理器计算资源。
组合上述的%NprocShared与%LindaWorkers在一起,获得14核计算资源:
1 2 | %NProcShared=2 %LindaWorkers=node1,node2:2,node3:4 |
3.1.2 内存控制: %Mem
- Gaussian通过设定%Mem的大小来分配内存进行计算
- 增加%Mem的值可以改善计算性能,Gaussian默认分配256MB内存来进行计算
- 多核共享内存并行计算时,%Mem值被%NProcShared值放大进行高效计算
- 分布式内存处理计算,每个Linda计算的CPU分配%Mem的内存
比如,分配4GB的内存:
1 | %Mem=4GB |
1 2 | %Mem=4GB %NProcShared=2 |
注意:上述组合会请求共8GB的内存。
1 2 | %Mem=4GB %LindaWorkers=node3:4 |
上述组合向node3请求了16G计算内存。
3.1.3 文件命名: %CHK, %RWF, %OldChk
检查点文件(Checkpoint file)是计算全程中间数据的存储容器,包含了计算过程与计算结束时的很多信息:
- 几何
- 分子轨道
- 力常数
检查点文件包含的信息可以被不同的关键词去重启计算、或为新计算提供信息:
- %oldchk=filename
- freq=(restart,number)
- geom=check
- geom=allcheck
- guess=read
- opt=readfc
3.1.4 文件保存: %Save, %NoSave
Scratch file在计算中生成:
- 检查点文件: filename[.chk]
- 读-写文件(Read-write file): filename[.rwf]
[.chk],[.rwf]表示后缀.chk,.rwf是可有可无的,尤其是linux等unix类文件系统对后缀没有要求。
默认情况下scratch file存储于$GAUSS_SCRDIR目录,成功计算后会自动被删除。如果想保存,需要自定义存储文件:以全路径的方式添加对应的Link0命令即可:
1 | %chk=path/filename[.chk] |
命名过的scratch file可以用%NaSave自动删除。下面的例子,即使两个文件都显示的加以命名,但是仅检查点文件被保留下来:
1 2 3 | %RWF=/tmp/my_molecule.rwf %NoSave %chk=my_molecule.chk |
3.2 Route部分
Route部分由一组关键组成,这些关键词定义了进行什么样的计算,格式要求:
- 必须以#开头
- 大小写不敏感,没有顺序依赖
- 关键词不可重复出现
- 关键词的可选项用括号分组
- 可以跨行写,意味着多行也是可以的
- 关键词有简写的方式
关键词选项的格式:
- keywork=option 或者 keywork(option)
- keyword=(option1, option2, …)
- keyword(option1, option2, …)
- keyword(option1=value, option2, …)
尽管Route部分没有顺序依赖,但还是推荐下述格式:
1 | # Method/BasisSet Keyword=option |
1 | # Method/BasisSet Keyword(option1=value, option2) |
举例:
1 2 3 4 5 | # SVWN5/3-21G Opt #P MP2/6-31G9d) IRC(Forward, MaxPonits=20) # TPSSTPSS/cc-pVDZ Integral(Grid=400440) # CCSD/GenECP MaxDisk=50GB Opt(RCFC,TS) # CBS-QB3 |
3.3 分子描述(Molecular Specifications)
分子描述由第一行的分子电荷(Molecular Charge)、总自旋多态性(Total Spin Multilicity)以及原子描述及原子核坐标组成:
- 第一行为整数的电荷(charge)与自旋多态性(multiplicity, 2S+1)
- 原子描述由元素符号、分子力场的原子类型及其偏电荷(partial charge),片段编号,PDB残基信息等组成
- 核坐标有多种不同的格式呈现,也可以从检查点文件读入,或者是从GaussView搭建
3.4 额外的输入信息
许多作业类型与关键词需要其它输入信息,通常这些信息是附在分子描述之后,比如:
- SCRF=Read
- IRC=Report
- GenECP
- Pop=NBORead
- Freq=ReadHindered
- CASSCF=SpinOrbit
当这些额外信息输入需要写到作业文件里时,需要注意顺序以及空白行分隔问题,请按照Gaussian用户手册上的顺序表来写。
举个GenECP的例子吧:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 | %mem=24gb %NprocShared=12 %chk=s0_td_triplet.chk #p td(triplet) apfd/genecp integral=ultrafinegrid td(triplet) apfd/genecp 0 1 C 0.72434200 -1.55556500 -1.05188100 C 1.56222000 -1.51980800 -2.17047400 H 1.86327500 -0.55771300 -2.57145300 C 1.63244000 -3.93306100 -2.29009300 [此处的前后有很多行被省略掉] H -2.97322100 4.36785100 2.76465800 C -1.14500800 3.22577300 2.75958700 H -0.69086600 3.73138700 3.60323600 C -0.46906000 2.21075300 2.11186300 H 0.52132600 1.89608800 2.42023000 N -0.85920000 -1.62796200 1.06970900 N -0.98196900 1.55753300 1.06979100 N 1.83862800 0.07126500 1.07165400 Ir -0.00000400 0.00028900 -0.02647500 C 2.00861200 -2.68229600 -2.78061200 H 2.65690600 -2.61967700 -3.65061700 Ir 0 LANL2DZ **** C H N -O 0 6-311+G(2d,p) **** Ir 0 LANL2DZ [此处还有一空白行] |
4. 其它
上述的Gaussian作业文件仅涉部分常见的内容,更多Gaussian作业文件与计算控制请参考:
5. FAQ
问题1: 如何在Gaussian计算后输出wfn文件或如何将Gaussian计算的结果转化为WNF文件。
答:以甲醛的优化与频率计算为例来说明这个问题
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 | %chk=D:\work\demo\wfn\wfn.chk # opt freq hf/3-21g output=wfn Structure: HCHO; Opt Freq at hf/3-21g level. 用output关键词加wfn选项可以让Gaussian输出WFN文件 同时, 需要在molecular specification结尾添加输出文件的名称(注意空白行) 0 1 C -2.43735772 0.30751708 0.00000000 O -1.21004072 0.30751708 0.00000000 H -3.02950272 1.24692108 0.00000000 H -3.02950272 -0.63188692 0.00003900 D:\work\demo\wfn\wfn.wfn !上面有行空白,不要忽略 !感叹号是告诉Gaussian,这是注释行,请Gaussian在读入时忽略该行 !第12、14行为空白行 !第13行为生成wfn文件的文件名与路径 |
6. 申明
如果发现错误请联系我们,我们很愿意在您的指导下改正:info@molcalx.com。



