通过高效的计算机辅助设计与优先级排序加速 METTL3 抑制剂的发现
摘要:RNA甲基转移酶METTL3通过在mRNA上写入表观遗传标记N6-甲基腺苷(N6-methyladenosine,m6A),参与调控急性髓系白血病(AML)及诸如卵巢癌(SK-ov-3)、头颈癌(FaDu)和肺癌(A549/NSCLC)之类多种实体瘤的发生发展。本文描述了化合物EP652的发现与优化过程,该化合物被选为体内概念验证分子,旨在抑制METTL3活性。通过对药代动力学(PK)性质的系统性优化,EP652经腹腔注射(IP)给药后在临床前肿瘤模型中展现出显著疗效,从而验证了药理学抑制m6A这一表观遗传标记作为新型抗癌治疗策略的可行性。

原文:Martin Slater. December 2, 2025. Accelerating discovery of a METTL3 inhibitor through efficient in silico design and prioritization
Cresset Discovery 拥有先进的分子建模专长,采用其自有的计算机模拟方法,结合体外至体内实验,推动药物发现中苗头化合物的高效识别与后续优化。最近发表的一篇论文1 记录了 Cresset Discovery 与 Epics Therapeutics 的合作成果。本文重点总结了 Cresset Discovery 在蛋白-配体相互作用分析、基于片段替换的新型分子设计与优化,以及构建预测模型以对各迭代方案进行优先级排序的工作,显著加速研发DMTA循环,加快了苗头化合物向先导化合物的转化进程。
前言
甲基转移酶样蛋白3(methyltransferase-like 3,METTL3)是mRNA序列的主要修饰酶,特异性地作为m6A表观遗传标记的“写入者”,该标记与肿瘤相关调控蛋白的命运及肿瘤发生密切相关。该酶以异源二聚体形式发挥作用,其中METTL3为催化结构域,而其伴侣蛋白甲基转移酶样蛋白14(METTL14)则作为RNA结合的底物识别元件。与其他甲基转移酶类似,METTL3通过同时结合辅因子S-腺苷甲硫氨酸(SAM,可视为Me+合成子)和底物来催化反应。一旦形成合适的甲基转移几何构型,即释放S-腺苷高半胱氨酸(SAH)和甲基化产物——此处为m6A标记的RNA序列。该靶点具有明确的可成药性,天然产物小分子抑制剂sinefungin(一种非选择性METTL3抑制剂)的存在即已证明,尽管其选择性不足,但为开发靶向该甲基转移酶的小分子药物提供了先例。
苗头化合物的识别与优化策略
在分析现有METTL3晶体结构数据之后,我们初步认为仅通过计算机模拟SAM(辅因子)结构难以实现靶点选择性,因此需要同时模拟SAM和底物的结合特征。然而,蛋白结构中固有的环区柔性为此类设计带来挑战。除该策略外,我们还开展了高通量筛选(HTS)和新型虚拟筛选,但均未能获得可用于药物开发的起始苗头化合物,具体原因在此不赘述。
在Caflisch团队2首次报道非核苷类METTL3抑制剂(图1)后,以及随后发表的系列研究3-5、Storm Therapeutics公司的报告与专利6-8和综述9等进一步证实了我们最初思路的合理性。值得注意的是,这些抑制剂的核心区域(图1中以青色标示)与底物结合位点重叠,表明其作用机制并非单纯的SAM竞争性抑制。早期数据及抑制剂的X-衍射晶体结构揭示了关键的适应性环区构象(adapted loop configurations),使我们得以理解当前所有抑制剂的几何空间需求,并据此采用基于配体的设计理念。
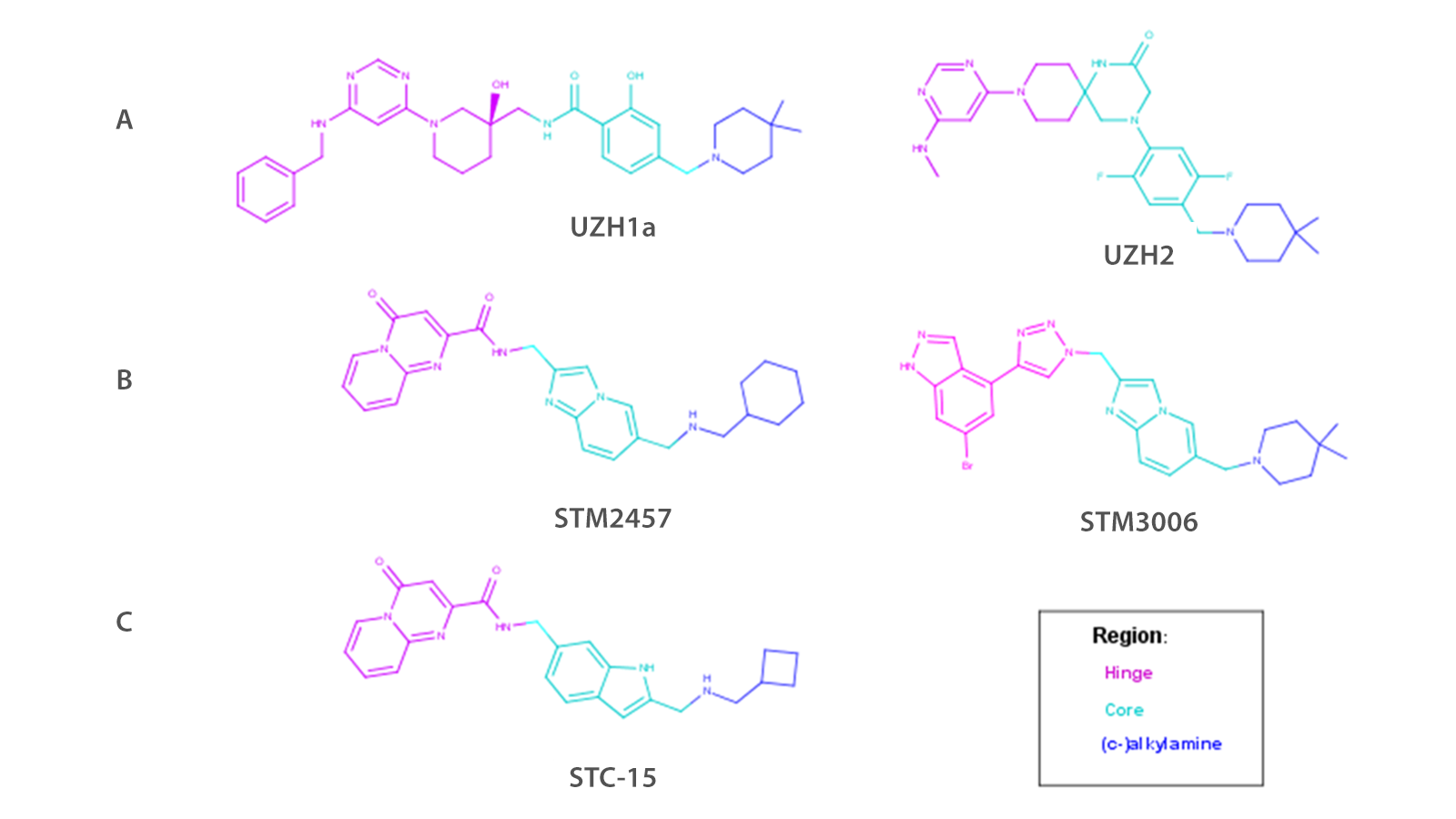
图1. 一些已知的METTL3/14抑制剂结构,源自:(A) Caflisch团队,(B) Storm Therapeutics,(C) Storm公司披露的临床候选化合物10。颜色编码表示构效关系(SAR)中对应的化学子区域。
如图2所示,我们系统分析了已知抑制剂的构效关系及其晶体结构。利用Cresset公司专有软件FlareTM 11 进行蛋白与配体的叠合比对,构建了预测性QSAR模型,并将其作为配体设计优先级排序的重要工具。基于结构和配体的设计思路,均以诸如对接11和静电互补性(Electrostatic ComplementarityTM 12 )之类的计算机打分指标为指导。通过Cresset专有工具SparkTM 14 进行片段替换,探索了替代性分子骨架。所有设计方案均经过计算机多参数理化性质评估,以确定最优候选结构。最终,药物化学团队通过评估合成可行性(即“化学路线预研”)作为每轮“设计-合成-测试-分析”(Design-Make-Test-Analyze,DMTA)循环前的最终决策依据。
将苗头化合物系列逐步优化至具备所需细胞活性(以及后续的体内活性)的过程,是一次系统性的化学探索与多参数优化实践,需在细胞活性(如细胞内摄取能力)与清除率、代谢稳定性等竞争性因素之间取得平衡。在此过程中,Kasumi-1细胞活性数据结合logP/logD计算所得的亲脂性配体效率(LLE)成为一项极为有效的优化指标。
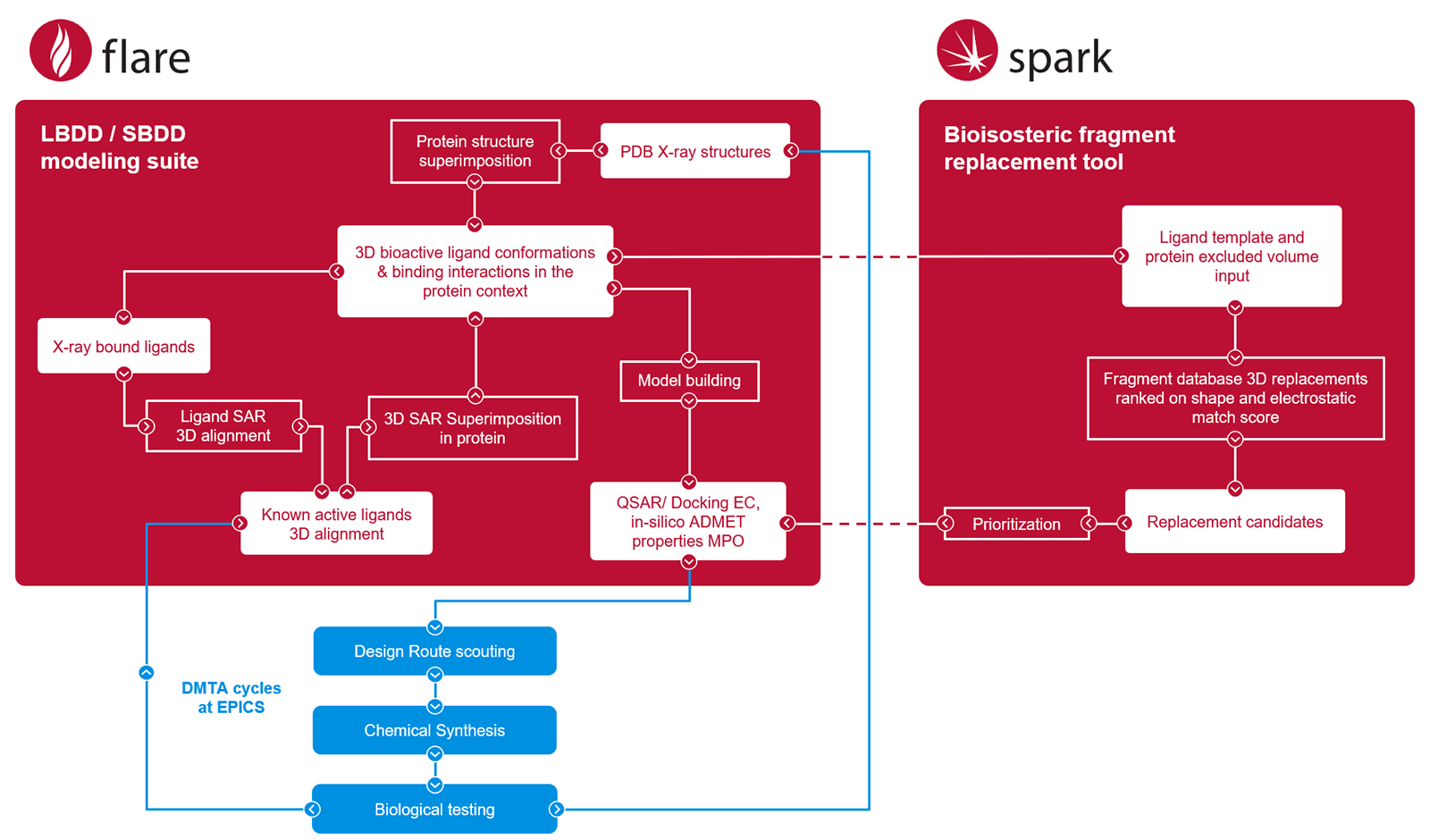
图2. 苗头化合物发现与开发的模拟工作流程
结果与讨论
结合上述计算机辅助设计与药物化学工作流程,我们获得了8个结构多样的化合物并推进至合成与测试阶段。初始化合物在酶学SPA检测中均表现出可测量的活性(IC50范围为2 nM 至2.4 µM),其中R构型的化合物1活性最优(2 nM)。随后对化合物1进行了全面的活性与药代性质表征,并完成了其与METTL3/14复合物的X-衍射晶体结构解析(见图3)。

图3. 化合物1在METTL3/14中的共晶结合模式
尽管该初始的苗头化合物在SPA酶学检测中活性强,在ATPlite细胞检测中表现中等,但在大鼠肝微粒体中代谢极快。此外,其对细胞色素P450酶(CYP3A4,IC50 = 0.8 µM)具有强抑制作用,若不进一步修饰则难以推进。后续的药物化学优化工作(详见全文)最终成功获得了优化了的候选分子 EP652。该分子在AML及特定实体瘤的小鼠模型中经腹腔给药后展现出显著的体内活性。
对已有化学型三维构象及电子特征的保持(如图4所示),体现了以该结构为基础,通过整合计算机辅助设计与药物化学迭代优化,在先导化合物优化中有效拓展新化学空间、发现新型化学型并指导其合成与测试优先级排序的策略价值。
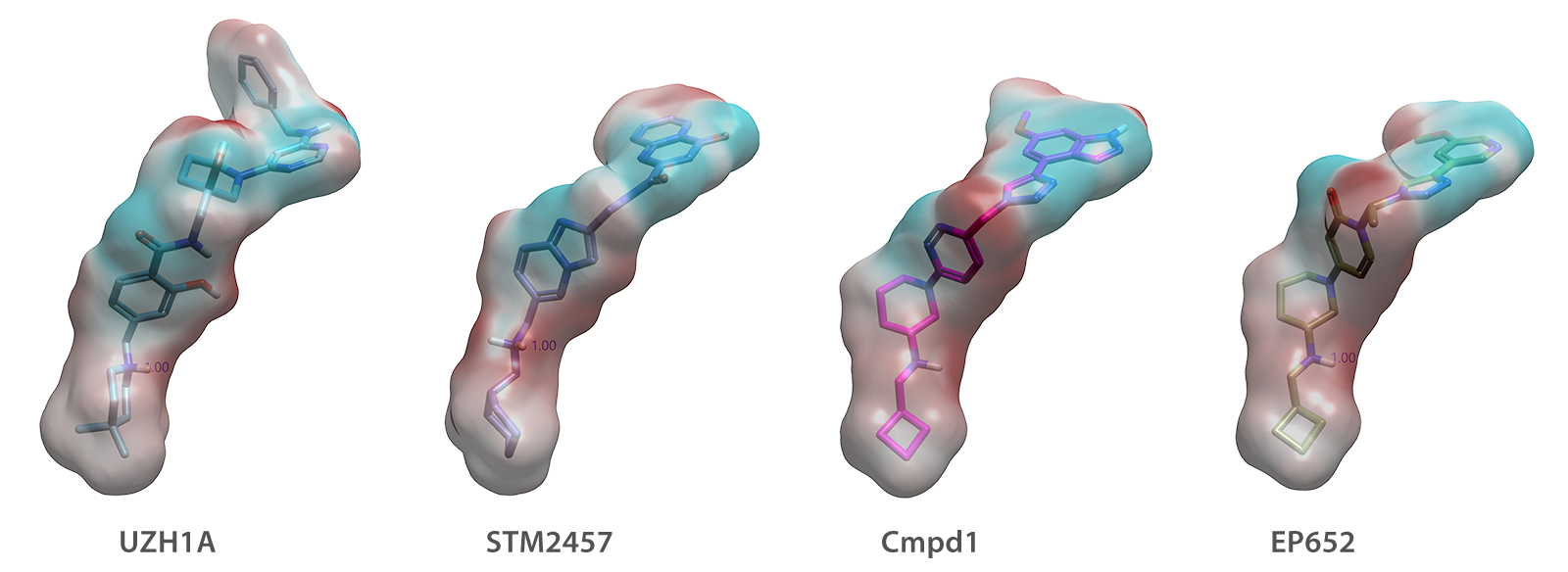
图4. UZH1A、STM2457、Compd1*、EP652在METTL3/14中的共晶结合模式叠合图(PDB代码分别为7ACD、7O2I、*未发表、9G4U)。在分子等势面中,青色代表负电区域,红色代表正电区域。
总结
我们展示了一种高度融合药物化学专业知识的高效计算机辅助工作流在苗头化合物的发现与优化中发挥了关键作用,显著加速了向候选药物分子的推进进程。
更多详情请参阅原始文献1:J. Med. Chem. 2025, 68(3), 2981–3003。
作者介绍

Martin Slater
Martin Slater博士在利兹大学和哈德斯菲尔德大学主修药物化学。1997年,他加入当时刚创立的BioFocus公司,担任高级研究员,通过开发创新的化学基因组学工具,以及设计出40多个在商业上取得成功的靶向蛋白质化合物库,为SoftFocus化合物库品牌奠定了坚实基础。马丁率先将Cresset公司的基于场的技术应用于G蛋白偶联受体(GPCRs)、激酶(Kinases)、离子通道(Ion Channels)和蛋白酶(Proteases)等靶点,用于化合物库构建和从头配体设计。他于2011年加入Cresset公司,现任发现部门总监,为制药、生物技术、农用化学品、学术界以及香精香料等领域的客户提供高质量的计算化学与分子模拟咨询服务。
文献
- Dutheuil, G., Oukoloff, K., Korac, J., Lenoir, F., El Bousmaqui, M., Probst, N., Lapin, A., Nakhabina, G., Sorlet, C., Parmentier,N., Karila, D., Ghavtadze, N., Casault, P., Claridge, S., Sapmaz, S., Slater, M.J., and Fraser, G.L. Discovery, Optimization, and Preclinical Pharmacology of EP652, a METTL3 Inhibitor with Efficacy in Liquid and Solid Tumor Models, J Med Chem 2025, Vol 68, Issue 3, 2981-3003 https://pubs.acs.org/doi/10.1021/acs.jmedchem.4c02225
- Moroz-Omori, E. V., Huang, D., Bedi, R.K., Cheriyamkunnel, S. J., Bochenkova, E., Dolbois, A., Rzeczkowski, M.D., Li, Y., Wiedmer, L., Caflisch, A. METTL3 inhibitors for epitranscriptomic modulation of cellular processes. ChemMedChem 2021, 16, 3035-3043.
- Bedi, R.K., Huang, D., Eberle, S.A., Wiedmer, L., Śledź, P., Caflisch, A. Small-molecule inhibitors of METTL3, the major human epitranscriptomic writer. ChemMedChem 2020, 15 (9), 744-748.
- Bedi, R.K., Huang, D., Li, Y., Caflisch, A. Structure-based design of inhibitors of the m6A-RNA writer enzyme METTL3. ACS Bio. Med. Chem. Au 2023, 3 (4), 359–370.
- Dolbois, A., Bedi, R.K., Bochenkova, E., Müller, A., Moroz-Omori, E.V., Huang, D., Caflisch, A. 1,4,9-Triazaspiro[5.5]undecan-2-one derivatives as potent and selective METTL3 inhibitors. J. Med. Chem. 2021, 64 (17), 12738-12760; Caflisch, A., Dolbois, A., Huang, D., Zalesak F. N6-adenosine-methyltransferase inhibitors in cancer treatment. PCT International Application WO 2022/243333 A1, May 17, 2022; Bedi, R.K., Huang, D., Li, Y., Caflisch, A. Structure-based design of inhibitors of the m6A-RNA writer enzyme METTL3. ACS Bio. & Med. Chem. Au 2023, 3 (4), 359-370.
- Yankova, E., Blackaby, W., Albertella, M., Rak, J., De Braekeleer, E., Tsagkogeorga, E., Pilka, E.S., Aspris, D., Leggate, D., Hendrick, A.G., Webster, N.A., Andrews, B., Fosbeary, R., Guest, P., Irigoyen, N., Eleftheriou, M., Gozdecka, M., Dias, J.M.L., Bannister, A.J., Vick, B., Jeremias, I., Vassiliou, G.S., Rausch, O., Tzelepis, K., Kouzarides, K. Small molecule inhibition of METTL3 as a strategy against myeloid leukemia. Nature 2021, 593, 597−601.
- Guirguis, A.A., Ofir-Rosenfeld, Y., Knezevic, K., Blackaby, W., Hardick, D., Chan, Y., Motazedian, A., Gillespie, A., Vassiliadis, D., Lam, E.E.N., Tran, K., Andrews, B., Harbour, M.E., Vasiliauskaite, L., Saunders, C.J., Tsagkogeorga, G., Azevedo, A., Obacz, J., Pilka, E.S., Carkill, M., MacPherson, L., Wainwright, E.N., Liddicoat, B., Blyth, B.J., Albertella, M.R., Rausch, O., Dawson, M.A. Inhibition of METTL3 results in a cell-intrinsic interferon response that enhances antitumor immunity. Cancer Discov. 2023, 13 (10), 2228–2247.
- Blackaby, W., Hardick, D., Thomas, E., Bookfield, F.A., Bubert, C., Shepherd, J., Ridgill, M.P. Preparation of imidazopyridines and related derivatives as MeTTL3 inhibitors. WO 2020/201773 A1, Oct 08, 2020; Blackaby, W., Hardick, D., Thomas, E., Bookfield, F.A., Shepherd, J., Bubert, C., Ridgill, M.P. Preparation of polyheterocyclic compounds as METTL3 inhibitors. WO 2021/111124 A1, Jun 10, 2021; Hardick, D., Blackaby, W., Thomas, E., Bookfield, F.A., Shepherd, J., Bubert, C., Ridgill, M.P. Preparation of heteroaromatic compounds as METTL3 inhibitory compounds. WO 2022/074379 A1, Apr 14, 2022; Hardick, D., Blackaby, W., Thomas, E., Bookfield, F.A., Shepherd, J., Bubert, C., Ridgill, M.P. Preparation of nucleosides as inhibitors of N6-adenosine-methyltransferase METTL3. WO 2022/074391 A1, Apr 14, 2022; Bubert, C., Ridgill, M.P., Canelli, T., Hardick, D. Processes for the preparation of inhibitory compounds. WO 2022/254218 A1, Dec 08, 2022; Bayle, E.D., Bucknell, S.J., Thomas, E.J.Hardick, D.J., Harper C., Byrne, D., Shepherd, J., Ridgill, M.P.Inhibitory compounds. WO 2024/126897 A1, Dec 12 2024.
- Fiorentino, F., Menna, M., Rotili, D., Valente, S., Mai, A. METTL3 from Target Validation to the First Small-Molecule Inhibitors: A Medicinal Chemistry Journey, J. Med. Chem. 2023, 66 (3), 1654-1677; Li, H., Zhang, Q., Feng, Q., You, Q., Guo, X. The development of small molecules targeting methyltransferase-like 3, Drug Discovery Today 2023, 28 (4), 103513.
- https://www.stormtherapeutics.com/site/assets/files/1460/storm-asco-poster-72×42-may24-v5b_final.3w27wb.pdf
- Flare™, version 10, Cresset®, Litlington, Cambridgeshire, UK; https://cresset-group.com/flare/; Cheeseright T.; Mackey, M.; Rose, S.; Vinter, A. Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation. J. Chem. Inf. Model. 2006, 46, 665-676.
- Lead Finder™, BioMolTech®, Toronto, Ontario, Canada; https://cresset-group.com/software/leadfinder.
- Bauer, M. R.; Mackey, M. D. Electrostatic Complementarity as a Fast and Effective Tool to Optimize Binding and Selectivity of Protein-Ligand Complexes. J. Med. Chem. 2019, 62, 3036-3050.
- Spark™, Cresset®, Litlington, Cambridgeshire, UK; https://cresset-group.com/software/spark/; Cheeseright, T., Mackey, M., Rose, S., Vinter, A. Molecular field extrema as descriptors of biological activity: definition and validation. J. Chem. Inf. Model. 2006, 46 (2), 665-676.



