摘要:Flare是Cresset基于结构与基于配体的药物设计平台,Flare V6即将在6月份发布。此次先睹为快介绍了三个计算化学科研人员感兴趣的新功能:结合口袋预测、小分子QM计算以及将图形界面计算参数导出为命令行计算所需的文件。
Flare™是一款基于配体与基于结构的药物设计平台,此次先睹为快让我们深入了解一些即将发布的Flare V6中的新功能。
结合口袋预测
识别小分子配体潜在的成药性结合位点是新靶标和先导化合物发现分子建模的关键步骤。Flare V6新增了基于 fpocket[1]的结合口袋预测模块,这是一种快速的蛋白口袋检测算法,用于识别、表征在蛋白结构内部或其表面上的口袋与空腔。
结合口袋识别方法的工作原理是用不同大小的“α-球”填充到蛋白结构中所有潜在的结合口袋和空腔:这些球在3D空间中接触四种原子,但不包含任何内部的原子。可以定位出结合位点最令人感兴趣的区域,例如蛋白质表面的裂缝,通常会出现较大的α-球体。下一步是寻找足够大的具有适当半径的α球簇。微调α球的最小/最大半径、填充口袋的最小球数、球簇的最大尺寸,可以搜索到不同类型的结合口袋,例如药物结合位点、水结合位点口袋、通道和大的溶剂暴露位点。可以根据球体周围原子的特性(例如疏水性)进一步表征α球簇,以帮助识别出具有成药性的结合位点。
图1显示了对KEAP1(PDB 4IFJ)apo结构进行的结合口袋预测实验的结果,该结构是NRF2介导的细胞保护反应的关键调节因子,人们日益认为这是氧化应激疾病的靶标。
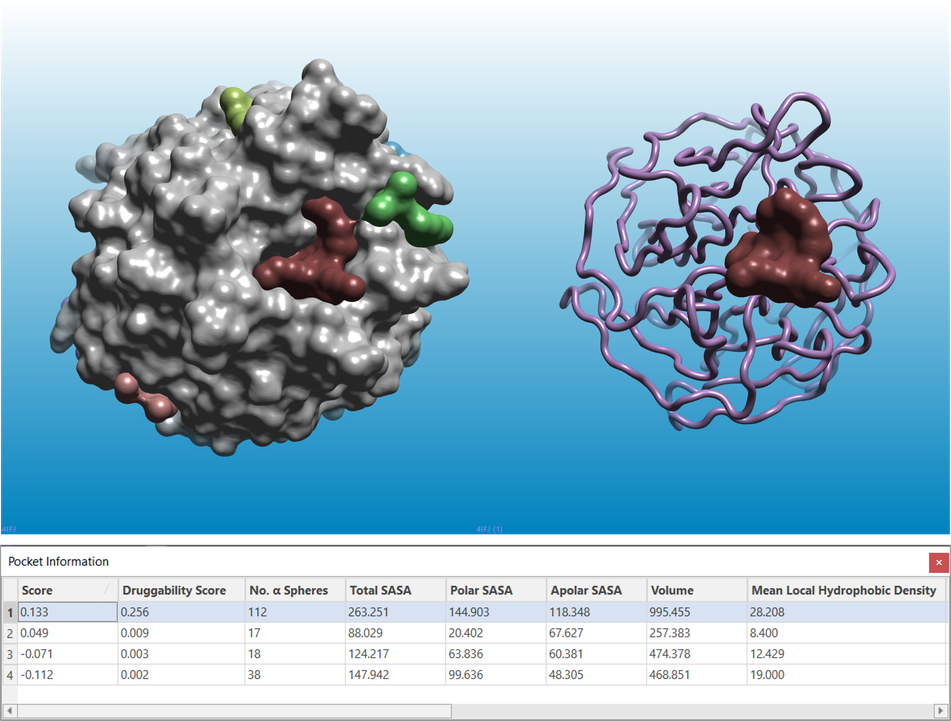
图1. 对KEAP1(PDB 4IFJ)的apo蛋白结构进行口袋预测的实验结果。左:KEAP1蛋白的溶剂排除表面显示为灰色。右图:KEAP1蛋白结构显示为粉色飘带。在这两张图片中,口袋都显示为多种颜色的表面。口袋信息表(底部)根据许多不同的参数报告口袋的特征。
计算打分和成药性打分最高的口袋(图 1和2中的棕色)是NRF2肽(PDB 4IFL,图2-左)和小分子抑制剂[2](例如PDB 5FNU配体)所在的结合位点(图2-右)。
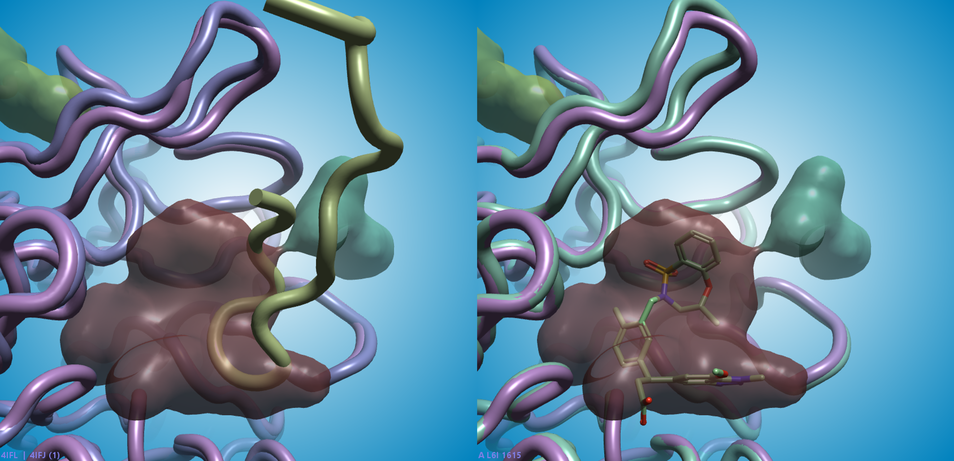
图2. 在PDB 4IFJ(apo KEAP1蛋白:左边和右边的粉色飘带)中发现的棕色口袋打分和成药性得分最高。左图:棕色口袋与PDB 4IFL(NFR2肽:绿色飘带;KEAP1蛋白:淡紫色飘带)中NRF2肽的结合位点相匹配。右图:PDB 5FNU中的纳摩尔小分子抑制剂结合在同一区域(配体:绿色棍状;KEAP1蛋白:绿色飘带)。
针对配体的QM计算
Flare V6的一个新模块是基于PSI4[3]实现的量子力学(QM)计算,这使您能够对配体、构象系综和对接计算的结合构象进行几何优化(最小化)与单点能计算。
当您选择其中一个计算选项时,Flare会打开一个配置面板,您可以在其中选择所需的设置(图3-左)。对于每种可用的QM方法(HF、MP2 和DFT),都有大量的基组可供选择(图3-右)。单点能计算可以在真空、水或多种有机溶剂中进行。计算是多线程的,您可以将Flare连接到Cresset Engine Broker™以访问远程计算资源并显著减少整体计算时间。
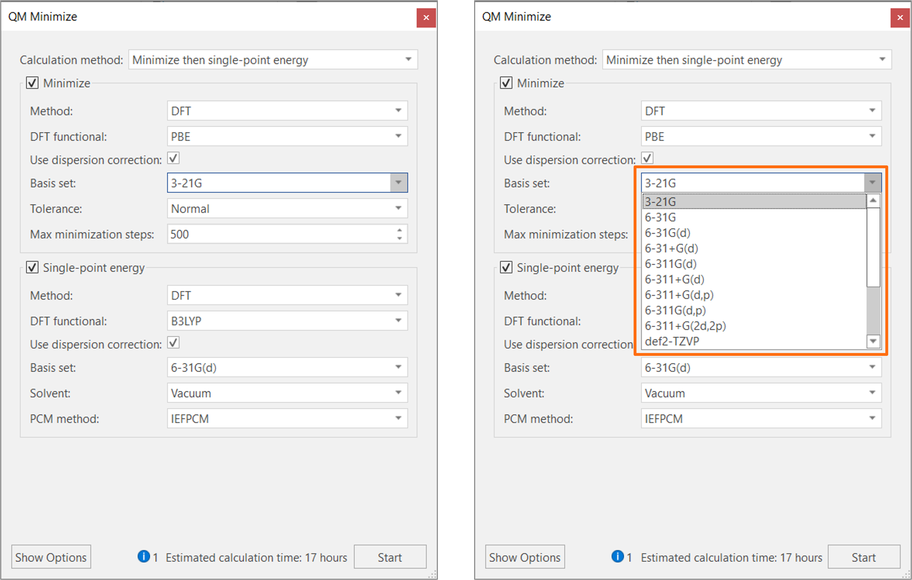
图3. Flare中QM优化与单点能计算面板
默认方法的选项在合理的速度和结果的准确性之间提供了最佳平衡。 计算面板底部的信息、警告和错误消息可帮助您在按下“开始”按钮之前了解您的计算是否设置正确。Flare中的QM计算可以分析配体、配体构象和配体对接结合构象的分子静电势(图 4)。

图4. PDB 4ZLZ中配体的QM分子静电势。青色:负静电势;红色:正静电势。
基于QM两面角扫描分析(图 5)可对配体中感兴趣的旋转键进行计算,并存储于本地以供将来参考。
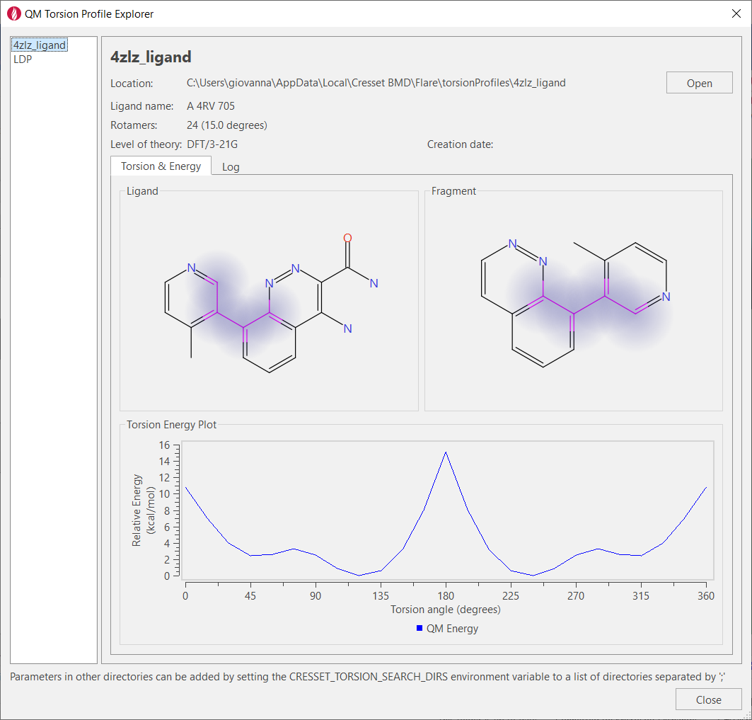
图5. PDB 4ZLZ配体高亮部分两面角基于QM的扫描结果
命令行计算试验让事情计算更简单
使用pyflare在命令行进行计算是非常重要的——例如,如果您正在考虑对数以万计的配体进行分子对接或分子叠合,又假如您希望在不打开Flare图形界面的情况下跑长时间的分子动力学模拟或自由能扰动计算(FEP)。在Flare V6中,我们让命令行实验变得更加简单,在设置中充分利用了 Flare用户友好的GUI,并在执行中充分利用了pyflare和Cresset Engine Broker的强大功能。例如,假设您要运行大型对接计算。您可以像往常一样在Flare GUI中设置您的计算,选择所有需要的选项:例如,设置对接网格并选择要使用的蛋白和蛋白链,以及是否使用快速、正常或精确的计算方法 ( 图 6)。
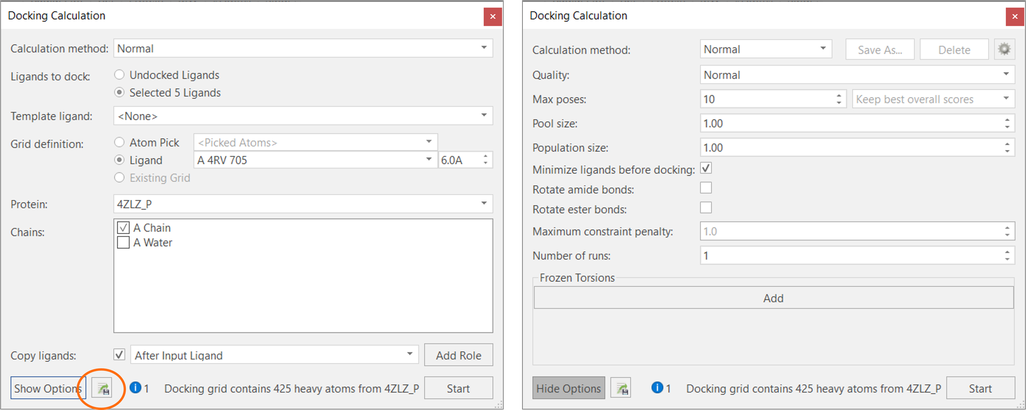
图6. 使用Flare对接计算面板(左)和对接高级选项(右)设置对接实验。按“导出”按钮保存从命令行运行实验所需的所有信息。
然后按下计算面板底部的“导出”按钮,即可将命令行计算实验所需的所有文件和信息保存在您选择的目录中(图 7)。如果您已将Flare配置为使用Cresset Engine Broker访问远程计算资源,脚本将自动使用它。
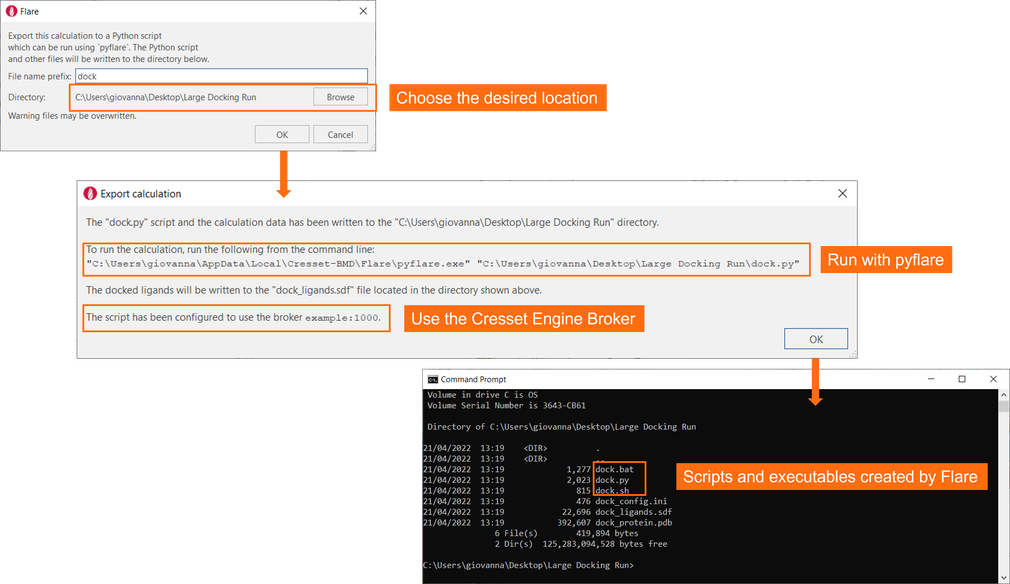
图7. Flare将在所选目录中保存所有必需的文件和信息,并提供有关如何运行实验的明确说明。
您现在可以使用pyflare和Flare创建的’dock.py’脚本开始对接实验。或者,如果您在Windows/Linux上工作,则使用’dock.bat’/’dock.sh’可执行文件。
文献
- Le Guilloux, V.; Schmidtke, P.; Tuffery, P. Fpocket: An Open Source Platform for Ligand Pocket Detection. BMC Bioinformatics 2009, 10 (1), 168. https://doi.org/10.1186/1471-2105-10-168.
- Davies, T. G.; Wixted, W. E.; Coyle, J. E.; Griffiths-Jones, C.; Hearn, K.; McMenamin, R.; Norton, D.; Rich, S. J.; Richardson, C.; Saxty, G.; et al. Monoacidic Inhibitors of the Kelch-like ECH-Associated Protein 1: Nuclear Factor Erythroid 2-Related Factor 2 (KEAP1:NRF2) Protein-Protein Interaction with High Cell Potency Identified by Fragment-Based Discovery. J. Med. Chem. 2016, 59 (8), 3991–4006. https://doi.org/10.1021/acs.jmedchem.6b00228.
- Smith, D. G. A.; Burns, L. A.; Simmonett, A. C.; Parrish, R. M.; Schieber, M. C.; Galvelis, R.; Kraus, P.; Kruse, H.; Di Remigio, R.; Alenaizan, A.; et al. P SI4 1.4: Open-Source Software for High-Throughput Quantum Chemistry. J. Chem. Phys. 2020, 152 (18). https://doi.org/10.1063/5.0006002.



