摘要:本文以DUD-E的EGFR数据集为例,演示了如何用Flare align进行虚拟筛选,并用多个指标评估了虚拟筛选的性能,并与分子对接、形状与药效团等方法进行了比较。在ROC AUC表征的整体性能上,Flare align与分子对接方法GLIDE、DOCK以及Surflex-Dock相当,但优于AutoDock Vina;与药效团方法AutoPH4相当;优于基于形状的方法Surflex-eSim以及WEGA。在BEDROC(alpha=80.5)表征的早期富集能力上,Flare align优于分子对接方法GOLD、GLIDE、Surflex-Dock与FlexX。Flare align与药效团AutoPH4相比,在BEDROC(alpha=321.9、80.5、20.0)表征的早期富集能力上略占优势。在EF ( top 1%、5%与10%)表征的富集能力上、Flare align远远地优于分子对接方法AutoDock Vina。而与药效团方法AutoPH4在EF(top 0.5%与1%)上表现出略占优势的富集能力。
肖高铿/2021-02-16 在Blaze DEMO服务器上实现
肖高铿/2023-03-13 因为BLAZE DEMO从2023年3月开始不再提供网络服务导致数据不能共享下载,所以用Flare align重复实验并通过百度网盘提供原始数据以供验证。
前言
场技术(Field Technology)是Cresset的核心技术1,2。Blaze的前身为FieldScreen,是场技术用于基于配体虚拟筛选的一个实现,适用于大规模虚拟筛选3-5。Flare的ligand alignment(以下简称Flare align或align)是场技术用于分子叠合与虚拟筛选的另一个实现,适用于小规模(几万个分子以内的)的分子叠合与虚拟筛选6。Blaze与Flare align的计算方法完全相同,区别在于:1) BLAZE使用GPU加速,几千万分子几个小时完成虚拟筛选;而Flare align使用CPU计算,速度慢。2) BLAZE的GUI比Flare align的GUI功能更加强大。
本文的主要目的是:以DUD-E的EGFR数据集7为例,演示Flare align/BLAZE的使用方法,并给出在该数据集上的虚拟筛选性能。
方法
基于场的虚拟筛选
基于场的虚拟筛选流程如图1所示,首先要选取一个已知的活性化合物作为参比分子并获得到它合理的3D构象(图1 步骤A),在配体-蛋白复合物结构中的生物活性构象为首选的3D构象;然后在3D构象上加上场点作为虚拟筛选的Query(图1 步骤B);对数据库中每个分子进行构象搜索,得到的低能构象也加上场点(图1 步骤C),将数据库分子构象的场点叠合到query的场点上,并比较场点模式的相似性,得到每个化合物与query的场点相似性值与分子叠合(图1 步骤D)。
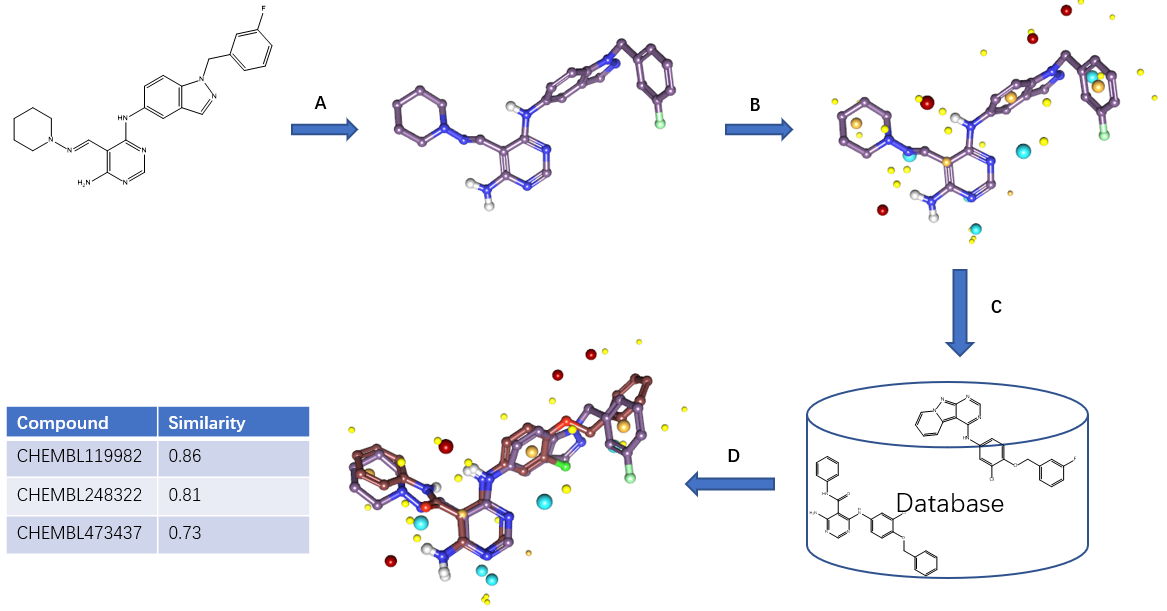
图1. 虚拟筛选流程
参比分子的准备
在本次实验中,参比分子直接采用DUD-E EGFR数据集的ligand.mol2,是从HYZ-EGFR kinase共晶结构PDB 2RGP中提取而得。
数据库分子的准备
直接采用DUD-E EGFR数据集actives与decoys(SDF格式)已经准备好的结构用于虚拟筛选,不进行额外的准备。
Flare align虚拟筛选
Flare align虚拟筛选用Flare自带的脚本align.py在命令行下完成,全部采用默认值:
actives数据集的虚拟筛选:
1 | pyflare align.py egfr/ligand.mol2 ../dude/egfr/actives_final.sdf --batch-size 120 >> egfr/actives.sdf |
decoys数据集的虚拟筛选:
1 | pyflare align.py egfr/ligand.mol2 ../dude/egfr/decoys_final.sdf --batch-size 120 >> egfr/decoys.sdf |
其中ligand.mol2是DUD-E/EGFR数据集从PDB 2PRG中提取出来的配体结构;actives_final.sdf与decoys_final.sdf分别为sdf格式的actives与decoys数据集;batch-size选项让flare align在虚拟筛选时以120个化合物为一批分批处理,以确保内存够用;actives与decoys数据集的虚拟筛选结果分别保存为子目录egfr下的actives.sdf与decoys.sdf。所有这些原始数据可在附件里下载得到。
需要注意的是,默认条件虚拟筛选并非最优,你可以看到有很多警告出现:该分子柔性键多,默认模式的构象搜索可能会导致分子叠合失败。但是,在本次测试中我们忽略该警告。
数据处理与性能评估
鉴于DUDE下载的actives与decoys数据集因为互变异构体与质子化状态的枚举,导致一个化合物会以不同的形式多次出现、并可能被虚拟筛选多次命中,因此对虚拟筛选结果按化合物名称进行去重,同名化合物仅以其中打分最高那个来代表,所有的性能评估指标都在去重的基础上进行计算。
在虚拟筛选过程中,有可能有的化合物处理失败而没有出现在结果文件里,为了保证性能评估的结果与其它文献报道的具有可比性,将缺失的化合物人为地分配一个很低的打分值0.001然后再进行性能评估。
用AUC、logAUC、三种α参数的BEDROC、富集因子等指标来评估虚拟筛选的性能,具体的计算方法参见前文8。
结果
数据预处理
在本次计算中,对叠合之后的结果用python的pandas的聚合函数groupby对化合物分值进行处理:同一个化合物的互变异构体、不同质子化状态仅保留打分最高的那个作为该化合物的打分值。actives与decoys数据集的处理结果如表1所示:actives有1个分子处理失败,decoys有1474个分子处理失败。因此,在之后的性能评估中,这些处理失败的分子给予一个0.01的打分值之后参与统计,以便得到的性能指标与文献报道的指标具有可比性,结果见附件all_score.csv。
表1、DUD-E EGFR数据集化合物统计
| EGFR数据集 | 输入的化合物数 | 输入的异构体数 | 输出的化合物数 |
|---|---|---|---|
| actives | 542 | 832 | 541 |
| decoys | 35020 | 35442 | 33546 |
根据表1,阳性化合物在总体化合物数的1.5%,EFMax = 65.6。记住这两个数值很重要,因为DUD-E数据集为预处理直接可用,很多文章在评估虚拟筛选方法时错误地将预处理过的质子化状态异构体、互变异构体当成不同分子,导致阳性化合物数被错误计算,一般是偏多了几百个,并最终导致性能统计学指标不具可比性。DUD-E数据集的特点是,Decoys子集的异构体数与化合物数接近,而actives的异构体数远远大于化合物数。不少文章将异构体数当成化合物数进行统计处理,这导致EFMax不同,而EFX不具可比性。
绘制ROC曲线与半对数ROC曲线
本次虚拟筛选的ROC AUC=0.85(图2),1000次采样boostrap计算的95%置信区间为[0.8342,0.8737],见表2。虽然ROC AUC常用于定量评估、比较不同的虚拟筛选方法,但是同样的ROC AUC也不能保证同样的性能8。ROC曲线是比ROC AUC更加直观地评估性能:曲线越靠近纵坐标,性能越好。从ROC曲线(图2)可以看到在假阳性很低的时候,曲线就非常靠近纵坐标,这是早期富集能力好的表现。
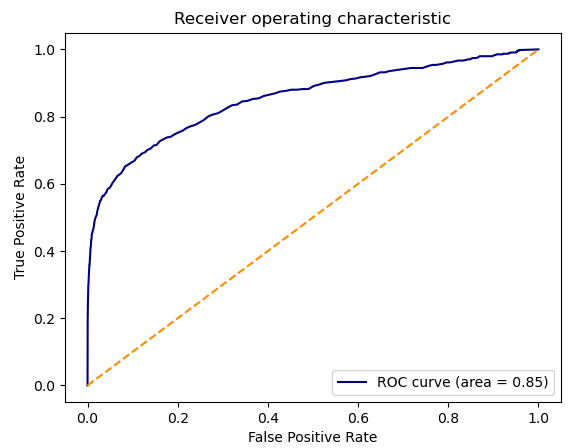
图2. 用共晶配体(PDB 2RGP)作为参比分子进行虚拟筛选的ROC曲线及ROC AUC
对ROC曲线图横坐标的假阳性率进行对数转换具有放大真阳性率表示的早期富集能力的效果9,这时得到半对数ROC曲线,如图3所示。半对数ROC曲线下面积用logAUCλ=0.001,较高的logAUCλ=0.001值意味着能更好地区分活性和非活性化合物,为虚拟筛选方法及其参数识别活性化合物的能力提供了很好的指标。
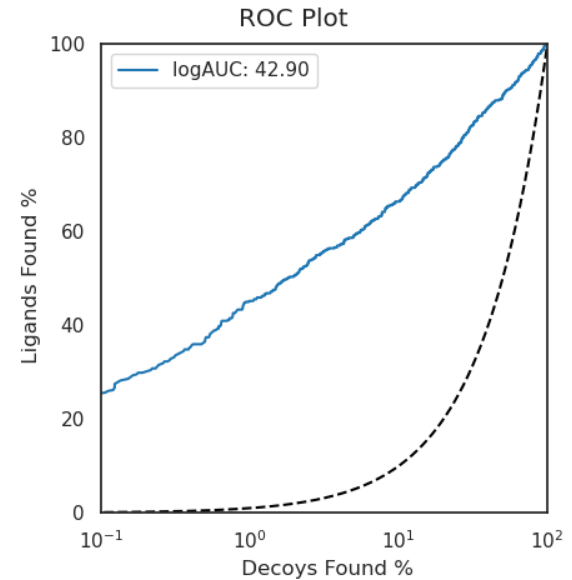
图3. 用共晶配体(PDB 2RGP)作为参比分子进行虚拟筛选的半对数ROC曲线及logAUCλ=0.001
需要注意的是,这里所说的logAUCλ=0.001指的是adjusted-logAUC,也就是图3蓝色实心曲线下面积减去灰色虚线曲线下面积之后的面积。logAUC与adjusted-logAUC一般互相通用,但经常有课题组用原始的logAUC,而不是adjusted-logAUC,需要请读者仔细甄别。在本文中,当λ=0.001时的logAUC与adjusted-logAUC分别为57.4与42.9,如表2所示。
表2、用PDB 2RGP的共晶配体进行虚拟筛选的性能指标
| 指标 | Flare align |
|---|---|
| AUC | 0.854 |
| CI 95%(1000 resample) | 0.8342 – 0.8737 |
| ER1% | 45.6 |
| logAUCλ=0.1% | 57.4 |
| adjusted-logAUCλ=0.1% | 42.9 |
| BEDROC α=321.9 | 0.762 |
| BEDROC α=80.5 | 0.562 |
| BEDROC α=20.0 | 0.583 |
| EF0.5% | 51.6 |
| EF1% | 35.9 |
| EF5% | 11.5 |
| EF10% | 6.5 |
如图4所示,与最近公开的一些虚拟筛选方法相比,就DUD-E EGFR数据集而言,Flare align与流行的分子对接方法GLIDE,DOCK以及Surflex-Dock具有相似的ROC AUC值10;但是明显地优于分子对接方法AutoDock 1.211;与基于配体形状的方法相比,显著优于最近发布的Surflex-eSim12与WEGA13,实际上优于Cleves等人12提到的其它基于配体3D形状的虚拟筛选工具 (ROC AUCMax=0.77,未在图4中给出);与最近Jiang等人14提出的自动药效团方法AutoPH4相当。

图4. Flare align与最近公开的一些虚拟筛选方法在DUD-E EGFR数据集上的ROC AUC比较
总的来说,在DUD-E EGFR数据集上,Flare align的总体虚拟筛选性能(ROC AUC)与当今最先进的分子对接、分子形状以及药效团方法相当。
用BEDROC评估性能
BEDROC是常用的早期富集能力指标,根据Chaput等人15对DUD-E 102个靶标的研究,当α=321.9、80.5与20.0时BEDROC的评估效果分别与打分靠前0.5%、2%以及8%富集因子(EF)的评估效果相当。当α为321.9、80.5、20.0时,Flare align的BEDROC分别为0.762,0.562与0.583,见表2。

图5. Flare align与几个流行的分子对接软件的BEDROCα=80.5比较
根据Chaput等人15的研究,流行的分子对接方法GOLD、GLIDE、Surflex-Dock与FlexX在DUD-E EGFR数据集上的BEDROCα=80.5分别为0.42、0.4、0.24与0.35,低于Flare align的0.562,见图5。这说明Flare align基于场点的早期虚拟筛选性能要优于这些最流行的分子对接虚拟筛选方法。
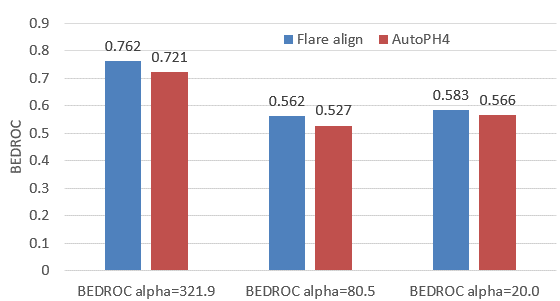
图6. Flare align与药效团方法AutoPH4的BEDROC比较
与Jiang等人14提出的自动药效团方法AutoPH4相比,当药效团从复合物结构识别的时候,Flare align与AutoPH4在α=321.9、80.5与20.0时的BEDROC相当,如图6直方图所示。
用富集因子(EF)评估性能
DUD-E EGFR数据集的EFMax=65.6,因此EF由命中率而定,最大为65.6。如表2所示,Flare align的EF0.5%、EF1%、EF5%与EF10%分别为51.6、35.9、11.6与6.5,极大地领先于Eberhardt等人11等人报道的AutoDock Vina 1.2的EF1%(=3.68)、EF5%(=2.76)与EF10%(=2.17),如图7所示。
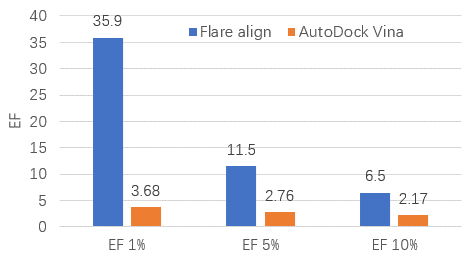
图7. Flare align与AutoDock Vina在DUD-E EGFR数据集上的富集因子比较
与Jiang等人14提出的自动药效团方法AutoPH4相比,如图8所示,Flare align在EF0.5%与EF1%上略占优势,这与前面BEDROC值的趋势是一致的。
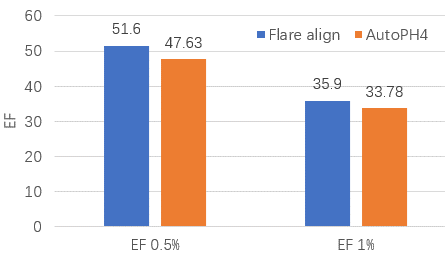
图8. Flare align与AutoPH4在DUD-E EGFR数据集上的富集因子比较
与其它基于3D相似性方法的比较
最近Jiang等人16在DUD-E与LIT-PCBA数据集上对ROCS、Phase Shape、SHAFTS、WEGA、ShaEP、Shape-it、Align-it、LIGSIFT、LS-align等9种基于3D相似性的虚拟筛选方法进行性能评估。其中,在DUD-E EGFR靶标上以共晶配体为参比进行虚拟筛选的总体性能指标ROC AUC与富集因子(EF)汇总如表3所示。
表3. Flare align与其它3D相似性方法在DUD-E/EGFR数据集上的虚拟筛选性能比较*
| 方法 | ROC AUC | EF1% | EF5% | EF10% |
|---|---|---|---|---|
| Flare align | 0.85 | 35.9 | 11.5 | 6.5 |
| ROCS_Comboscore | 0.67 | 14.59 | 5.87 | 3.58 |
| ROCS_Colorscore | 0.70 | 17.54 | 6.28 | 4.01 |
| ROCS_ShapeTanimoto | 0.58 | 3.14 | 2.51 | 1.98 |
| Phase Shape_Mmod | 0.81 | 12.00 | 6.06 | 4.64 |
| Phase Shape_Ele | 0.70 | 11.08 | 5.36 | 3.73 |
| Phase Shape_Pharm | 0.85 | 15.88 | 9.27 | 6.12 |
| Shape-it | 0.46 | 1.11 | 0.74 | 0.74 |
| Align-it | 0.79 | 16.62 | 7.43 | 5.08 |
| ShaEP_best | 0.62 | 4.99 | 2.81 | 2.22 |
| ShaEP_shape | 0.59 | 4.43 | 2.40 | 2.13 |
| ShaEP_ESP | 0.58 | 2.03 | 1.48 | 1.31 |
| SHAFTS | 0.89 | 36.56 | 13.53 | 7.63 |
| WEGA | 0.67 | 6.65 | 3.88 | 3.10 |
| LIGSIFT | 0.78 | 18.65 | 7.61 | 4.99 |
| LS-align | 0.72 | 17.54 | 6.32 | 4.16 |
*除了Flare align之外的数据均来源于Jiang等人16的研究。
以ROC AUC表征的总体虚拟筛选性能上看,SHAFTS最优(ACU = 0.89),Flare align与Phase Shape_Pharm次之(AUC均为0.85)但优于其它方法,如图9所示。
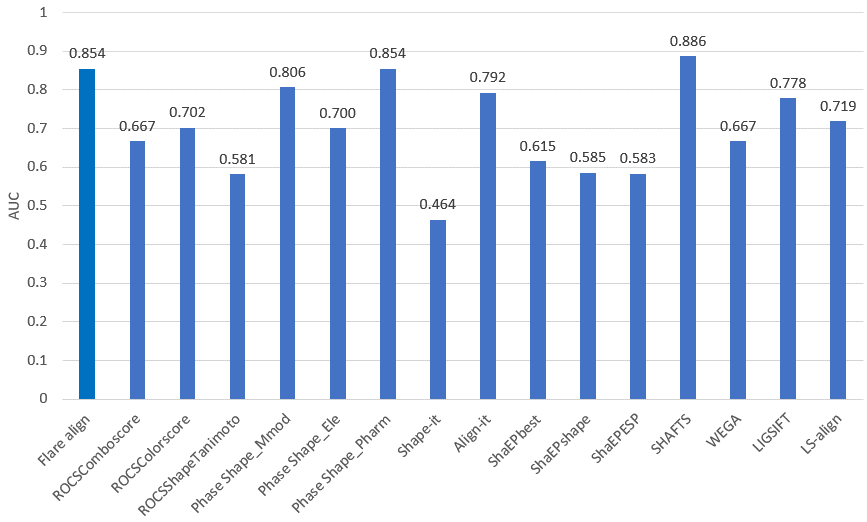
图9. Flare align与其它9种3D相似性方法在DUD-E/EGFR数据集上虚拟筛选总体性能(ROC AUC)比较
在Top 1%,5%与10%富集因子(EF)表征的虚拟筛选性能上,Flare align与SHAFTS相当,以一倍的优势优于其它方法,如图10所示。
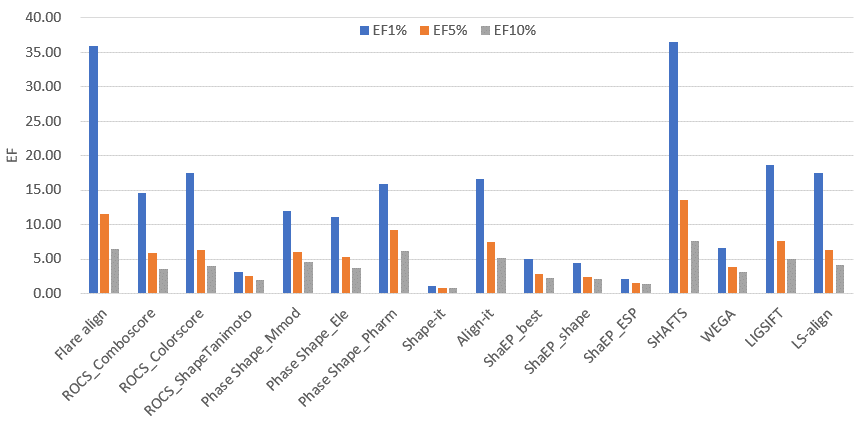
图10. Flare align与其它9种3D相似性方法在DUD-E/EGFR数据集上虚拟筛选的富集因子(EF)比较
这里我们看到AUC表征的总体虚拟筛选性能与EF表征的富集能力可以不一致,Phase Shape_Pharm与SHAFTS、Flare align有相似的ROC AUC值,但其表征早期富集能力的EF1%只有SHAFTS与FLare align的一半,这说明不能仅用AUC来评估虚拟筛选性能。总的来说,Flare align表现出优秀的虚拟筛选总体性能与早期富集能力。
结论
本文以DUD-E的EGFR数据集为例,演示了如何用Flare align进行虚拟筛选,并用多个指标评估了虚拟筛选的性能,与一些分子对接、形状与药效团等方法进行了比较。
在ROC AUC表征的整体性能上,Flare align与分子对接方法GLIDE、DOCK以及Surflex-Dock相当,但优于AutoDock Vina;与药效团方法AutoPH4相当;优于基于形状的方法Surflex-eSim以及WEGA。
在BEDROCα=80.5表征的早期富集能力上,Flare align优于对接方法GOLD、GLIDE、Surflex-Dock与FlexX。在BEDROC(α=321.9、80.5、20.0)表征的早期富集能力上,Flare align比药效团方法AutoPH4性能略优。
在EF1%、EF5%、EF10%表征的富集能力上、Flare align远远地优于分子对接方法AutoDock Vina。在EF0.5%以及EF1%表征的富集能力上,Flare align比药效团方法AutoPH4性能略优。
与其它9种基于3D配体虚拟筛选方法相比,Flare align也表现出优秀的总体虚拟筛选性能与早期富集能力。
附件
- DUD-E EGFR用Flare align叠合的原始数据:https://pan.baidu.com/s/1VL9G_zyQGUNoE09kK2UGYA 提取码:ymfr
文献
- Cheeseright, T.; Mackey, M.; Rose, S.; Vinter, A. Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation. J. Chem. Inf. Model. 2006, 46 (2), 665–676. https://doi.org/10.1021/ci050357s.
- Cresset核心技术. http://www.molcalx.com.cn/cresset-science
- Blaze, https://www.cresset-group.com/software/blaze
- Cheeseright, T. J.; Mackey, M. D.; Melville, J. L.; Vinter, J. G. FieldScreen: Virtual Screening Using Molecular Fields. Application to the DUD Data Set. J. Chem. Inf. Model. 2008, 48 (11), 2108–2117. https://doi.org/10.1021/ci800110p.
- Cheeseright, T.; Mackey, M.; Rose, S.; Vinter, A. Molecular Field Technology Applied to Virtual Screening and Finding the Bioactive Conformation. Expert Opin. Drug Discov. 2007, 2 (1), 131–144. https://doi.org/10.1517/17460441.2.1.131.
- Flare Ligand Alignment. http://www.molcalx.com.cn/cresset/flare/ligand-alignment
- DUD-E EGFR dataset. https://dude.docking.org/targets/egfr
- 肖高铿. 如何进行虚拟筛选的方法学验证.墨灵格的博客. 2016-09-22. http://blog.molcalx.com.cn/2016/09/22/virtual-screening-methodology-validation.html
- Mysinger, M. M.; Shoichet, B. K. Rapid Context-Dependent Ligand Desolvation in Molecular Docking. J. Chem. Inf. Model. 2010, 50 (9), 1561–1573. https://doi.org/10.1021/ci100214a.
- Cleves, A. E.; Jain, A. N. Structure- and Ligand-Based Virtual Screening on DUD-E + : Performance Dependence on Approximations to the Binding Pocket. J. Chem. Inf. Model. 2020, 60 (9), 4296–4310. https://doi.org/10.1021/acs.jcim.0c00115.
- Eberhardt, J.; Santos-Martins, D.; Tillack, A. F.; Forli, S. AutoDock Vina 1.2.0: New Docking Methods, Expanded Force Field, and Python Bindings. J. Chem. Inf. Model. 2021, acs.jcim.1c00203. https://doi.org/10.1021/acs.jcim.1c00203.
- Cleves, A. E.; Johnson, S. R.; Jain, A. N. Electrostatic-Field and Surface-Shape Similarity for Virtual Screening and Pose Prediction. J. Comput. Aided. Mol. Des. 2019, 33 (10), 865–886. https://doi.org/10.1007/s10822-019-00236-6.
- Puertas-Martín, S.; Redondo, J. L.; Ortigosa, P. M.; Pérez-Sánchez, H. OptiPharm: An Evolutionary Algorithm to Compare Shape Similarity. Sci. Rep. 2019, 9 (1), 1–24. https://doi.org/10.1038/s41598-018-37908-6.
- Jiang, S.; Feher, M.; Williams, C.; Cole, B.; Shaw, D. E. AutoPH4: An Automated Method for Generating Pharmacophore Models from Protein Binding Pockets. J. Chem. Inf. Model. 2020, 60 (9), 4326–4338. https://doi.org/10.1021/acs.jcim.0c00121.
- Chaput, L.; Martinez-Sanz, J.; Saettel, N.; Mouawad, L. Benchmark of Four Popular Virtual Screening Programs: Construction of the Active/Decoy Dataset Remains a Major Determinant of Measured Performance. J. Cheminform. 2016, 8 (1), 56. https://doi.org/10.1186/s13321-016-0167-x.
- Jiang, Z.; Xu, J.; Yan, A.; Wang, L. A Comprehensive Comparative Assessment of 3D Molecular Similarity Tools in Ligand-Based Virtual Screening. Brief. Bioinform. 2021, 22 (6), 1–17. https://doi.org/10.1093/bib/bbab231.



