摘要:本文探讨了基于片段的药物发现(FBDD)面临的挑战,并以PNMT抑制剂为例,介绍了Cresset discovery的建模专家如何应用他们的深度专业知识,结合使用生物等排体替换和建模分析,对这些小的共晶结构片段进行生长操作,评估其对蛋白的静电互补性,并在蛋白质结合位点里分析蛋白-配体的相互作用。
作者:Martin Slater/24 April 2023
编译:肖高铿
前言
基于片段的药物发现(FBDD)是人们公认的推进创新药物设计的有效方法,并已用作开发全新、多样的苗头化合物的高价值技术。FBDD已为几种已批准的、进入临床研究阶段药物的开发做出了贡献,包括Vemurafenib、Venetoclax、Pexidartinib和Onalespib。
将诸如等温滴定量热法(ITC)、NMR、SPR、MS和X-衍射晶体学之类实验方法鉴定出来小的、弱亲和力的结合片段通过合并、连接和生长等方法转化为合适的配体是FBDD面临的一个挑战。这是药物化学经验和计算专业知识发挥重要作用的地方,也是获得专业软件解决方案发挥作用的地方。Cresset Discovery的建模专家运用他们深厚的计算知识和经验,探索和优化生物等排体置换,使用Spark1——一个经过验证的片段连接、生长和合并的最佳独立软件工具之一2——来对起始的先导化合物进行优先性排序。
识别蛋白靶标的片段结合位点
当通过核磁共振和X-衍射方法鉴定出片段时,蛋白靶标结合口袋是已知的。然而,如果通过SPR和ITC鉴定片段,则可能需要确定片段结合位点。Cresset Discovery的顾问应用口袋检测来识别和表征潜在的成药性结合位点3。
用FBDD设计PNMT抑制剂的案例
苯基乙醇胺N-甲基转移酶(PNMT)通过催化去甲肾上腺素的甲基化,参与中枢神经系统(CNS)肾上腺素(肾上腺素)的合成。靶向PNMT的治疗方法有望治疗多种疾病,包括血压控制、帕金森病、阿尔茨海默病和垂体激素分泌异常。
这是一个具有挑战性的药物靶标,因为已经解释的具有底物/抑制剂的共晶结构显示出封闭的活性位点(PDB 1HNN与3HCD)(图1A),并且没有apo晶体结构可用。此外,s-腺苷-l-同型半胱氨酸(AdoHcy)通常结合到晶体结构的辅酶(cofactor)结合位点,在没有AdoHcy的情况下去,得不到该酶结晶的结构。快速晶体结构浸泡法鉴定的片段与PNMT的X-衍射结构(PDB 3KPJ、3KPU、3KPV、3KPW、3KPY、3KQM、3KQS、3KQT、3KQV、3KQW、3KQO、3KQP、3KQQ、3KQY、3KR0、3KR1和3KR2,如图2A算例所示)可用来说明基于FBDD方法的基础4,Cresset Discovery的CADD科学家团队经常在项目中采用这种方法。
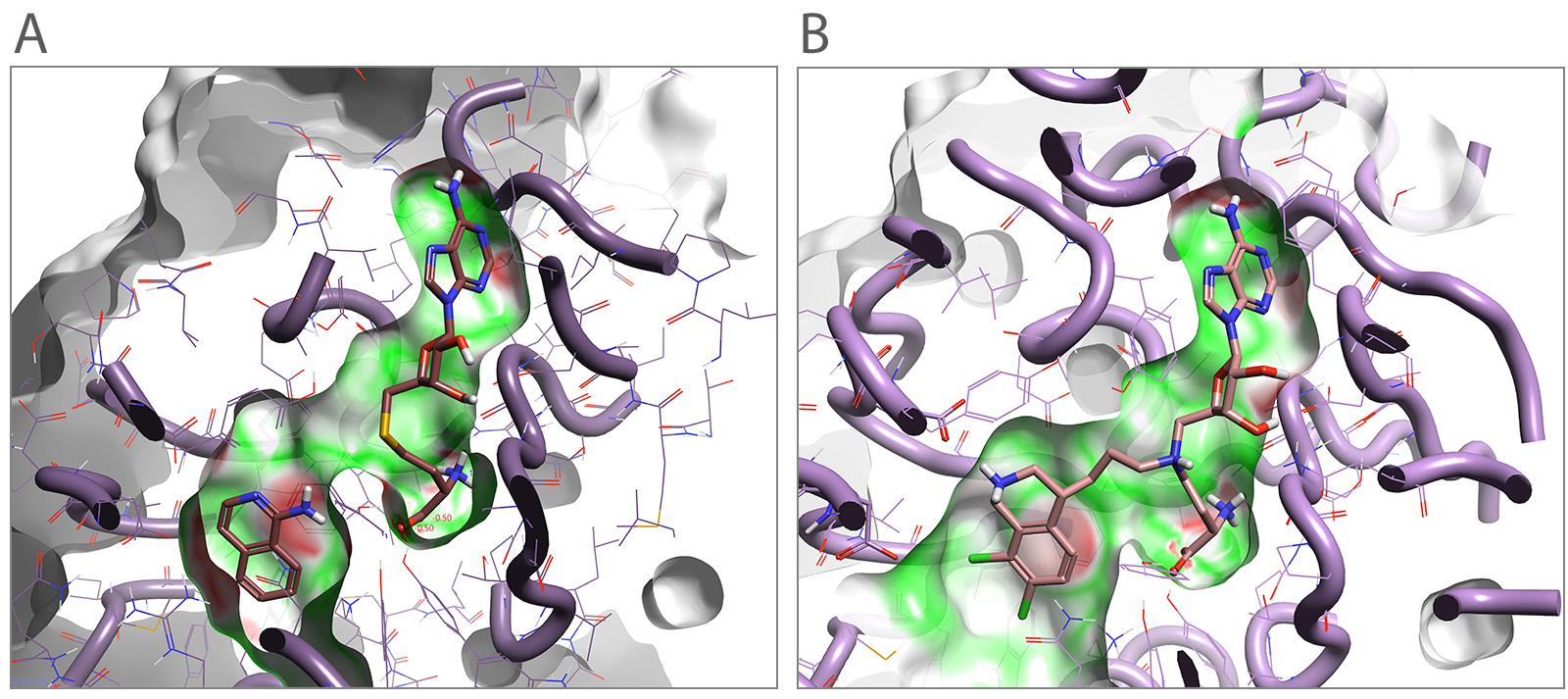
图1. A)人类PNMT与一个片段以及s-腺苷-l-同型半胱氨酸(AdoHcy)共结晶结构。B)说明如何在结合口袋里将片段连接在一起。蛋白结合口袋用静电互补性打分着色,绿色表示互补性好,红色表示互补性差。
在文献研究中,人们使用基于片段的筛选库来鉴定和然后获得片段的共晶结构。PNMT与片段共结晶需要用到s-腺苷-l-同型半胱氨酸(AdoHcy,图1A),其与辅因子位点结合。这些共晶的片段小库置换了去甲肾上腺素结合袋中发现的磷酸盐。
Cresset Discovery的科学家应用从头设计的专业知识,结合使用生物等排体替换1和建模3分析,对这些小的共晶结构片段进行生长操作,评估其对蛋白的静电互补性,并在蛋白质结合位点里分析蛋白-配体的相互作用。此外,片段作为配体设计的起点,通过设计的连接臂与AHdoHcy进行连接,以填充完整的配体辅因子结合口袋。
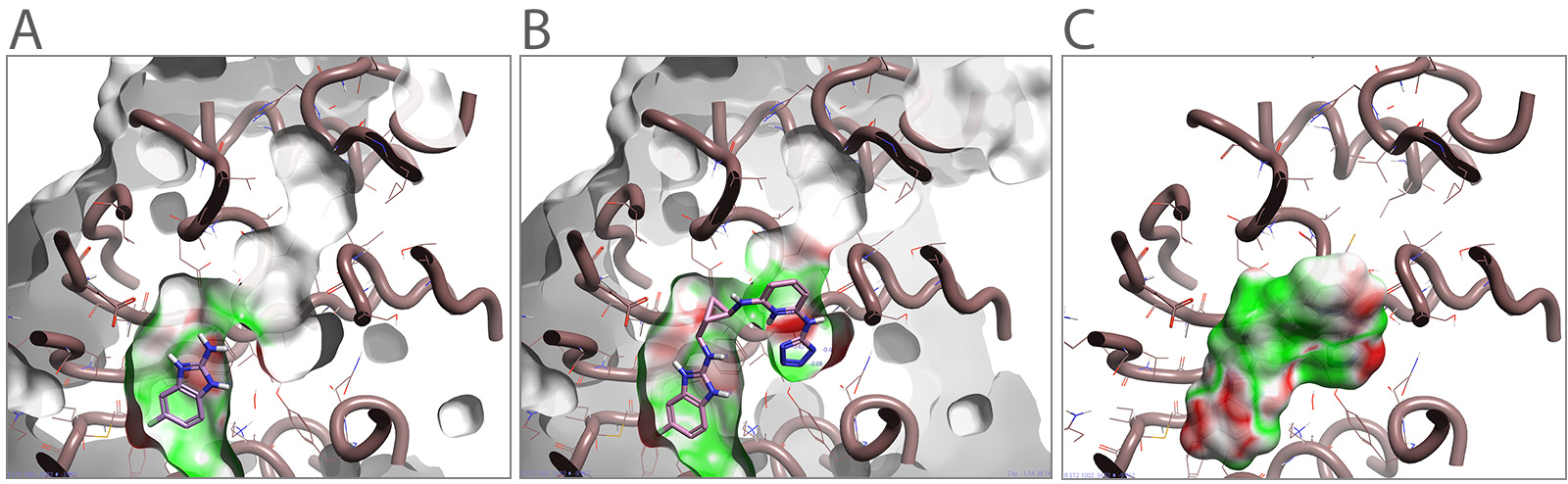
图2. A)PNMT与片段共晶结构的一个例子;B)使用s-腺苷-l-同型半胱氨酸指导片段在PNMT结合袋中生长;C)根据与蛋白结构的静电互补性对配体表面着色。
从起始配体开始,并针对一个或多个参比分子(在本算例中为AdoHcy)进行建模,Cresset Discovery的科学家绘制了蛋白质内同一口袋的不同区域。预先将起始分子和参比分子都在3D中相对于彼此叠合好。配体连接实验还可以从共晶的片段和AdoHcy作为两个起始分子开始,然后用不同连接臂连接。这会产生非常有价值的起始先导化合物,以加速用全新的FBDD配体来产生IP。
除了配体生长与连接之外,Cresset Discovery可以提供专业的解决方案来进行配体大环化、水分子替换,甚至生成片段数据集。
与Cresset Discovery团队合作加速您的FBDD项目
作为您的计算化学CRO,我们拥有所需的技能和资源,通过片段生长和连接、促进从头配体设计和增加配体修饰的多样性来支持您的FBDD发现项目。想了解我们如何与您的团队合作,请联系我们开启一个保密的在线讨论。
相关文章
- 了解Cresset Discovery都提供什么样的服务:http://blog.molcalx.com.cn/2020/06/27/cresset-discovery-service.html
- 了解Cresset Discovery服务流程:http://blog.molcalx.com.cn/2022/10/26/engaging.html
参考文献
- Spark™, Cresset®, Litlington, Cambridgeshire, UK; https://www.cresset-group.com/spark/; Cheeseright T., Mackey M., Rose S., Vinter, A.; Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation. J. Chem. Inf. Model. 2006, 46 (2), 665-676
- Matthew P. Baumgartner and David A. Evans, Side chain virtual screening of matched molecular pairs: a PDB-wide and ChEMBL-wide analysis, Journal of Computer-Aided Molecular Design 2020, 34, 953–963. https://doi.org/10.1007/s10822-020-00313-1
- Flare™, Cresset®, Litlington, Cambridgeshire, UK; https://www.cresset-group.com/flare/; Cheeseright T., Mackey M., Rose S., Vinter, A.; Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation J. Chem. Inf. Model. 2006, 46 (2), 665-676; Bauer M. R., Mackey M. D.; Electrostatic Complementarity as a Fast and Effective Tool to Optimize Binding and Selectivity of Protein–Ligand Complexes J. Med. Chem. 2019, 62, 6, 3036-3050; Maximilian Kuhn, Stuart Firth-Clark, Paolo Tosco, Antonia S. J. S. Mey, Mark Mackey and Julien Michel Assessment of Binding Affinity via Alchemical Free-Energy Calculations J. Chem. Inf. Model. 2020, 60, 6, 3120–3130
- Nyssa Drinkwater, Hoan Vu, Kimberly M. Lovell, Kevin R. Criscione, Brett M. Collins, Thomas E. Prisinzano, Sally-Ann Poulsen, Michael J. Mcleish, Gary L. Grunewald and Jennifer L. Martin, Biochem. J. 2010, 431, 51-61. https://doi.org/10.1042/BJ20100651



