摘要:本文是前期在线视频研讨会的文字讲解,以BTK抑制剂为例,回顾了如何在CADD软件解决方案Flare中用XED力场和QSAR方法可靠地构建QSAR模型,演示了如何用Hit Expander从一个起始化合物开始快速地自动结构修饰生成系列衍生物,然后用建好的预测性QSAR模型预测这些新化合物的活性,最后获得有潜力的新设计。
作者:Matt Giblin/30 October 2023
编译:肖高铿
前言
Hit Expander是分子建模平台Flare™最近引入的一个组件,它使化学家能够通过操纵具有保守变化的潜在分子来快速探索化学空间。通过添加小官能团或进行芳香单原子替换(例如,芳香碳原子转化为芳香氮原子)可以快速生成类似物库,随后可以使用Flare的其他组件进行分析。
今年早些时候,我们的应用科学家Boyli Ghosh主持了一场独家在线研讨会,期间她介绍了一个使用Hit Expander设计新类似物库的案例研究,随后使用预测性QSAR模型预测每个类似物与目标蛋白质的相对结合亲和力。该案例研究为药物发现项目中促进发现和优化先导化合物提供了一个示例工作流程。
对于无法参加网络研讨会或有兴趣了解的人,本文总结了主要议题,并重点介绍了所演示的Flare功能。我们用网络研讨会中每个小节的时间注释了标题,可通过此链接索取会议录像。
用计算方法辅助新药候选化合物的发现[04:51]
人们越来越多地在药物设计中使用计算方法以帮助发现新的候选药物。为了高效地将计算合成与基于配体的药物设计相结合,Cresset在Flare中开发并引入了Hit Expander模块。
在本次网络研讨会上探讨的案例研究中,我们聚焦于BTK(Bruton’s tyrosine kinase)抑制剂的设计,此类抑制剂通过B细胞抗原受体(BCR)信号传导参与调节B细胞发育、活化和存活。抑制BTK有可能为淋巴瘤、炎性疾病和其他自身免疫性疾病提供治疗方法。
从一个苗头化合物开始化学探索[06:23]
Hit Expander(图1)的设计旨在实现对单个苗头化合物的快速化学探索,其工作原理是在所选化合物的所有可用位置添加可选的取代基。在适当的情况下,还可以进行芳香族单原子取代。
通过Hit Expander生成的配体变体可在Flare里进一步实验:通过自由能微扰(FEP)、静电互补性(EC)和预测性QSAR模型等方法对最有前景的分子进行优先级排序。
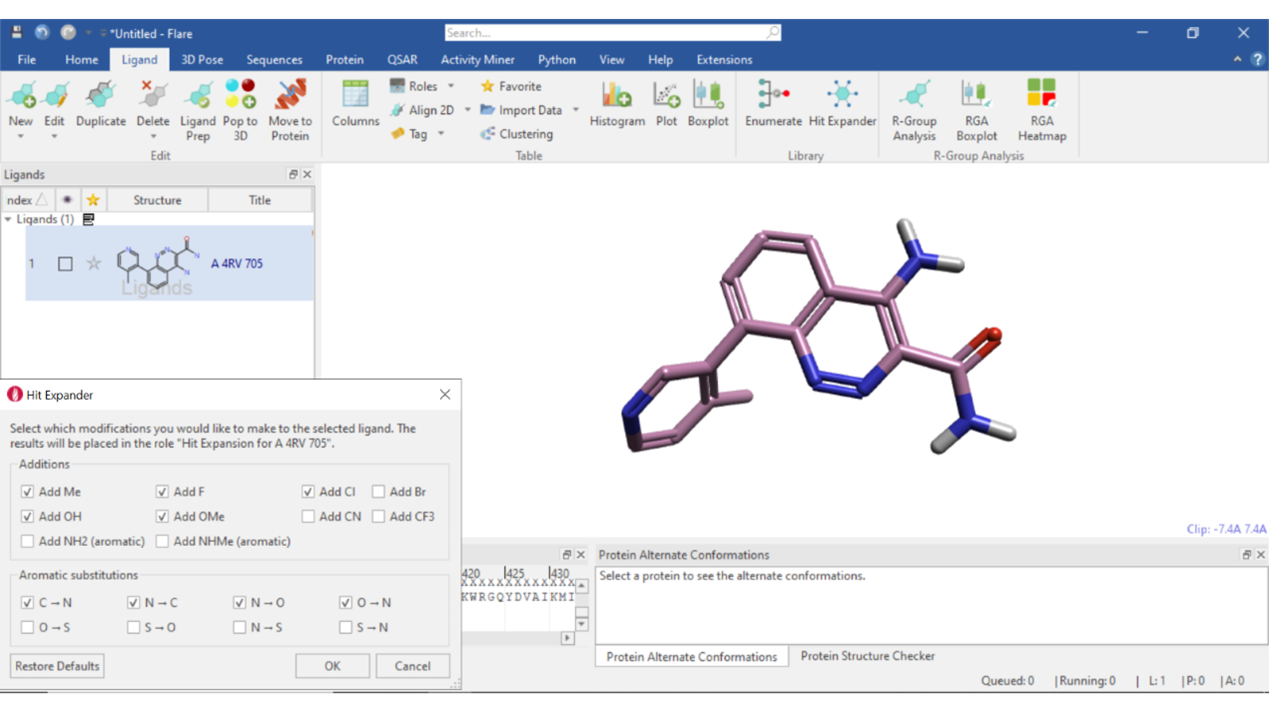
图1. 在Flare里创建Hit Expander实验
预测新化合物的BTK抑制活性[07:05]
本次网络研讨会的算例来自美国专利2015/0038510公开的90个BTK抑制剂。图2总结了在Flare中处理这些化合物的工作流程。
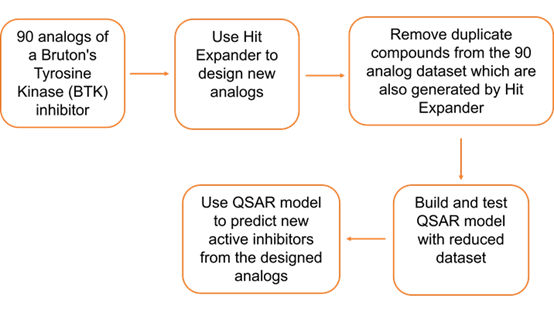
图2. 处理90个BTK抑制剂的Flare Hit Expander/QSAR工作流
Cresset独特的XED力场与其他传统力场不同之处在于不仅在原子中心分配电荷,而且在原子中心之外也分配有单极“XED”点。这种复杂的内部静电模型意味着力场可以很好地模拟极化效应、取代效应、原子电荷各向异性和芳香-芳香相互作用。
在本次网络研讨会中,Boyli介绍了XED力场的背景概述,以及它如何通过场点的生成和可视化来支持对分子设计的更好理解。
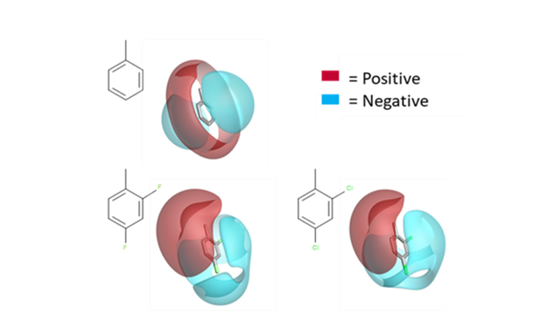
图3. 用Cresset的XED力场来呈现正、负分子场
3D叠合——QSAR实验的基础[14:24]
为了在Flare中生成QSAR模型,需要采取一系列关键步骤,首先从已知的BTK晶体结构中定义参比配体的生物活性构象。在这种情况下,这些信息可以从现有的实验数据中获得,否则可以使用Flare中的FieldTemplater™进行药效团建模,为您的配体系列生成生物活性构象假设。
然后将数据集叠合到该参比配体上,通过目视检查可以识别叠合不佳的分子。然后进行人工干预,以确保训练集中的所有分子都得到很好的叠合,并准备好进行QSAR实验。
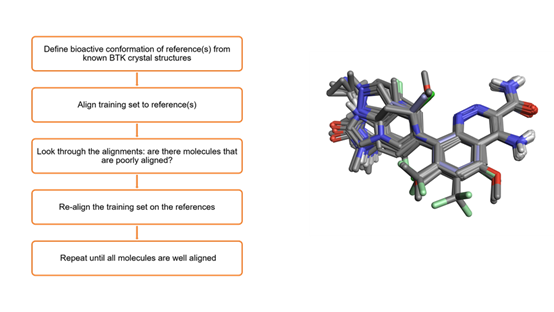
图4. BTK配体数据集的3D叠合工作流
Flare演示:配体叠合[17:19]
首先,使用PDB ID将靶标蛋白导入Flare GUI,并使用适当的质子化状态。然后启动Flare的蛋白准备过程来优化蛋白以进行后续实验。还将参比配体导入其晶体结构中,并在靶标蛋白的活性位点内可视化。
将90个BTK抑制剂数据集导入并叠合到初始参比配体上,然后将数据集分割为训练集与测试集(30%)。
用Hit Expander生成新的配体设计[24:43]
从晶体结构(包含在测试集中)中提取出的共晶配体作为Hit Expander实验的初始起点。在本实验中,选择了4个的官能团(Me、F、Cl和OMe)用于结构修饰。还选择了另外3个配体,其中一个含有未取代的吲唑,另外两个根据不同的活性水平而选择以扩大所生成苗头化合物的多样性。
基于上述这些输入的设计,Hit Expansion实验共产生了72个新的苗头化合物。在初始90个分子组合中,将重复出现的分子删除以避免QSAR建模出现任何偏差。
用QSAR模型预测新生成分子的活性[31:30]
将训练集和测试集中的剩余分子用于QSAR模型构建,模型类型选择回归模型(Regression Models)。完成所需的设置后,计算过程开始,首先为分子数据集添加场。
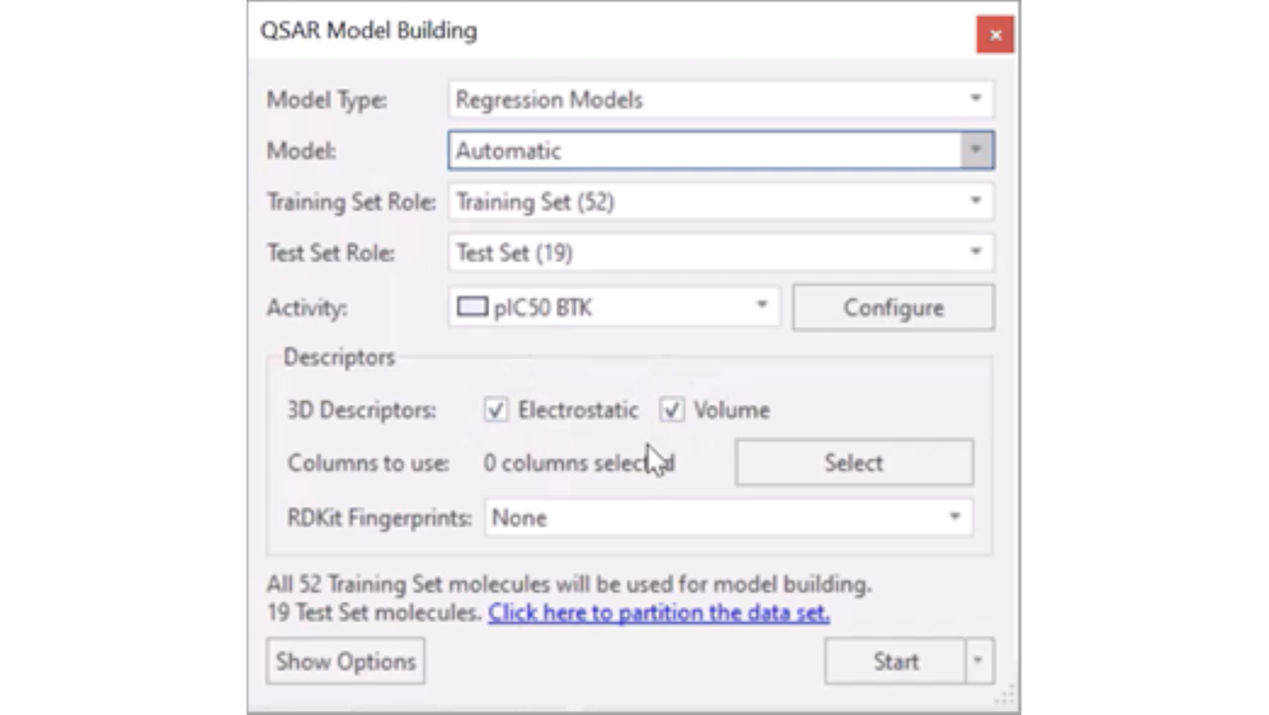
图5. 在Flare里设置QSARR模型实验
实验结果计算的训练集R2值为0.98,训练集交叉验证R2值为0.76,测试集R2值为0.66。这些结果表明,QSAR模型具有良好的预测活性能力,因此可以用于预测Hit Expander生成化合物的活性。
选中所有Hit Expander生成的化合物并可视化,可以看到常见官能团以及关键的正、负静电场紧密聚集。
在90个配体的初始数据集和Hit Expander生成的分子集合中存在19个“重复”化合物,将这些选中并设为收藏(favorites)。这使得可以绘制它们预测的活性值相对于已知活性值的对比图。
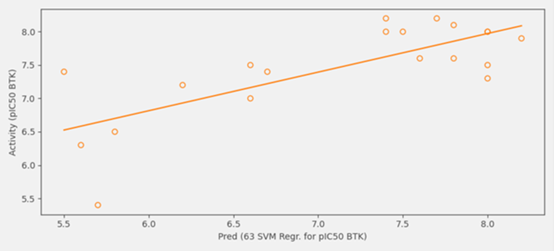
图6. 19个“重复”化合物的pIC50预测值与实验值对比图。
R2值为0.562,表明已知活性值和预测活性值之间具有良好的相关性。利用建好的预测性QSAR模型,基于Hit expander从枚举数据集中预测的pIC50值,鉴定出几个新的潜在活性类似物。
结论[37:39]
本案例研究表明,Flare的XED力场和QSAR建模可以可靠地用于构建BTK抑制剂数据集的预测性模型。Hit Expander帮助产生了新的想法,这些想法被验证过的QSAR模型预测为纳摩尔级活性。
点击请求访问网络研讨会记录,可以查看完整的Flare工作流程演示。此外,您还可以访问我们最近的所有网络研讨会,涵盖了各种计算化学技术。
加速您的小分子发现项目
想要在您自己的项目上尝试此工作流,您可申请Flare试用版并了解如何将Hit Expander/QSAR方法组合应用于其他基于配体的药物设计项目,在这些项目中,没有蛋白结构信息可用。



