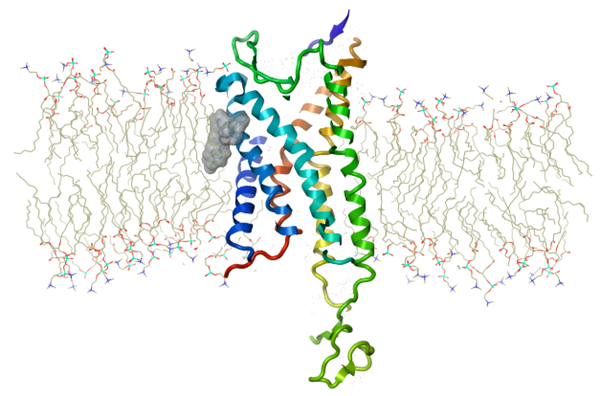
摘要:G蛋白偶联受体(GPCR)是一类非常大、非常重要的药物靶点。高精度预测此类靶标与配体的结合亲和力非常困难,尤其暴露于脂质与膜界面上GPCR配体。在本次报告中,我们以一组暴露于脂质结合位点的P2Y1配体为例,来展示如何用Flare FEP来精确计算这类GPCR配体的结合自由能。
日期
- 日期:2024年1月18日
- 时间:北京时间晚上10点
- 时长: 45分钟
- 形式:在线会议,注册后发送会议链接
- 费用:免费
注册
参加在线会议,请点击:注册连接
摘要
G蛋白偶联受体(GPCR)是一类非常大、非常重要的药物靶点。想要足够高的精度模拟这类靶标与配体的相互作用是非常困难的,因为此类体包含了蛋白质、配体、膜脂和溶剂等非常多的必要原子。这些具有挑战性的体系需要更快、更精确的计算解决方案。在本次报告中,我们将讨论一种可在Cresset软件平台Flare™中使用的计算化学工作流,可用来精确计算一组暴露于脂质结合位点的P2Y1配体结合自由能。
这个工作流涉及的方法有:
- Dynamics:分子动力学模拟(MD)
- 3D-RISM:水的分析
- Conf Hunt & Align:配体叠合用于FEP计算
- Flare FEP:相对结合自由能计算
我们的结果表明,在进行相对结合自由能(RBFE)计算之前使用MD、水分析和仔细的配体叠合可以可靠地预测蛋白质-配体结合亲和力,即使在配体结合于脂质-蛋白质界面的情况下也是如此。直到最近,如此大的系统(93,255个原子)的计算成本仍然过高。通过确保稳定、适当的水合结合位点作为RBFE计算输入,可以高效地设置RBFE作业,以产生准确的结果。通过自适应λ调度和自动中间体生成,使得RBFE计算本身的效率得到提高。结果表明,30个配体的预测结合自由能与测量的实验亲和力非常接近,平均无符号误差为1.17kcal/mol。通过这些方法的使用,这个困难的计算问题现在可以相对快速地得到解决,并且原则上该工作流可以扩展到药物发现中的其他膜蛋白靶标。
主讲人:Jennifer Brookes博士

Jennifer Brookes博士在理论计算化学和生物物理学方面有着15年多的科研经验和背景。她的科学贡献包括探索嗅觉中量子效应的可能性,量化光合作用中有效的能量转移过程,以及抗体-抗原对中的蛋白-蛋白相互作用。珍妮弗的研究使她从伦敦国王学院转到伦敦大学学院、麻省理工学院、哈佛大学,最近又回到伦敦大学学院伦敦纳米技术中心,担任1100万英镑的EPSRC IRC传染病预警传感系统首席计算和理论生物物理学家。作为Cresset的FEP应用科学家,Jennifer Brookes的职责包括支持客户使用自由能微扰计算来可靠准确地预测配体结合自由能,为他们的药物发现项目提供信息与理论指导。



