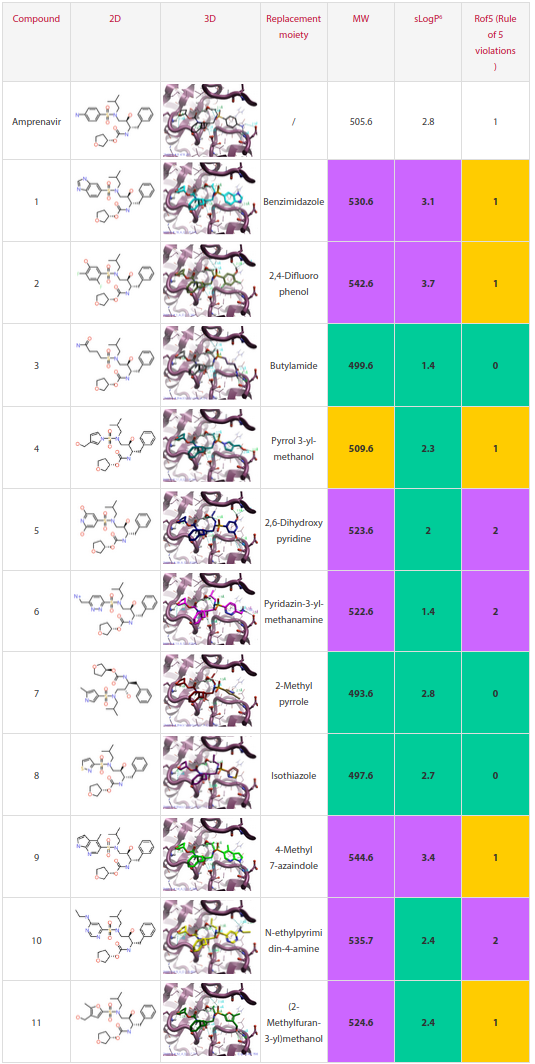
摘要:本文探讨了类药化合物中苯胺片断替换的必要性以及如何用其他化学基团进行替换的研究工作流程,展示了Cresset Discovery团队如何运用他们的深厚知识和计算化学技术,利用诸如Cresset生物等排替代工具Spark™来优化类小分子的有效性和安全性。
原文:Giorgia Zaetta/2024-01-10 Aniline replacement in drug-like compounds
编译:肖高铿
前言
苯胺类化合物是含有氨基的芳香族化合物,由于其结构多样性和潜在的药理学益处,已被发现可用于许多候选药物。然而,苯胺片段的加入并非没有局限性与挑战。
苯胺类化合物因其与生物靶标相互作用并调节生理过程的能力而被用于药物发现。想要理解为什么可能有必要对候选药物中苯胺片段进行替换,需要探索其潜在缺点背后的具体原因。
为什么要替换苯胺
有几个令人信服的原因使得我们考虑替换苗头化合物、先导化合物或候选药物中的苯胺。其中一个主要顾虑是代谢不稳定。苯胺通常易受体内各种代谢酶的影响,这可能导致药物快速排泄,降低其有效性。此外,苯胺与毒性问题和潜在的不良副作用有关。
虽然苯胺可见于几种上市药物的化学结构中,但值得注意的是,苯胺基的存在并不一定意味着毒性或不良反应。苯胺只是这些复杂分子中一部分,这些药物的整体安全性和有效性取决于它们的完整化学结构和它们与身体的相互作用。在某些情况下,可以探索化学修饰来改善这些药物的特性或减少与苯胺部分相关的任何潜在问题。此外,修饰苯胺可以增加受体选择性,从而减少脱靶效应及其相关毒性。
在本文中,我们描述了探索苯胺替代品以及用其他化学基团替代的研究工作流程。展示了Cresset Discovery团队如何运用他们的深厚知识和计算化学技术,如Cresset的生物等排替代工具Spark™1来优化类小分子的有效性和安全性。
如何替换苯胺
为了找到最适合用作本研究起点的药物,对ChEMBL数据库进行了查询,流程如图1所示。通过搜索,发现187种FDA批准的药物在其化学结构中至少含有一个苯胺。对这些药物进行了进一步分析,确定了19类离散的化学系列(通过聚类分析)的代表性化合物。其中,溴芬酸(Bronfenac)因母核环中存在苯胺而被选中,氨雷那韦(Amprenavir)因为具有一个苯胺片断的末端取代基而被选中。此外,虽然溴芬酸是一种小分子,但氨雷那韦是一个分子量较大、表现出溶解度稍差的PK特征。溴芬酸没有公开的与COX-2的复合物晶体结构,因此作为以配体为中心实验的参比分子;而氨雷那韦与其药理学靶标HIV蛋白酶的共晶结构已知,因此作为基于结构分析的参比分子。
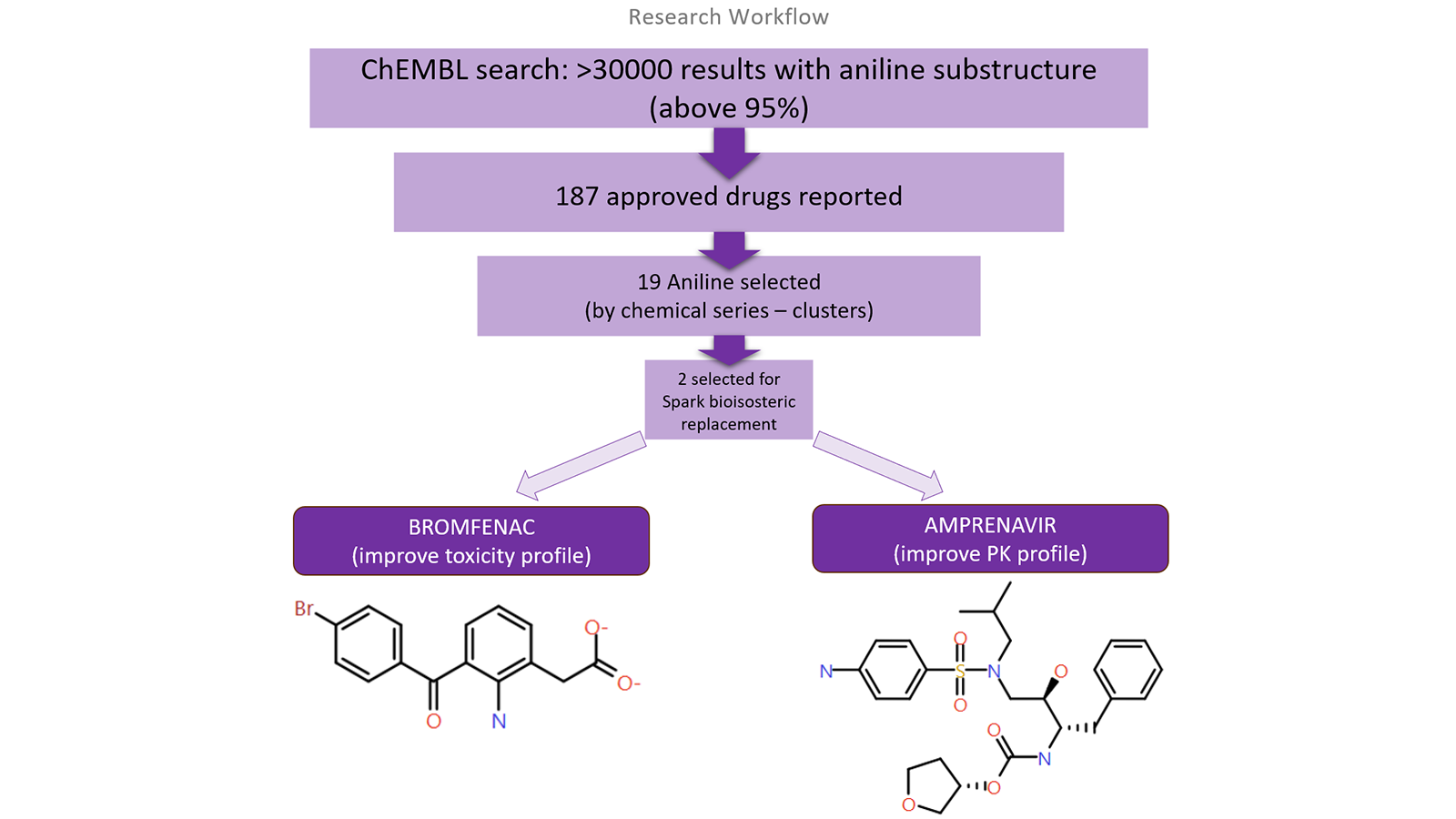
图1. 探索含有苯胺片段化合物的研究流程
使用Spark来识别苯胺的生物等排体。搜索的片段数据库为ChEMBL2和Enamine(商品非常常见和常见)片段数据库,共包含200,000多个片断。Spark用场与形状进行搜索,结果识别出了一系列生物等排体,比如苯并咪唑、取代苯酚、酰胺、吡咯、吡啶片段等。逐步的工作流程解释如下图2所示。
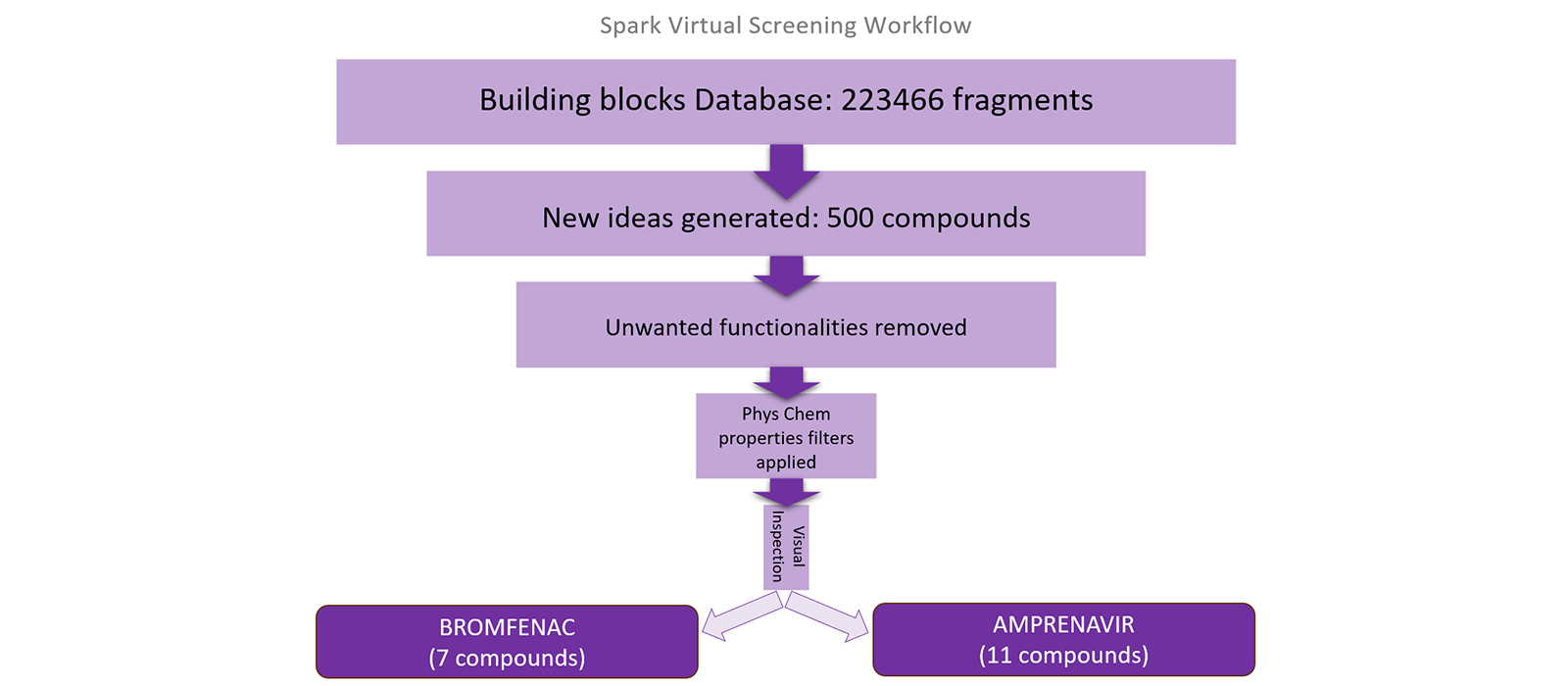
图2. 用于苯胺基团替换的Spark虚拟筛选工作流
微调药理学性质
用不同的化学基团替换苯胺类化合物为优化药物特性提供了一种强有力的策略。这种方法使研究人员能够微调药物的各种药理学特性。例如,通过选择特定的化学片断来替换苯胺可以提高药物的生物利用度、溶解度和受体选择性。
AMPRENAVIR: 替换苯胺以改善PK特性
Amprenavir是一种蛋白酶抑制剂,用于治疗HIV感染。它于1999年获得FDA批准,每天服用两次。在PDB数据库4中已经有几个Amprenavir与靶标的共晶结构。在7N6X蛋白结构中,配体在HIV蛋白酶活性位点内发生了多次相互作用而与蛋白质结合,如图3所示。值得注意的是,在这种情况下,苯胺与Asp30相互作用,因此,任何旨在改善该化合物PK特征的替换都需要能够维持或改善与Asp30或周围残基的相互作用。
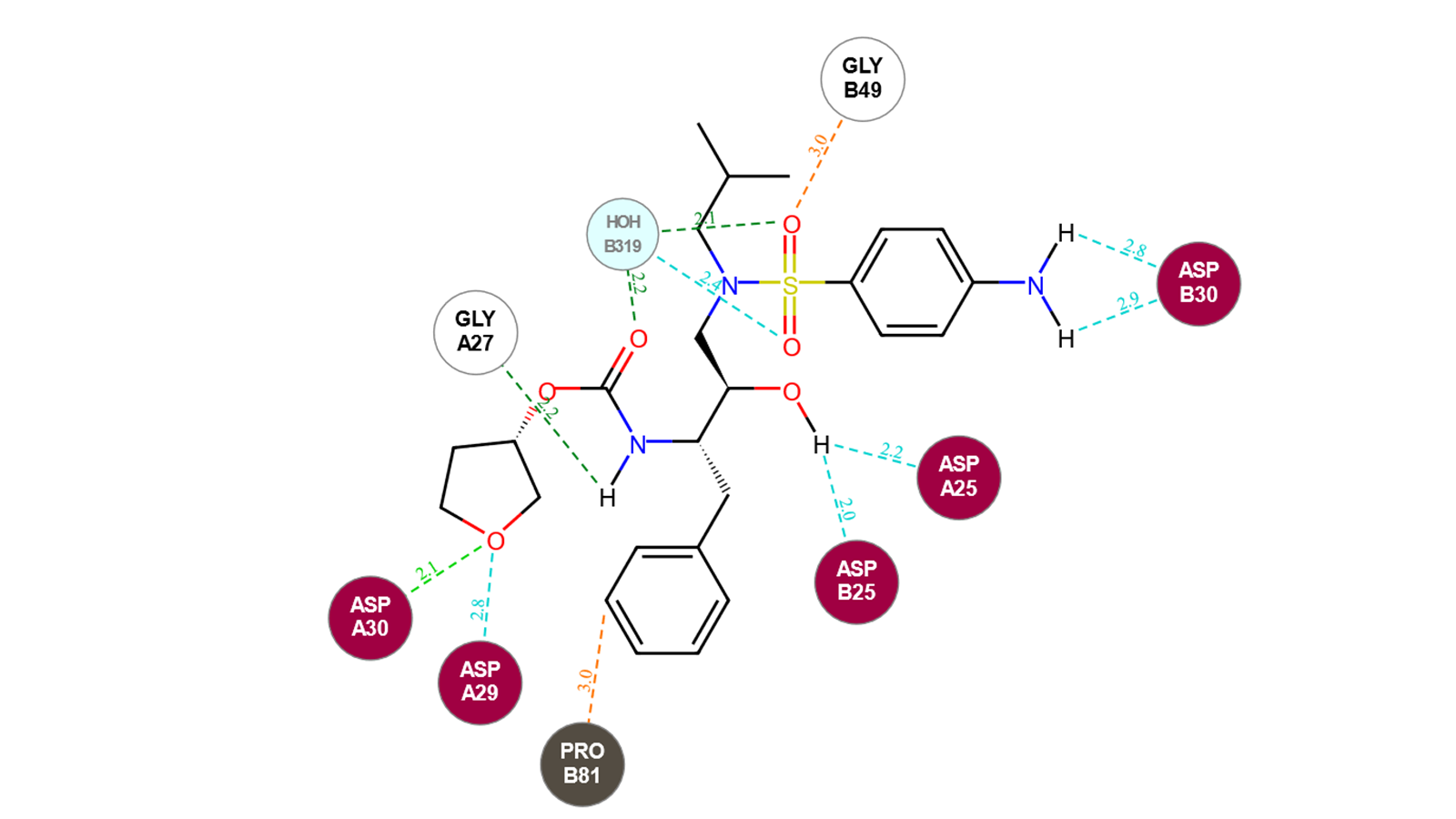
图3. Amprenavir与HIV蛋白酶活性位点(PDB 7N6X)的2D相互作用图。相互作用图例:淡绿色为强氢键(供体/受体距离在2.6Å以内,角度在理想范围之内),深绿色为一般氢键(供体/受体距离在2.6Å以内,角度在允许范围之内),青灰色为弱氢键(供体/受体距离在3Å以内,供体角度大于100°,受体角度为标准氢键+20°),桔黄色为立体碰撞,疏水接触未显示。
选择苯胺基团作为要替换的片段(图4高亮部分),选择连接点原子(图4中的蓝色圆圈)以确保该实验的结果仅包含原始配体的苯胺生物等排体。将蛋白7N6X的排除体积用作该实验的高级基于结构的选项。
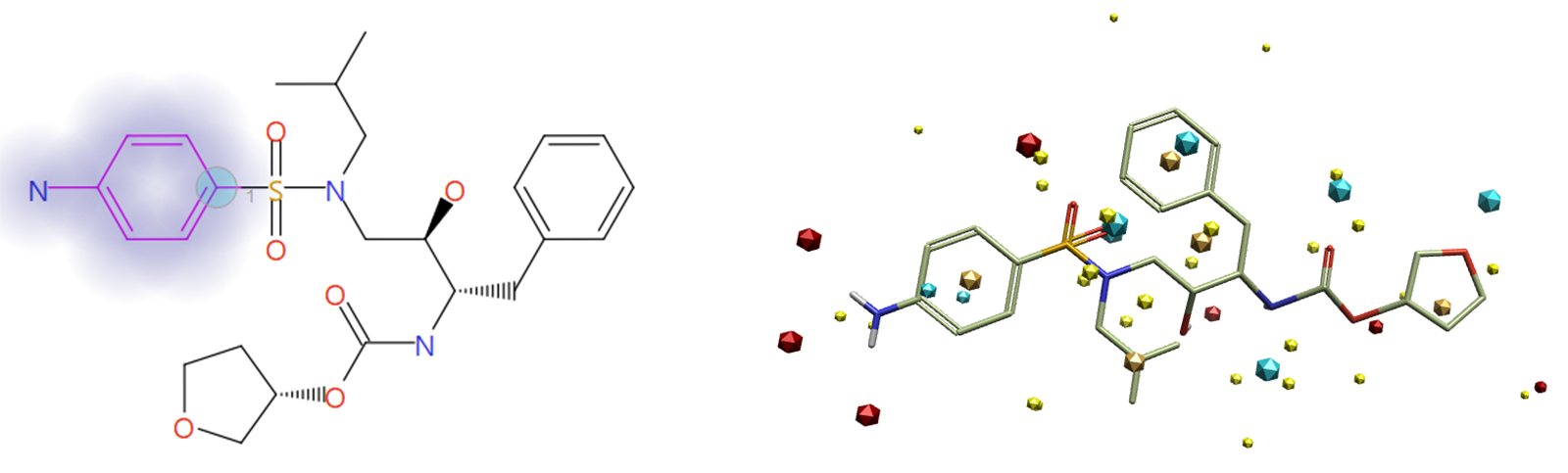
图4.在复合物结构中需要被替换的苯胺片段及其连接点(左)以及共晶配体的场点(右;蓝色:负静电场,红:正静电场,桔黄色:疏水场,黄色:vdW场)
在Spark生成的500个新结构中,经过严格的物理化学性质评估,选出了66个,以确保只考虑具有更好PK特性的化合物。然后进行对接实验,化合物按照LF ∆G打分5进行排序。
打分最高的11个化合物如表1所示。考虑到实验的目的是找到具有更好PK特性的化合物,因此从Spark分析中选择化合物3、7和8作为最佳结果。这些化合物还可以在活性位点发生额外的相互作用:化合物3含有一个一级酰胺,与Asp29和Asp30主链通过氢键相互作用;化合物7含有一个吡咯,与结晶水HOH322发生相互作用;化合物8异噻唑与Asp30主链的羰基发生相互作用。
表1. Amprenavir生物等排体替换的部分精选结果。汇总了所得到的新设计,其中第4列呈现了苯胺的替换片段。性质用颜色进行编码并与Amprenavir进行比较:绿色 – 更好的值,黄色 – 相同值,紫色 – 更差的值。图片使用Flare™7。
减轻毒性和脱靶效应
通过仔细设计和选择替换的化学基团,Cresset Discovery的专家们可以减轻苯胺类化合物可能引起的不良反应。此外,化学修饰可以通过减少脱靶效应来帮助改善药物的安全性。苯胺类在未经改变的情况下,可能会与脱靶相互作用,导致意想不到的副作用。通过用专一性更强、靶向性更好的化学结构来取代苯胺片断,可以大大降低脱靶相互作用的风险,提高药物的整体安全性。
BROMFENAC: 替换苯胺以克服毒性问题
溴芬酸(Bromfenac )是一个被美国食品和药物管理局批准上市的小分子非甾体抗炎药(NSAID)。其主要活性是通过抑制COX-2来阻断前列腺素的合成。在过去的二十年中,该药物因其毒性,特别是观察到的严重肝炎和肝功能衰竭病例(一些甚至需要移植)8,9,已被一些国家从市场上撤回。
有人假设溴芬酸的毒性作用与代谢有关。当含有苯胺的NSAIDs被肝脏代谢时,它们可以被转化为反应性代谢物。这些反应性代谢物可能会与肝脏细胞中的蛋白质和其他分子结合,特别是细胞色素P450酶,引起细胞损伤。在某些情况下,这些反应性代谢物会干扰正常的细胞过程,导致肝损伤。
选择苯胺基团作为要替换的片段(图5中高亮片断),选择连接原子(图5中的蓝色圆圈)以确保该实验的结果仅包含原始配体的苯胺生物等排体。
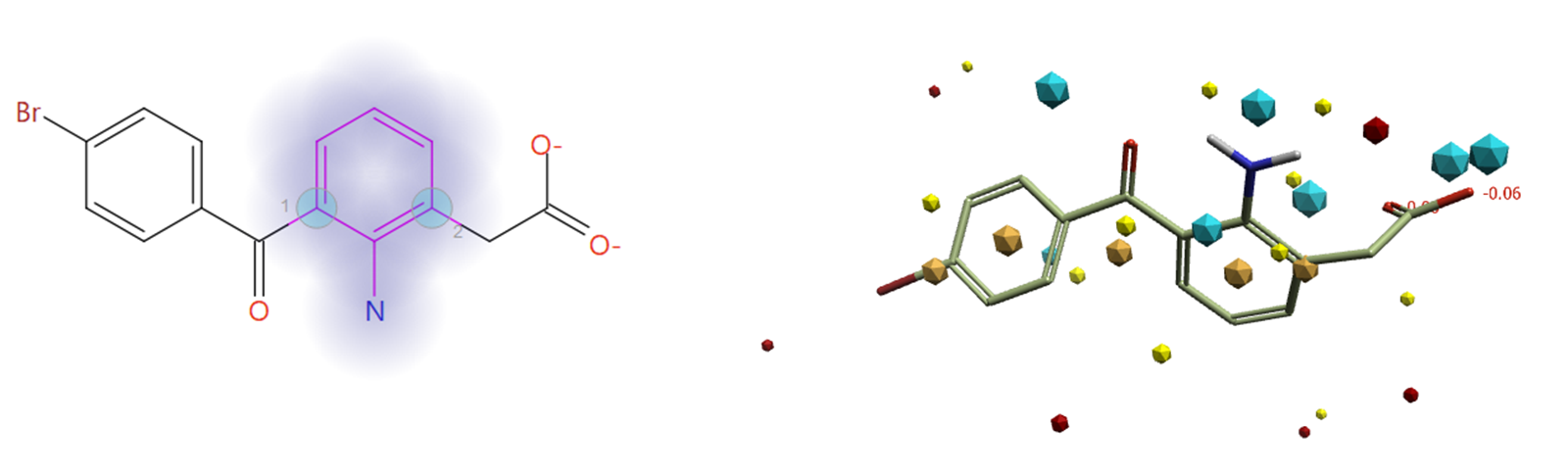
图5. BROMFENAC需要被替换的苯胺片段及其连接点(左)以及配体的场点(右;蓝色:负静电场,红:正静电场,桔黄色:疏水场,黄色:vdW场)
在产生的500个新化合物中,有27个是类药化合物,其中最好的7个结果如表2所示,其中展示了可能替代物的化学多样性。
表2. Bromfenac生物等排体替换的部分精选结果。汇总了所得到的新设计,呈现了苯胺的替换片段。
| 化合物 | 2D结构 | 替换片段 |
| Bromfenac |
|
– |
| 1 |
|
Di-methyl phenyl |
| 2 |
|
2-Hydroxypyridine |
| 3 |
|
Para-chloro pyridine |
| 4 |
|
2,5-Dimethyl pyrrole |
| 5 |
|
Indole |
| 6 |
|
3-Chloro pyrrole |
| 7 |
|
2-Methylthiophene |
结论
出于几个方面的原因, 用其他化学基团代替类药化合物中的苯胺片断是药物化学的一项决策。苯胺虽然用途广泛,但可能表现出代谢不稳定或毒性等不良性质。通过用不同的官能团替换苯胺,药物化学家可以微调化合物的药理学性质,增强生物利用度、溶解度或靶受体的选择性等等。此类替换也可以减轻与苯胺相关的潜在毒性或脱靶效应。
通过化学修饰解决代谢不稳定、毒性和脱靶效应等问题,Cresset Discovery的专家团队可以为更安全、更有效的药物发现铺平道路。通过使用Spark和Flare之类的创新工具,我们的专家以及制药行业同行可以为开发更好的药物取得重大进展。
参考文献
- Spark™, Cresset®, Litlington, Cambridgeshire, UK; https://www.cresset-group.com/spark; Cheeseright T., Mackey M., Rose S., Vinter, A.; Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation. J. Chem. Inf. Model. 2006, 46 (2), 665-676
- https://www.ebi.ac.uk/chembl
- Revertant mutation V48G alters conformational dynamics of highly drug resistant HIV protease PRS17. Burnaman, S.H., Kneller, D.W., Wang, Y.F., Kovalevsky, A., Weber, I.T. (2021) J Mol Graph Model 108: 108005-108005
- Protein Data Bank, https://www.rcsb.org
- Docking score optimized to provide an accurate estimate of protein-ligand binding energy, on the assumption that the pose is correct. Stroganov O.V. et al., Lead Finder: an approach to improve accuracy of protein-ligand docking, binding energy estimation, and virtual screening, J. Chem. Inf. Model. 2008, 48(12), 2371-2385
- Wildman-Crippen calculated logP. This value is derived by applying the rules of Wildman and Crippen and represents a good estimate of the actual logP of the molecule. S. A. Wildman, G. M. Crippen, Prediction of Physicochemical Properties by Atomic Contributions, J. Chem. Inf. Comput. Sci., 1999, 39, 868-873
- Flare™, Cresset®, Litlington, Cambridgeshire, UK; https://www.cresset-group.com/flare; Cheeseright T., Mackey M., Rose S., Vinter, A.; Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation J. Chem. Inf. Model. 2006, 46 (2), 665-676
- Wan Po, AL. (2007). Postmarketing Surveillance. In Comprehensive Medicinal Chemistry II (pp. 755-777). Elsevier
- Code of Federal Regulations 216.24: Drug products withdrawn or removed from the market for reasons of safety or effectiveness



