摘要:本文以几个结构多样的BRD4抑制剂为例,展示了FLARE的WaterSwap模块预测结合自由能的性能。结果表明,WaterSwap可以精确的预测配体的结合自由能。
作者:肖高铿
首次:2018-04-15
更新:2018-06-05。
一. BRD4抑制剂及算例介绍
溴结构域蛋白的异常与很多疾病密切相关, 比如癌症、炎症、免疫性疾病以及心血管疾病等。如溴结构域蛋白BRD4通过识别组蛋白上乙酰化的赖氨酸从而招募p-TEFb, 进而磷酸化RNA聚合酶Ⅱ, 最终引起下游基因表达, 如癌基因c-Myc基因等。而这些基因的表达异常会导致癌症等疾病。另外一些异常的溴结构域蛋白可能形成致癌突变的融合体, 如TY-82细胞中BRD4基因易位到睾丸核蛋白 (NUT) 基因座中产生BRD4-NUT 融合蛋白, 进而导致c-Myc过表达, 促进NUT 中线癌(NMC) 的形成。
BRD4抑制剂作为治疗药物的前景获得越来越多的关注,Aldegi M(2016)等人1成功地用GROMCAS精确计算过这11个BRD4抑制剂的结合自由能。其中10个抑制剂采用了抑制剂-BRD4复合物晶体结构做为计算的起点。本文以这10个结构多样的BDR4小分子抑制剂为算例(见图1)来考察FLARE/WaterSwap的预测结合自由能计算精度。
|
|
|
| 4OGI | 3MFX |
|
|
|
3U5J | 4MR4 |
|
|
|
4HBV | 3U5L |
|
|
|
4MR3 | 4J0R |
|
|
|
4OGJ | 3SVG |
图1. 10个结构多样的BRD4抑制剂及其与BRD4复合物的PDB代码
二.计算流程及结果
2.1 计算方法
在FLARE中下载对应的晶体结构,用Protein Preparation来准备结构。手动检查并修订(请务必检查配体结构的原子类型、键的类型、质子化状态,并编辑为正确的形式)配体结构。按照《FLARE教程 | WaterSwap计算结合自由能》操作步骤进行绝对结合自由能计算,迭代次数预设为3000步,获得预测值与误差,并与实验值进行比较。
预先说明:(1)Flare v1在准备复合物结构时会遇上已知的配体的质子化状态错误,请在Ligand框下右键点击该配体、并选择Edit,请修正后再计算。(2)使用远程计算时,为了能与本文计算结果一致,请将并发任务的初始迭代步数设为3000而不是初始的150(150适合于定性计算而不是定量,即便如此,我推荐该值为1000)。
2.2 计算结果
WaterSwap报告了四种结合自由能方法的计算结果,结合自由能预测值为四种方法的平均值,误差为四种结合自由能的最大值与最小值差值,如表1所示:
表1.结合自由能实验值与计算值比较
| PDB ID | Kd(nM) | ΔGexp(kcal/mol) | ΔGCalc(kcal/mol) | ΔGGROMACS(kcal/mol) |
|---|---|---|---|---|
| 4OGI | 37 | -10.14 | -33.73±1.46 | -10.4±0.5 |
| 3MXF | 49 | -9.97 | -32.60±1.40 | -9.5±0.4 |
| 4J0R | 386 | -8.75 | -25.26±1.29 | -9.4±0.8 |
| 3U5L | 640 | -8.45 | -24.25±1.20 | -9.9±0.8 |
| 4MR3 | 1142 | -8.11 | -22.85±1.50 | -9.2±0.5 |
| 4MR4 | 1142 | -8.11 | -21.28±1.50 | -5.9±0.5 |
| 3U5J | 2460 | -7.65 | -21.73±0.94 | -7.8±0.3 |
| 4OGJ | 164 | -9.26 | -35.57±1.48 | -9.4±0.8 |
| 4HBV | 23000 | -6.33 | -11.28±1.03 | -5.9±0.2 |
| 3SVG | 4800 | -7.26 | -19.01±1.30 | -7.7±0.4 |
ΔGCalc: Waterswap计算值;ΔGGromacs: Aldeghi M等人的GROMACS计算值
其中结合自由能实验值ΔGexp由Kd(nM)按公式ΔG=-RTlnK计算而来。WaterSwap计算的结合自由能不能与实验值直接比较,因为在计算结合自由能的过程中蛋白的alpha-碳原子被约束不动、仅侧链可以运动,蛋白熵的变化被低估了;配体在结合位点里也不能采集到全部的空间取向与构象变化,配体的构象熵变也被低估。虽然如此,可以将实验值与计算进行回归分析比较,结果如图2所示。
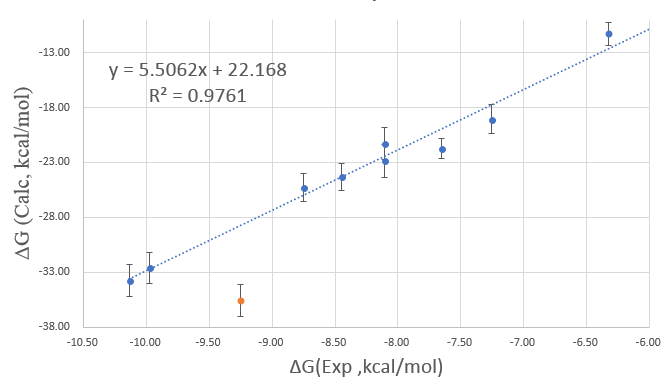
图2.BRD4抑制剂的结合自由能实验值与计算值比较(回归系数计算不包含4OGJ)
如图2所示,4OGJ计算的自由能值与实验值偏差较大,进一步分析可以判断为离群,但不影响对化合物活性好(nM水平)与差(uM水平)的分组。在不包含4OGJ的情况下,预测值与实验值的线性回归系数R2为0.976、Pearson相关系数为0.951,计算值与实验值有极强的相关性。同时,我们也注意到,测试用的化合物之间具有不同的骨架,这克服了相对自由能计算方法、以及传统QSAR对训练集具有公共骨架或母核的要求。
2.3 4OGJ离群原因的分析
4OGJ复合物结构的配体其Kd=164nM,相当于结合自由能-9.26kcal/mol。如图3所示,计算结果表明WaterSwap在2240步的时候达到收敛:移动平均线基本平直。此时,结合自由能的计算值为35.57±1.48kcal/mol。

图3.算例4OGJ的WaterSwap计算结果
图2显而易见地提示黄色的点4OGJ可能离群了。观察4OGJ复合物结构,如图4所示,发现该体系是二聚体,并且一个配体同时与两个链发生相互作用,而且两个配体还非常靠近。这与其它BRD4抑制剂只与一个链相互作用完全不同,而我们在进行计算的时候,仅用了其中一个A链(B链被删除)。这可能是该体系计算出现离群的原因。
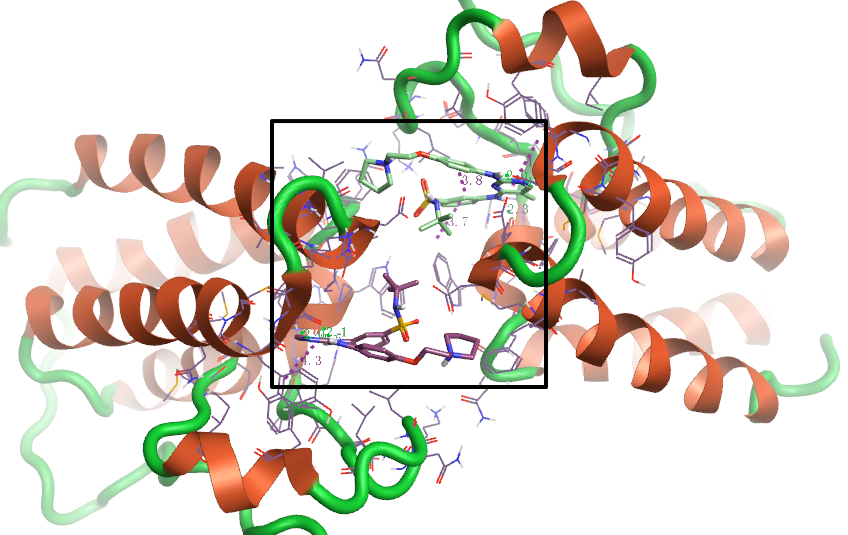
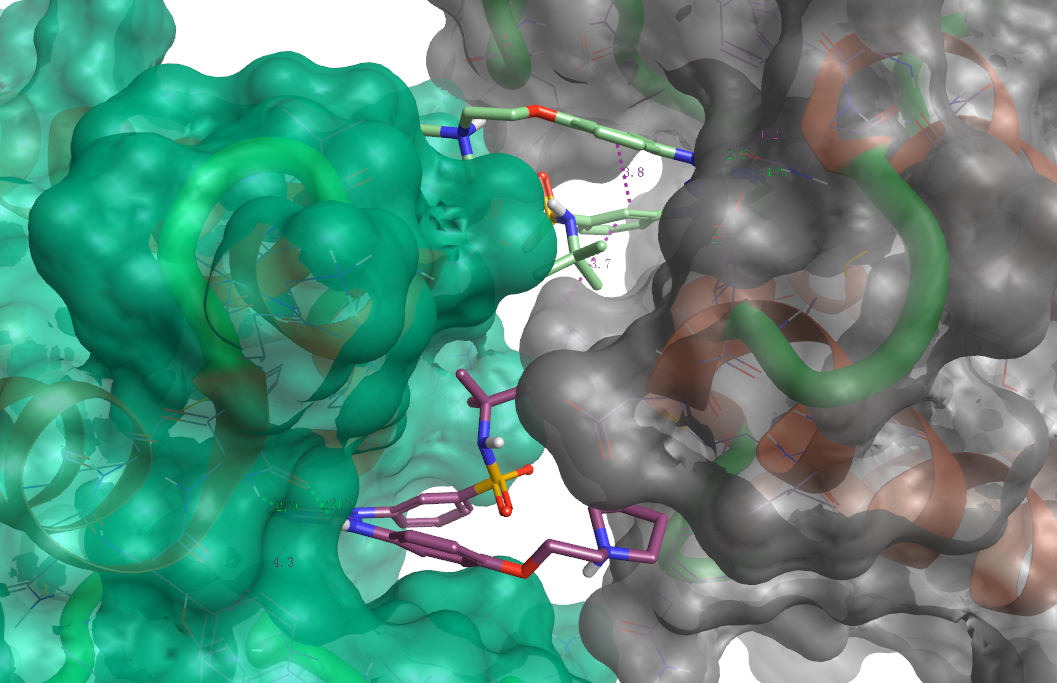
图4.算例4OGJ不同于其它BRD4抑制剂的结合模式:化合物跟两个链的蛋白都有相互作用。
2.4 复合物结构4MR4与4MR3的结合模式(2018-04-26更新)
(1)问题:结构相近,为什么结合模式差异很大
如图6所示,4MR4配体与4MR3配体仅差别一个羟乙基,4MR3配体是4MR4配体的一个子结构。但是4MR3配体并不采用4MR4配体的pose,为什么呢?这是一个有趣的问题。
|
|
|
| 4MR4 | 4MR3 |
图6.结合模式差异:在结合位点里取向完全不同
本部分尝试能否在结合自由能上能获得部分解释。根据X-RAY结果,可以预期:采取了4MR4配体的pose之后,4MR3配体的结合自由能应该要有较大幅度的上升。
(2)构造假想的4MR3配体结合模式
将4MR4配体的羟乙基切掉,模拟4MR3配体与4MR4配体采取同一个结合模式并计算结合自由能。
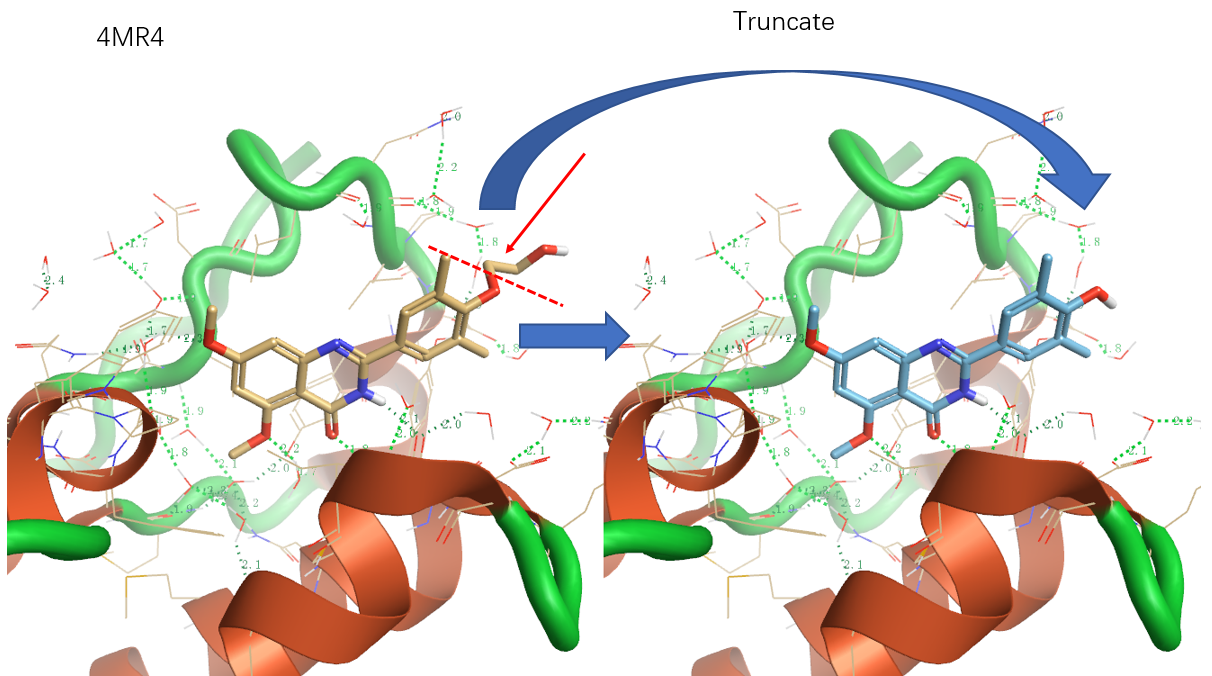
图7.将4MR4的羟乙基去除,模拟4MR3配体采用4MR4配体同样pose的结合模式:预期去掉羟乙基后体系的结合自由能应该上升
(3)结果
如图8所示,4MR4配体截取掉羟乙基后的结合自由能为-16.64±1.47kcal/mol。其中蛋白贡献的自由能为4.90kcal/mol,甚至对结合有点不利;去溶剂化对结合自由能贡献为-19.55kcal/mol。
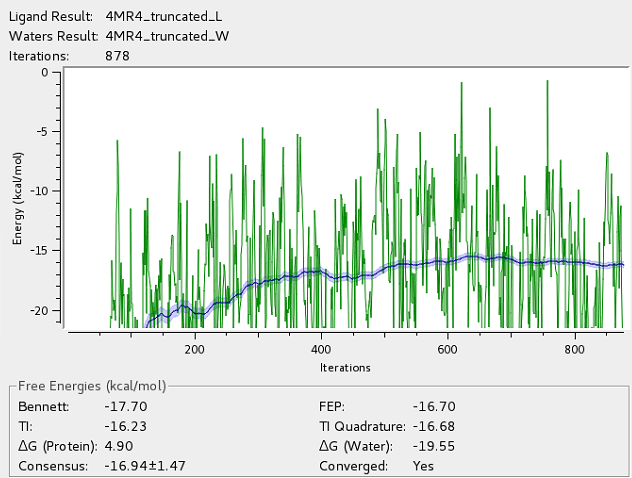
图8.将4MR4配体的羟乙基去除后结合自由能计算结果
如图9所示,4MR3晶体复合物结构计算的结合自由能为-22.85±1.50kcal/mol。其中蛋白贡献的自由能为-1.82kcal/mol;去溶剂化对结合自由能的贡献为-19.90kcal/mol。

图9.4MR3的结合自由能计算结果
如图10所示,4MR4晶体复合物结构计算的结合自由能为-21.28±1.50kcal/mol。其中蛋白贡献的自由能为6.84kcal/mol;去溶剂化对结合自由能的贡献为-25.93kcal/mol。

图10.4MR4的结合自由能计算结果
(4)小结
如表2所示,4MR4配体裁掉羟乙基之后,其计算的结合自由能由-22.85kcal/mol上升到-16.94kcal/mol,两者差异巨大,与预期相符。结合自由能的上升主要是因为两种不同结合模式与蛋白的相互作用作用能差异带来的。虽然4MR3比4MR3的配体在蛋白相互作用上有更大的优势,但4MR4的羟乙基在去溶剂化自由能上具有更大的优势,最终导致两者结合模式差异巨大(分别为-25.93kcal/mol与-19.55kcal/mol)。
表2.不同Pose的结合自由能与组成比较
| Ligand | Pose | ΔGCalc(kcal/mol) | ΔGProtein(kcal/mol) | ΔGWater(kcal/mol) |
|---|---|---|---|---|
|
|
4MR4 | -21.28±1.50 | 6.84 | -25.93 |
|
|
4MR4 truncated | -16.64±1.47 | 4.90 | -19.55 |
|
|
4MR3 | -22.85±1.50 | -1.82 | -19.90 |
比较4MR4-truncatd与4MR3这两个pose的自由能组成:相似的去溶剂化自由能,差异主要原子蛋白-配体相互作用能,后者比前者能量更低,4MR3的pose因此更稳定。
本计算再一次说明了正确的结合模式(pose)是正确预测结合自由能的基础:不正确的pose(4MR4-truncated)给出了错误的结合自由能(-16.64±1.47kcal/mol),与正确pose的结合自由能(-22.85±1.50kcal/mol)相比具有显著地差值。因此,采用正确的pose是WaterSwap计算成功的关键,使用docking预测的pose要努力排除不正确的pose(注:打分最佳的pose通常不是正确的pose,见博文:十种分子对接软件的性能评估)。
2.5 WaterSwap与打分函数比较
每个Waterswap结合自由能计算约花费16个物理核心两天的时间,这么昂贵的计算方法是否比快速、便宜的分子对接打分函数有明显优势呢?为了回答这个问题,首先我们需要选择一个合适的打分函数去打分。在Lead Finder分子对接性能测试一文中我们用CASF2013数据集评估了Lead Finder dG score、VS score以及Rank score的打分性能。发现Lead Finder的dG Score与实验值之间具有强相关(Pearson correlation = 0.66),优于其它的打分函数,因此选用Lead Finder的dG score与WaterSwap进行比较。
我们用Lead Finder的dG score对每个复合物结构打分,结果如图11所示:dG Score与实验值之间线性回归系数R2为0.338,Pearson相关性系数为0.582,具有弱相关性。在这个算例里,WaterSwap的打分性能(Pearson相关性=0.951)显著地优于分子对接Lead Finder的dG score打分函数。
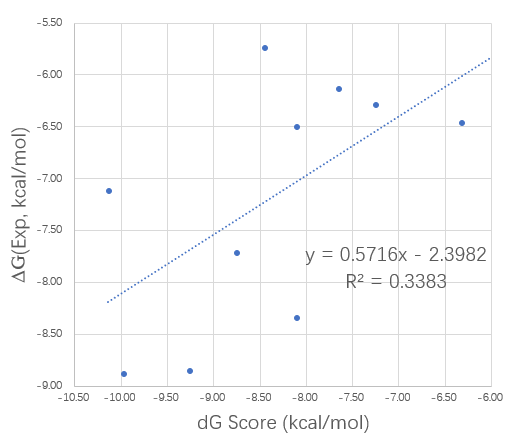
图11. 结合自由能实验值与分子对接打分值的比较
总的来说,在计算值与实验值相关性上,WaterSwap表现出极强相关而dG score代表的docking score表现出弱相关,两者的Pearson相关性分别为0.951与0.582。
2.6 WaterSwap与Aldegi M(2016)研究的比较
Aldegi M(2016)从10个晶体结构、1个对接计算的结构出发计算化合物的绝对结合自由能。我们将其中10个从晶体结构出发计算的结合自由能值与实验值进行比较,结果如图12所示:计算值与实验值线性回归系数R2=0.68,Pearson相关系数为0.824;而Waterswap在线性回归系数(R2=0.976)与Pearson系数(0.951)上表现更优。
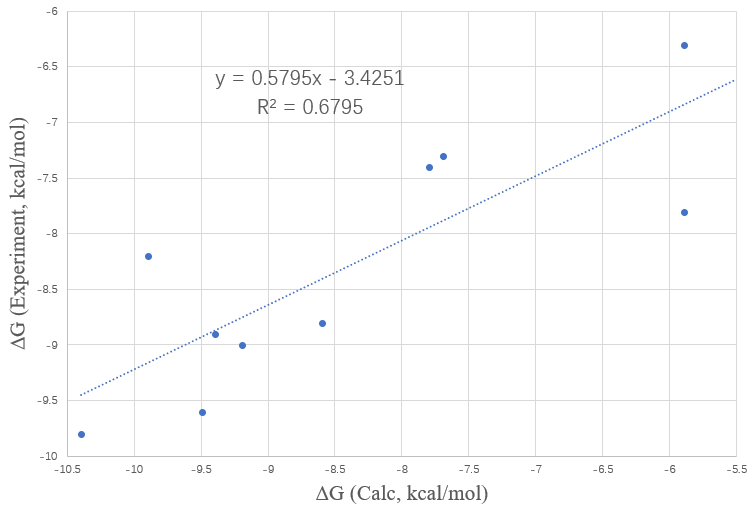
图12.在Aldegi M(2016)的研究中10个BRD4抑制剂(原文为11个化合物,这里将model的那个化合物刨去,以便比较)的结合自由能计算值与实验值比较
三. 结论
本文用FLARE/WaterSwap从抑制剂-酶复合物晶体结构为起始点计算了10个结构多样的BRD4抑制剂结合自由能,结果表明:
- WaterSwap的计算值可以精确的预测实验值;
- 可以适用于结构多样的化合物预测结合自由能;
- 出现一个离群的计算结果,但是不影响定性判断(活性高与低的区分),计算值与实验值具有极其显著的相关性;
- 复合物的初始结合模式很重要,决定了计算结果的对错;
- WaterSwap与dG Score代表的打分函数相比,前者与实验值的之间具有极强的相关性,而或者只有弱相关,Pearson相关性系数分别为0.951与0.582。这说明WaterSwap昂贵的计算物有所值。
- Waterswap与Aldegi M(2016)采用的GROMACS计算结合自由能相比,两者计算值与实验值的之间都的线性回归系数R2分别为0.976与0.680;而Pearson相关性系数分别为0.951与0.824。WaterSwap表现出更好的性能。
- 本文的算例从酶-抑制剂复合物晶体结构出发,并不能代表分子对接的pose结果如何。为此,我们准备了另一个HSP90的算例,采用手动对接,再进行结合亲和力计算。
四. 文献
- Aldeghi, M.; Heifetz, A.; Bodkin, M. J.; Knapp, S.; Biggin, P. C. Accurate Calculation of the Absolute Free Energy of Binding for Drug Molecules. Chem. Sci. 2016, 7 (1), 207–218.
五.相关主题
1.FLARE教程 | WaterSwap计算结合自由能
http://blog.molcalx.com.cn/2017/07/09/flare-tutorial-waterswap.html
2.Flare教程 | WaterSwap-如何整合公有云加速结合自由能的计算
http://blog.molcalx.com.cn/2018/02/14/cresset-broker-tutorial.html
3.案例 | 用SPARK分子内环化技术设计大环BRD4抑制剂
http://blog.molcalx.com.cn/2018/03/09/using-spark-to-design-macrocycle-brd4-inhibitors.html
六. 联系我们获取算例
我们提供已经计算好的算例以方便您重现计算结果。
试用软件下载:http://www.cresset-group.com/try-a-free-demo



