摘要:本文讲解了如何用KNIME从ChEMBL数据库提取特定靶标的化合物、并对化合物进行初步的数据清洗、多样性分析、构象搜索、聚类与基于配体的药效团建模。
一. 背景介绍
训练QSAR模型或药效团模型需要训练集化合物,如何快速的获取训练集化合物呢?ChEMBL、BindingDB、PubChem与ZINC为我们提供了狠多的便利。本文以EGFR激酶抑制剂为例,介绍如何用LigandScout的ChEMBLDB extractor(KNIME接口)从ChEMBL数据库里提取化合物。
二. 建立KNIME工作流
1. 确认靶标的信息
检索UNIPROT数据库1,野生型、人源的EGFR Kinase其获取号为P00533,ID为EGFR_HUMAN。
2. 使用ChEMBL-DB extractor提取EGFR激酶配体
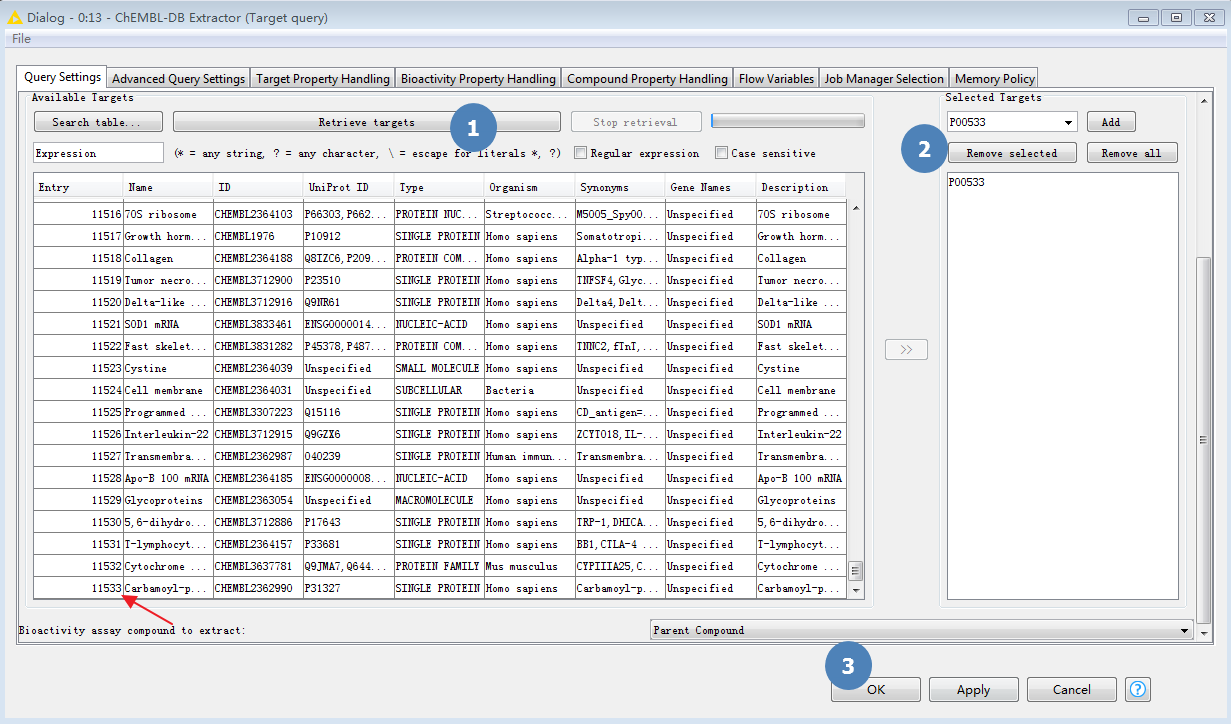
图1. EGFR激酶的配体提取
点击Retrieve target(操作1)会发现,ChEMBL数据库收集了11533个靶标的数据(2018-06-03测试),我们只对EGFR激酶感兴趣,用EGFR激酶的Uniprot获取号P00533作为过滤条件,提取ChEMBL数据库的EGFR激酶配体(操作2、3)。
3. 数据清洗
因为我们获取数据的目的是建立QSAR模型或药效团模型,因此需要考虑数据的可靠性,所以设定如下清洗流程:
- 仅保留数据可信度为8,9的数据
- 活性类型、活性值与单位
- 测试类型
- 多种活性值以及数据冲突的处理
- 性质过滤与脱盐
此类数据为分子水平实验获得,具有最高的可信度。
仅采用Ki,Kd,IC50或EC50的活性类型数据。除去活性值表示为“大于、小于”的活性类型,仅保留“等于”的活性值,并且活性单位为nM。
ChEMBL数据库的活性测试类型分功能与结合两种类型,我们仅对结合感兴趣,因此设定了结合类型过滤。
有的化合物活性值有多个文献报道,每个文献的活性值都不同,甚至有冲突。我们将均值作为化合物的活性值。你可以做任意的方式处理。
请参见:教程 | KNIME数据分析平台之化合物性质计算与过滤,这里不再重复介绍。脱盐不是必须,因为不管什么化合物的什么盐可以用其ChEMBL的母体化合物代替。
注意事项:
- 野生型与突变型的实验数据可能混合在一起
- 多种机制的数据混合在一起
请自行将实验方法的description部分提取出来,确认实验数据是野生型还是突变型的酶。
这个流程不能区分机制,需要手动阅读文献区分。请自行生成文献来源与摘要,在KNIME里是非常简单的事。
4.标签
在本练习中,将活性值小于1000nM的定义为活性化合物(active);将活性值大于100000nM的定义为非活性化合物(in-active),在两者之间的定义为可忽略(ignore)。对数据加标签以便我们建立QSAR与药效团模型的训练集与测试集。
在建立药效团模型或SAR模型时,我们需要用到训练集与测试集(图2)。在训练集与测试集里除了包含活性化合物外最好还需要包含非活性化合物(图2)。虽然文献里通常只用活性化合物建立模型,如果用上非活性化合物建立模型会更加准确(需要方法学验证加以确认)。活性(IC50小于1000nM)与非活性(IC50大于100000)的定义是相对的,我这里用只是练习用,请在真正研究中视情况而定。
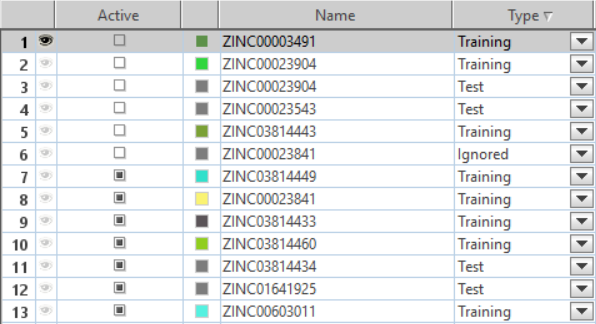
图2. 数据集分训练集与测试,各自都包含活性与非活性数据。信息用的越多,模型越准确。
5. 指纹图谱与多样结构类型数据集的准备
Ligandscout/Expert提供了ECFP指纹图谱计算节点与多样结构选择的计算节点(Diversity Picker),后者需要依赖前者的指纹图谱才能进行计算。
6. 化合物聚类
3D-QSAR或药效团建模的前提假设是:训练集化合物以相似的结合模式结合到同一个结合位点。因此,需要对化合物进行构象分析与药效团聚类:属于同一类的数据才作为建模的训练集与测试集。在本文,我们用Ligandscout的Pharmacophore cluster节点来实现聚类。
7. 药效团模型生成
Ligand-based Pharmacophore modeling节点自动按每个cluster做为训练集生成药效团模型;也可以手工选择对某个cluster训练药效团模型。
三. 小结
整个流程如图3所示。
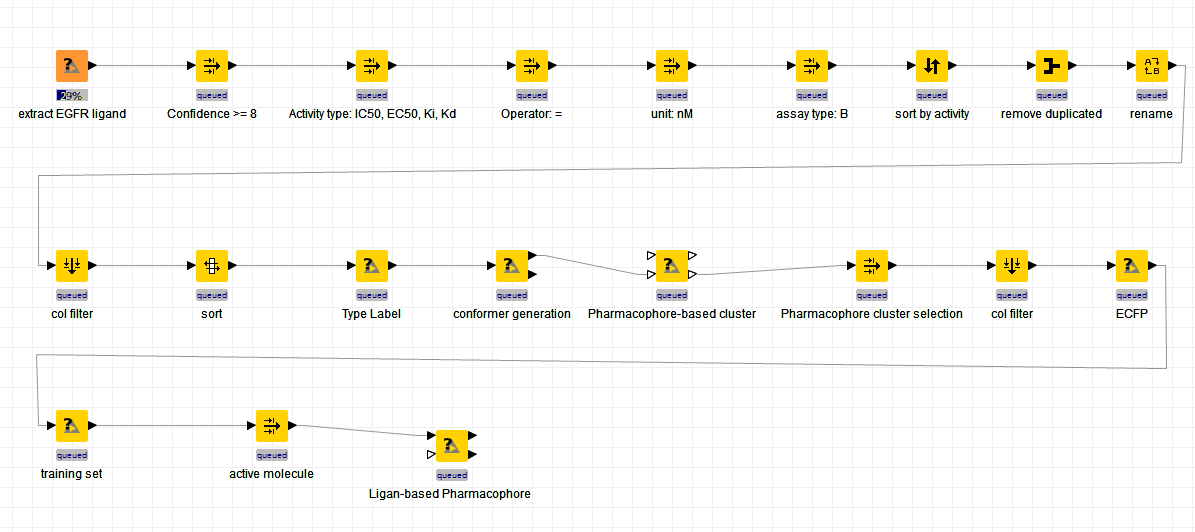
图3. 数据提起与药效团生成的整个KNIME工作流
下载工作流:ligand-based-pharmacophore.knwf
四. 接下来能做什么?
除了上面的药效团训练之外,接下来还可以:
- 3D-QSAR建模
- 虚拟筛选
- decoy生成与模型验证
五. 相关主题
- KNIME工作流| 化合物性质的计算与过滤
以常见的化合物物理性质计算为例,演示了如何建立KNIME工作流,实现:读入一个SDF格式化合物数据库,进行脱盐处理,计算性质(氢键受体、供体数量,油水分配系数,极性表面积,可选择键数量,分子量),性质过滤,输出结果到一个SDF格式文件。
六. 文献
- Uniprot. http://www.uniprot.org
- Knime. http://www.knime.org



