摘要:Flare V2是Flare的最新版本,一款开创性的基于结构的药物设计软件。Flare V2包含了:1)蛋白结构准备;2)蛋白静电相互作用势分析;3)静电互补性打分;4)WaterSwap结合自由能计算;4)3D-RISM水分子的位置值与稳定性计算;5)分子对接;6)Pyhton API任务自动化与自定义工作流。

FLARE能干什么
- 分析、获取关键的蛋白-配体静电信息以保护新的分子设计
- 用静电互补打分预测新设计化合物的活性
- 即时了解配体与蛋白的互补性并用于分子设计
- 设计新分子并将之对接到蛋白里
- 优化复合物以获得最佳的相互作用
- 计算自由能以指导先导物优化
- 计算蛋白里水分子的位置与稳定性
- 提供Python API、定制自己的工作流
用蛋白-配体静电信息完善分子设计
Flare用XED力场计算蛋白活性位点的蛋白与配体的静电特征,相互作用势分析提供了蛋白与配体结合过程的关键信息、并进一步帮助您完善分子设计。用蛋白-配体静电信息可以:
- 揭示结合位点的静电特征
- 比较整个蛋白家族的静电模式、设计选择性的化合物
- 设计具有更理想静电性质的分子以增强结合亲和力
- 比较多个配体的静电以理解构效关系
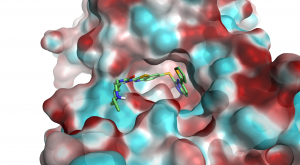 |
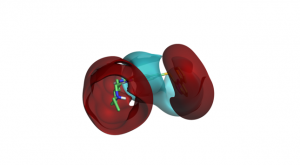 |
 |
| 结合口袋里静电势分布 | 配体的静电势等值图 | 静电互补性分析 |
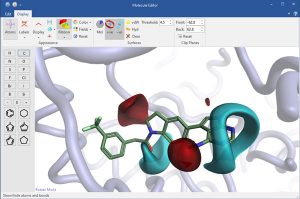 |
 |
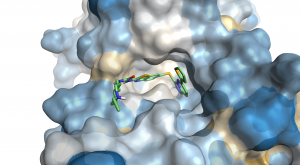 |
| 在结合位点里设计新分子、获得即时反馈、观察静电变化。 | 用蛋白相互作用势表面指导配体原子的放置 | 蛋白的溶剂可及表面用疏水性着色后的效果 |
快速、互动的活性预测
鉴于Flare V1发布之后,用户就不断提议:需要一款能够定量评估配体-蛋白静电互补性的方法,这一定将是一种对新设计分子的进行优先性排序快速、可靠的方法。
现在这个提议被满足了:基于Cresset的可极化XED力场,我们引入了静电互补打分(Electrostatic Complementarity,EC score)。EC打分可以快速地进行活性预测、可视化反馈、理解配体结合的机制、理解构效关系以及对化合物进行优先性排序。
通过蛋白相互作用势与配体的静电特征比较可以预测化合物的活性,Flare可以即时可视化静电互补打分、提供配体结构优化的提示。使用静电互补打分你可以:
- 预测新设计分子的活性
- 即时了解配体与蛋白的互补性
- 发现互补不足的位置、发现需要优化的空间
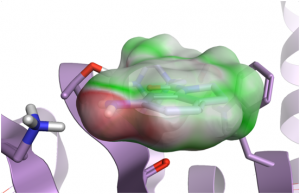 |
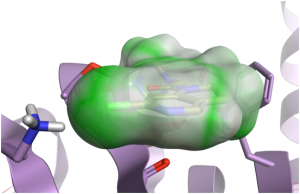 |
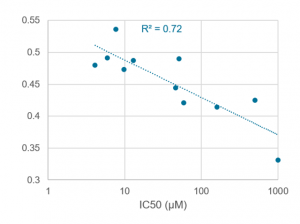 |
左图:活性较弱的XIAP衍生物与蛋白(PDB 5C7D)结合位点的互补性差些(红色区域);中图:而活性强的化合物互补性高(绿色区域);右图:静电互补打分与XIAP衍生物的活性值高度相关。
快捷、精确的分子对接
Flare采用用配体柔性、蛋白刚性的分子对接预测非共价结合的配体-蛋白复合物3D结构,还支持多蛋白构象的系综对接来模拟蛋白结合位点柔性的对接。在Flare的对接计算引擎是Lead Finder,你可以实现:
- 一次计算实验将多个分子对接到结合位点里
- 预测活性分子的3D结构
- 用BROKER可以并行计算将几百个化合物对接到结合位点里
- 进行非共价结合的对接计算
- 快速发下与结合位点匹配的化合物
 |
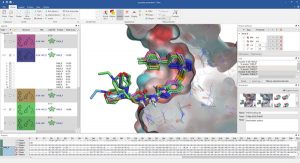 |
|
| 多个衍生物对接到一个蛋白里,并展示蛋白疏水表面 | 将化合物对接到多个蛋白里,并展示了PDB 5HLW的静电势表面 | |
Waterswap:计算结合自由能
WaterSwap用蒙特卡洛采样结合热力学积分方法、FEP等研究计算配体-蛋白绝对结合自由能。它可以用来:
- 研究配体的结合自由能
- 将能量拆解为每个残基的贡献以发现配体的优化空间
- 计算一系列配体的结合自由能(ΔG)以优选您设计的分子
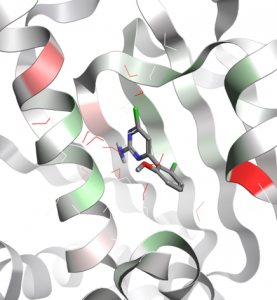 |
 |
 |
绿色残基表明配体从中获得相互作用的主要能量贡献;红色残基表示该残基更愿意与水结合、意味着改进配体设计的机会。
3D-RISM:是分子的位置与稳定性预测
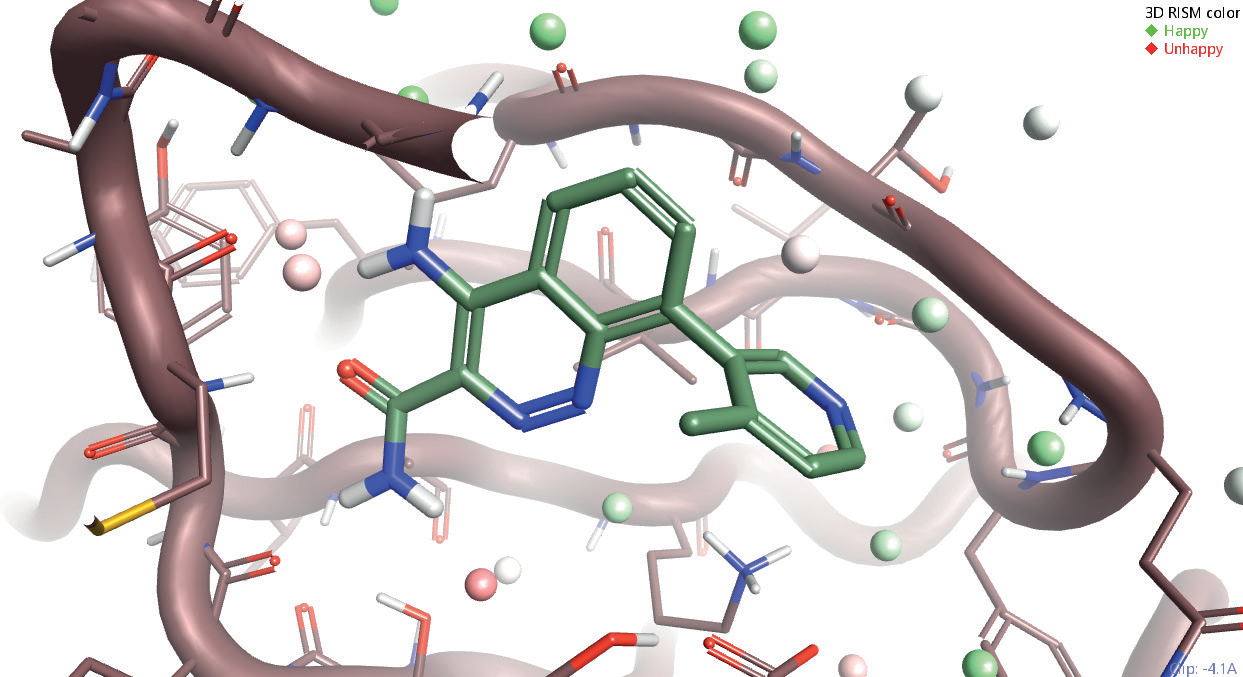
3D-RISM着色: 绿色-Happy; 红色-Unhappy
活性位点附近水分子的位置与能量学对于理解配体的结合是至关重要的。哪个水分子是紧密结合地、哪个水分子在能量上是不利的知识为结构-活性关系提供的重要信息,并可以帮您决定配体的原子应该如何摆放。Cresset的3D-RISM采用XED力场进行分析、为您提供可靠的水分子分析结果。用3D-RISM您可以:
- 理解蛋白的水能量学特征
- 计算配体周围水分子的优选位置
- 即使是笔记本,也能帮你分分钟设计新的配体、理解水分子的相互作用
- 在分析的时候考虑了稳定的水分子作用,让蛋白的相互作用势分析更加完美
用Python实现任务定制与自动化
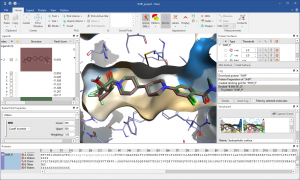
Flare提供Python API,你可以创建自己的工作流、任务自动化、自定义菜单等。通过Python API可以调用Flare的全部功能与化学信息学软件RDKit。使用API你可以建立自动化任务、按需定制自己的界面。还可以与外部的图形化、统计学软件、Jupyter笔记本等整合。命令行Python脚本还可以让你进行简单的自动化Flare计算。
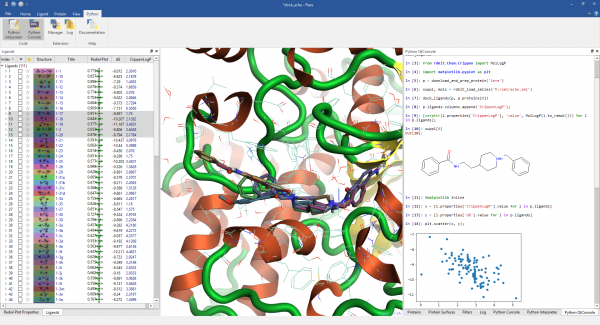
可以直接键入Pyton命令、以文本或图形方式展示结果。上面的截图展示了一个脚本实例:调用RDKit读入一系列SMILES编码的化合物、用XED力场生成3D结构、用Lead Finder将化合物对接到蛋白里、绘制了RDKit计算的logP与Lead Finder预测的结合自由能(ΔG)的散点图。Flare的Python API可以与其它Python模块组合用来产生、分子结构。
授权类型
| Flare | Flare Viewer | Flare for Academics | |
|---|---|---|---|
| Commercial organizations | Yes | Yes | |
| Academics | Yes | Flare for Academics recommended | Yes |
Protein-centric operations |
|
|
|
| Dedicated protein table enabling rapid inspection of specific chains or residues | Yes | Yes | Yes |
| Protein sequence alignment and superposition | Yes | Yes | Yes |
| Protein preparation | Yes | Yes | |
| Perform single point mutation for your proteins | Yes | Yes | |
| Refine the structure of the protein active site by flipping and changing the protonation/tautomeric state of relevant residues | Yes | Yes | Yes |
| Protein minimization using the XED force field | Yes | Yes | |
| Calculate and color protein molecular surfaces by atom, secondary structure and hydrophobicity | Yes | Yes | Yes |
| Calculate and color protein molecular surfaces by electrostatic potential, Electrostatic ComplementarityTM | Yes | ||
| Display protein interaction potentials for the protein active site | Yes | ||
| Control every protein surface with individual display options in the dedicated protein surfaces table | Yes | Yes | Yes |
| 3D-RISM for water stability and positioning | Yes | ||
Ligand-centric operations |
|
|
|
| Easy access to all ligand actions in the dedicated ligand tab menu | Yes | Yes | Yes |
| Dedicated ligand table to store all ligands in your project with full visibility control, sortable on any column | Yes | Yes | Yes |
| Protein association (every ligand has a parent protein) | Yes | Yes | Yes |
| Calculated physico-chemical properties for each ligand | Yes | Yes | Yes |
| Ligand-design in the active site (molecule editor) | Yes | Yes | Yes |
| Perfect ligand design using ligand and protein electrostatics | Yes | ||
| Perfect ligand design using ligand electrostatics | Yes | Yes | Yes |
| Show Electrostatic Complementarity maps towards the protein of interest | Yes | ||
| Fast and interactive activity prediction using Electrostatic Complementarity scores | Yes | ||
| Calculate radial plot multi-parametric scores to select the compounds with the best properties | Yes | Yes | Yes |
| Minimize your ligands in the protein active site | Yes | Yes | |
| Quick, easy and accurate docking | Yes | Yes | |
| Tackle the flexibility of the protein active site with ensemble docking | Yes | Yes | |
| Investigate ligand-protein energetics with WaterSwap | Yes | ||
| Filter on numerical values, structure, tags | Yes | Yes | Yes |
GUI |
|
|
|
| Ribbon menu structure for quick identification of commands and controls | Yes | Yes | Yes |
| Drag and drop ligands between protein and ligand tables | Yes | Yes | Yes |
| Visualize protein-ligand complexes | Yes | Yes | Yes |
| Visualize protein-ligand interactions | Yes | Yes | Yes |
| Focus on active site | Yes | Yes | Yes |
| Compare protein-ligand complexes | Yes | Yes | Yes |
| Grid the 3D window by protein and ligand to compare and contrast | Yes | Yes | Yes |
| Capture 3D view to the storyboard to track and communicate ideas | Yes | Yes | Yes |
| Summary and detailed logging of calculations and events | Yes | Yes | Yes |
| Extended picking widget that enables complex queries | Yes | Yes | Yes |
Python |
|
|
|
| Create and automate workflows using the Python API | Yes | With supported version | Yes |
| Access the RDKit cheminformatics toolkit | Yes | With supported version | Yes |
| Upgrade Flare with Python modules for graphing, statistics, Jupyter Notebook | Yes | With supported version | Yes |
| Automate and distribute Flare calculations using pyFlare and Cresset released Python scripts and snippets | Yes | Yes | |
| Expand the functionality of the Flare GUI using Cresset released Python extensions | Yes | With supported version | Yes |
Remote processing |
|
|
|
| Cresset Engine Broker | Optional – please enquire | Optional – please enquire | |
Support |
|
|
|
| Email support | Yes | Optional – please enquire | Limited* |
* In some countries – please enquire to see if you are eligible for support.



