摘要:3D-RISM可以用来预测结合位点里水分子的位置与稳定性:哪个水分子是紧密结合地、哪个水分子在能量上是不利,为结构-活性关系提供的重要信息,并可以帮您决定配体的原子应该如何摆放。用3D-RISM可以:理解蛋白的水能量学特征;设计新的配体、理解水分子相互作用;考虑稳定的水分子作用,让蛋白的相互作用势更加完美。
肖高铿/2017-06-18
一. 前言
全部的生化反应都发生在水环境中,因此水分子的计算非常重要。3D-RISM可以用来预测结合位点里水分子的位置与稳定性:哪个水分子是紧密结合地、哪个水分子在能量上是不利,为结构-活性关系提供的重要信息,并可以帮您决定配体的原子应该如何摆放。用3D-RISM您可以:理解蛋白的水能量学特征;设计新的配体、理解水分子相互作用;考虑稳定的水分子作用,让蛋白的相互作用势更加完美。
二. 操作步骤
1. PDB结构下载与准备
下载PDB 4ZLZ并按默认流程准备复合物结构,操作步骤参见《Flare教程|分子对接-结合模式预测》的蛋白结构下载与准备部分。
准备完毕,配体4RV在在Ligand表单里,准备好的受体为Protein表单的4ZLZ_P, 见Figure 1。
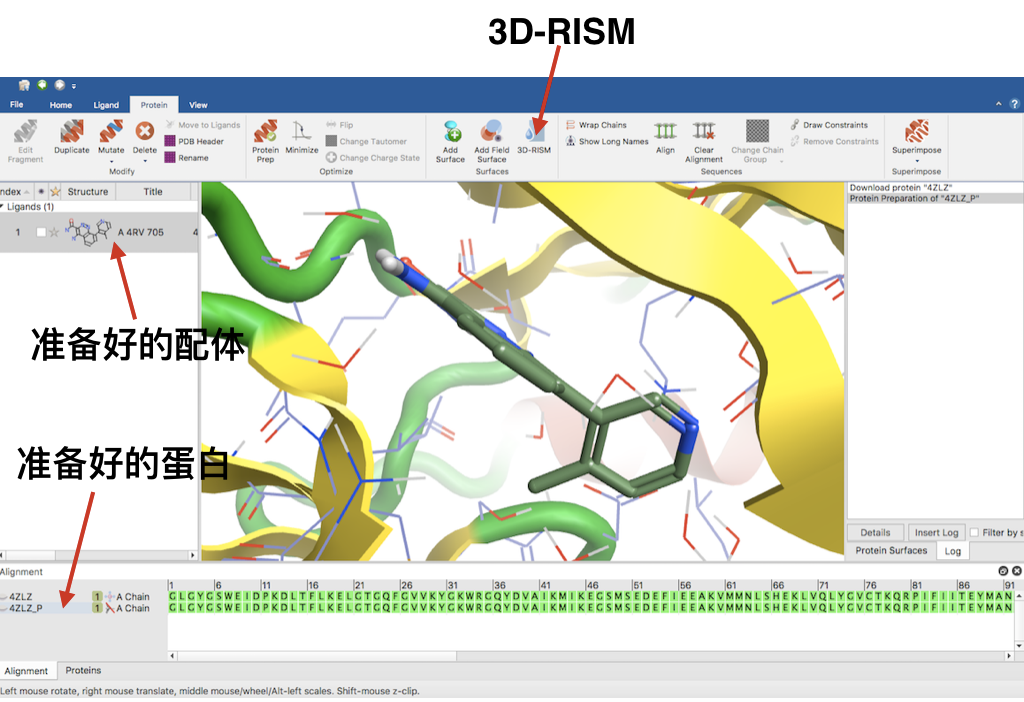
Figure 1. PDB的4ZLZ结构准备
2. 3D-RISM计算
- 3D-RISM的参数设定
- 结果可视化分析
在Ligand表单单击选中配体4RV;在Protein表单单击选中蛋白4ZLZ_P;再单击3D-RISM命令(见Figure 1)弹出3D-RISM计算对话框(Figure 2)。
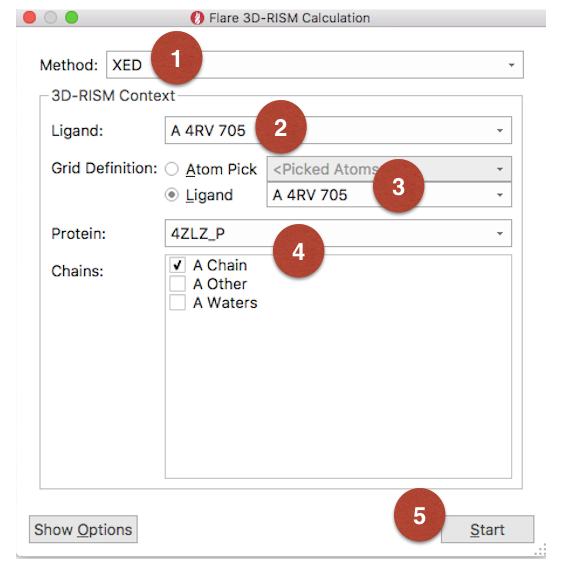
Figure 2. 3D-RISM的参数设定
参数设定如Figure 2所示:
Method:XED;Ligand: A 4RV 705;GRID definition: Ligand > A 4RV 705;Protein: 4ZLZ_P;Chain: A Chain。
点击Start按钮开始计算。
3D-RISM的计算完毕后,再蛋白表面视窗中自动用等值图展示计算结果。如果蛋白表面视窗没有打开请点击View->Docks->Protein Surface打开。
如图3所示,氧(ρO) 与氢(ρH)密度等值图反映了3D-RISM计算发现氧、氢原子(即水分子)高概率出现的区域,等值图根据 ΔG进行着色。负值 ΔG(绿色)代表“Happy”(favorable)水分子:说明3D-RISM预测这些水分子呆在蛋白里比呆在水中更加稳定;正值ΔG(红色)等值图表示”Unhappy”(unfavarable)水分子,这些水分呆在蛋白里不如呆在水中稳定,因此容易被配体替换。其中显示等值图的密度阈值可以通过调节转子来调节。
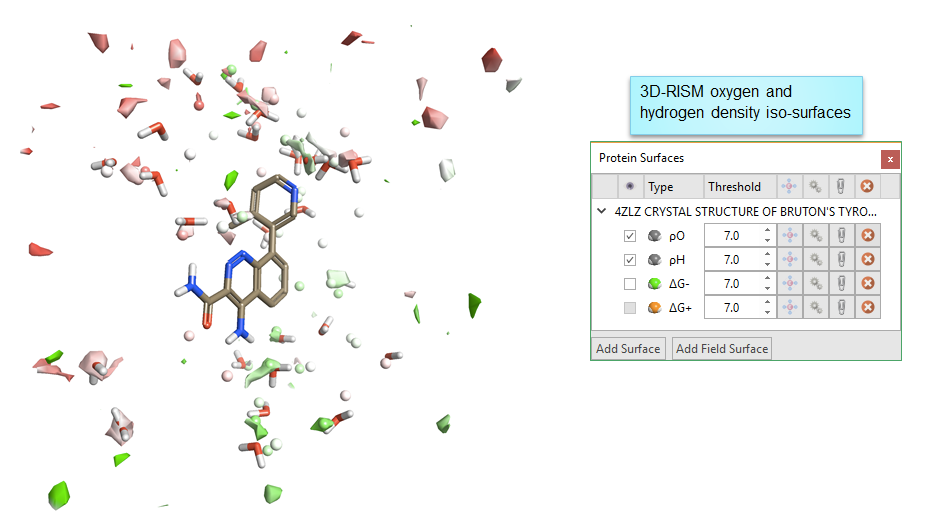
Figure 3. 3D-RISM的氧(ρO) 与氢(ρH)密度等值图: 绿色为Happy Water,红色为Unhappy Water。
如图4所示,注意:3D-RISM ΔG等值图的负值ΔG区(绿色)与正值ΔG区(红色)不一定是高密度的氧原子、氢原子区。
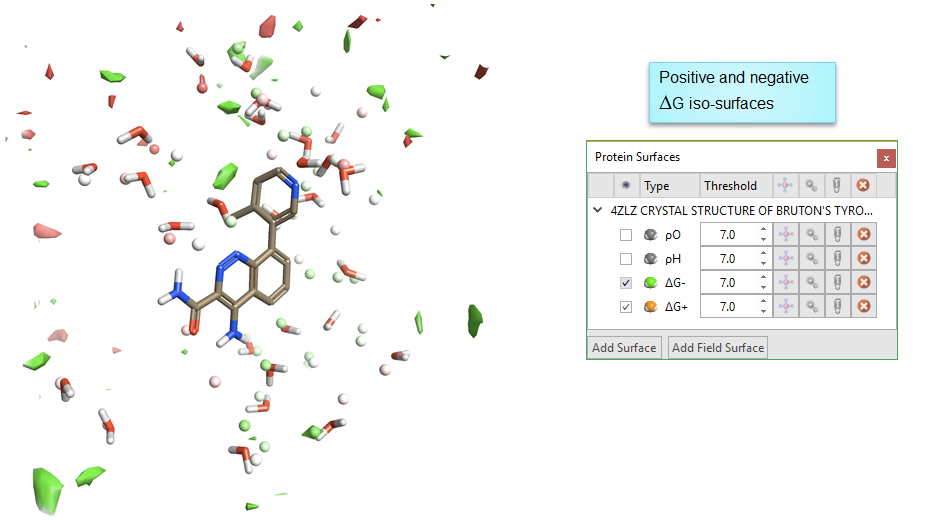
Figure 4. 3D-RISM的ΔG等值图
如图5所示,3D-RISM计算还会再生成一条新的水Chain添加到蛋白结构里,标示为Chain B(或者C,D…) 3DR。这个链上的水分子对应于3D-RISM预测的水分子高密度区。”Happy”水分子为绿色(ΔG负值),”Unhappy”水分子(ΔG正值)为红色。
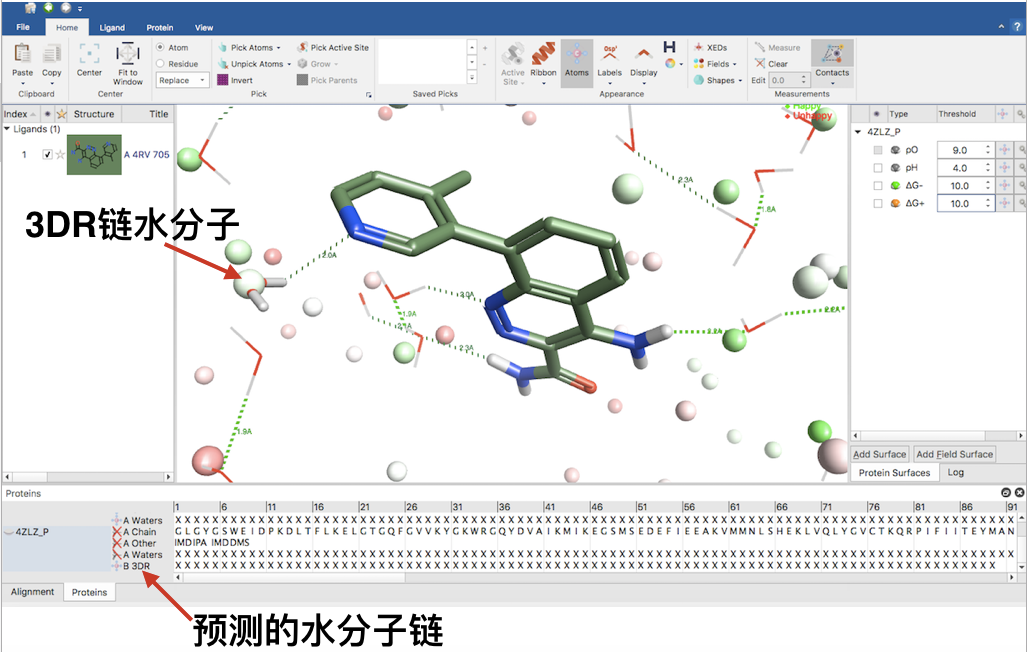
Figure 5. 3D-RISM计算完毕,在Protein表单增加了一个Chain B 3DR;该链的水分子在3D视窗区展示为球状。
四. 视频演示
五. 之后可以做什么
- 分子对接中蛋白结构的准备
- SPAKR骨架替换中设计替换水分子的配体
- Ligandscout里将Unhappy水分子设为配体的一部分,得到新的药效团模型
- 基于结构的设计,指导配体结构改造
参见教程:http://blog.molcalx.com.cn/2017/06/15/displacing-crystallographic-water-molecules-with-spark.html
六. 联系我们
试用下载:http://www.cresset-group.com/try-a-free-demo



