摘要:OMEGA负责生成小分子的3D构象,专门为计算机辅助药物设计中大规模数据库化合物的构象分析而设计,它可快速、可靠地产生多构象数据库。OMEGA生成的构象数据库可用于分子对接虚拟筛选(比如FRED)、形状比较虚拟筛选(比如ROCS)、以及药效团识别与虚拟筛选。本教程讲解如何用OMEGA进行构象搜索的原理与方法。
一.OMEGA介绍
OMEGA是专为药物设计所需的大规模化合物构象分析而设计。它可以高速、靠谱地产生多构象数据库。
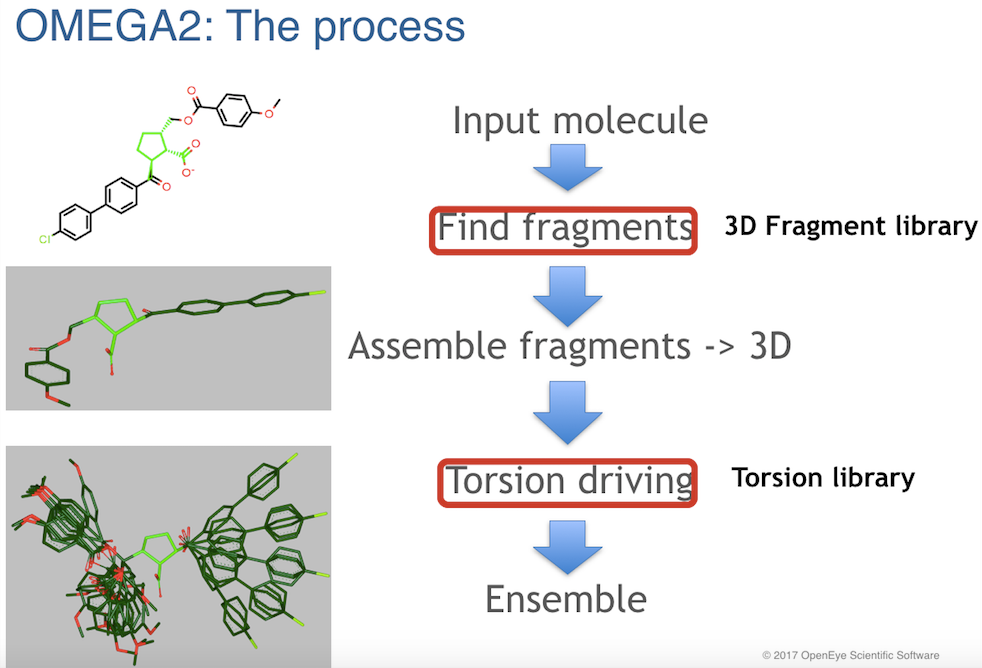
Figure 1. OMEGA的算法
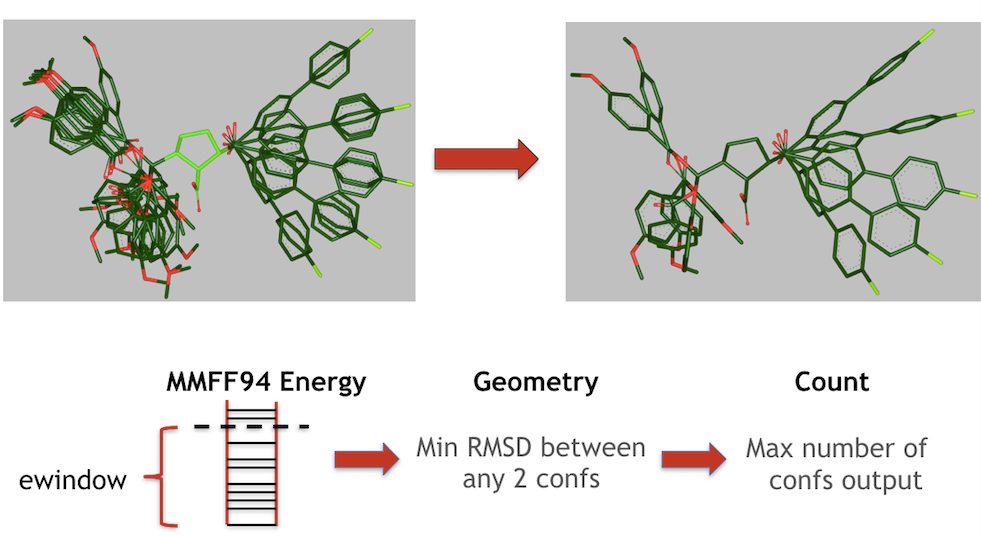
Figure 2. OMEGA对构象进行聚类、按能量排序
二.OMEGA的性能
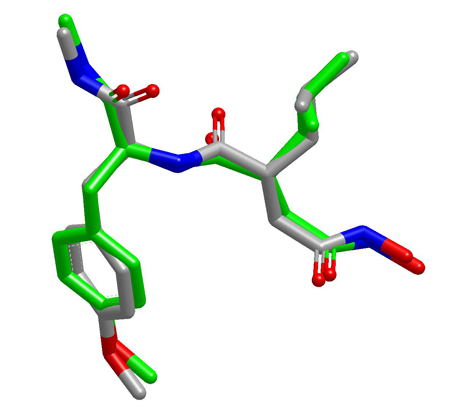
Figure 3. OMEGA可以高质量地重现PDB里的生物活性构象
OMEGA的特性
- 非常快速(1-2 sec/molecule), 穷尽地、基于规则地构象搜索
- 用距离几何法将化合物从1D或2D生成3D结构
- 基于RMS距离与构象能生成多样的构象集
- 用户自己可以定义搜素的分辨率
- 全自动的结构特征叠合
- 优秀地重现类药分子的固态结构
- 支持MPI在各种计算平台上进行分布式计算
三.OMEGA教程
3.1 Windows操作系统
- OMEGA计算环境
- 使用OMEGA
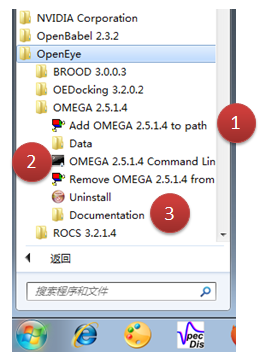
Figure 4. 在Windows开始菜单>OpenEye>OMEGA下可以找到OMEGA的全部资料
(1)将OMEGA加入到PATH环境变量,以便您可以在命令行直接键入OMEGA支持的命令即可使用
(2)点击OMEGA command line则直接进入OMEGA环境,可以直接开始使用
(3)Documentation为OMEGA的使用说明
进入OMEGA命令行后,获取OMEGA的全部使用参数,可以键入命令:
omega2.bat –help all
假设待构象搜索的文件为foo.smi, 采用预设值进行构象搜索,输出为oof.oeb.gz,则键入命令:
omega2.bat -in foo.smi -out oof.oeb.gz
3.2 Linux或Mac操作系统
设定好环境变量后直接键入命令omega2:
[me@linuxbox me]$omega2
如果使用–help all,可以获得全部参数,强烈建议阅读
[me@linuxbox me]$omega2 --help all
3.3 多核并行
1. 单机多核并行
语法:命令 -mpi_np [核心数] 参数
单机8核并行的命令如下:
[me@linuxbox me]$omega2 -mpi_np 8 -in foo.sdf -out foo.oeb.gz
2. 跨节点并行
假设需要在5个节点上计算,每个节点32核心。则先需要准备一个节点文件(host.txt),内容如下:
1 2 3 4 5 | c1 slots=32 c2 slots=32 c3 slots=32 c4 slots=32 c5 slots=32 |
那么该跨节点并行的命令如下:
[me@linuxbox me]$omega2 -mpi_hostfile host.txt -in foo.sdf -out foo.oeb.gz
四.文献
1. Conformer Generation with OMEGA: Algorithm and Validation Using High Quality Structures from the Protein Databank and Cambridge Structural Database, P.C.D. Hawkins, A.G. Skillman, G.L. Warren, B.A. Ellingson and M.T. Stahl, J. Chem. Inf. Model., 2010, 50, 572.
2. Conformer generation with OMEGA: Learning from the dataset and analysis of failures, Hawkins, P.C.D., Nicholls, A.N., J. Chem. Inf. Model., 2012, 52, 2919.
3. Conformational Analysis of Drug-Like Molecules Bound to Proteins: An Extensive Study of Ligand Reorganization upon Binding, E. Perola and P.S. Charifson. J. Med. Chem. 2004,47, 2499-2510.
4. Comparison of Conformational Analysis Techniques to Generate Pharmacophore Hypotheses Using Catalyst, R. Kristam, V.J. Gillet, R.A. Lewis and D. Thorner. J. Chem. Inf. Model.2005, 45, 461-476.
五.免费试用
请参考:http://blog.molcalx.com.cn/2017/05/31/openeye-evaluation.html



