不同寻常的蛋白配体相互作用-PDB可视化算例
摘要:本文试图为Kuhn 等人(2019)总结的蛋白-配体相互作用模式提供PDB算例,并用基于结构的软件Flare Viewer准备好,以便可以用故事板(Story board)的方式对各种相互作用模式进行3D可视化分析、浏览、编辑、注释,方便大家进一步了解这些相互作用。
肖高铿/2021-05-09
1. 前言
本文试图为Kuhn 等人(2019)总结的显著高频非经典蛋白-配体相互作用模式提供PDB算例,并用免费的图形化软件Flare viewer准备好,以便可以用故事板(Story board)的方式对各种相互作用方式进行3D可视化分析、浏览、编辑、注释,方便大家进一步了解这些相互作用。本文重点关注一个原子或基团变化而引起活性巨大变化的应用案例,这需要大量的时间进行准备,所以本文的更新将是漫长的过程。
在Kuhn等人的视线法中,需要用到bondi原子半径来计算接触的距离。假设原子半径为rL的配体原子L与原子半径为rP蛋白原子P之间的距离为d,若满足如下方程(1)的关系:
$$
d\ \leq\ r_{L} + r_{P} + \ x
$$
则认为配体原子L与蛋白原子P之间具有接触。其中,x为增量0.5。半径参考了Manjeera(2019)等人的文章,这里做成一个字典方便使用。
1 2 3 4 5 6 7 | radius = { 'H':1.10, 'He':1.40, 'Li':1.81, 'Be':1.53, 'B':1.92, 'C':1.70, 'N':1.55, 'O':1.52, 'F':1.47, 'Ne':1.54, 'Na':2.27, 'Mg':1.73, 'Al':1.84, 'Si':2.10, 'P':1.80, 'S':1.80, 'Cl':1.75, 'Ar':1.88, 'K':2.75, 'Ca':2.31, 'Ga':1.87, 'Ge':2.11, 'As':1.85, 'Se':1.90, 'Br':1.83, 'Kr':2.02, 'Rb':3.03, 'Sr':2.49, 'In':1.93, 'Sn':2.17, 'Sb':2.06, 'Te':2.06, 'I':1.98, 'Xe':2.16 } |
Flare viwer提供了pick功能,可以方便找到与特定配体原子接触的蛋白原子,并进行角度与距离测量,具体操作过程参见博文在基于结构的设计中用Flare Viewer来提高视觉分析的效率。
如果需要实现可视化分析自动化,可以参考《在Flare里将与某个残基相互作用的配体过滤出来》提供的计算蛋白-配体原子对距离的代码来实现。
2. 芳香碳键连的氯与蛋白受体相互作用
2.1 Cl…C_ali_apol相互作用算例
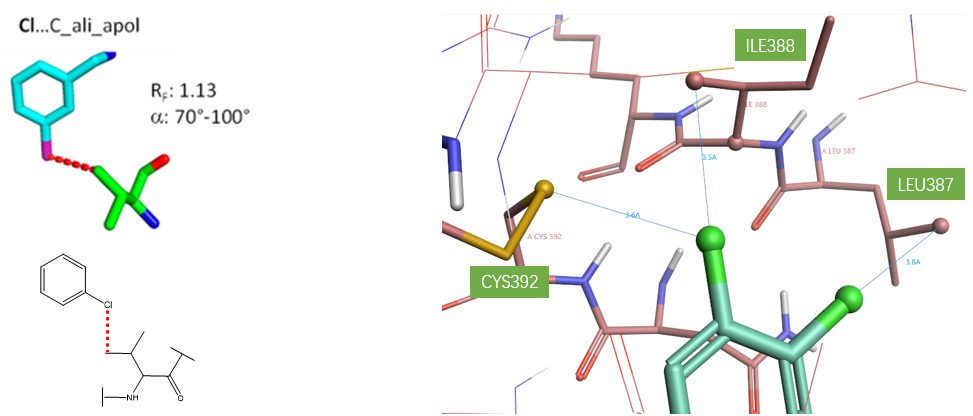
Figure 1. Cl…C_ali_apol相互作用, PDB 6A7P.相互作用的原子用球表示
找到一个纯粹通过疏水相互作用而提高活性的例子很难,但可以找到主要通过疏水相互作用为主的例子(见视频1),Sakurama(2019)等人[1]的研究就提供了非常好的应用算例。如图2所示,比起Deschloro-ARP,ARP(Aripiprazole,PDB 6A7P的共晶配体)在苯环上多了两个氯因而有更多的Cl…C_ali_apo相互作用,从而与人血白蛋白结合亲和力提高了50倍。
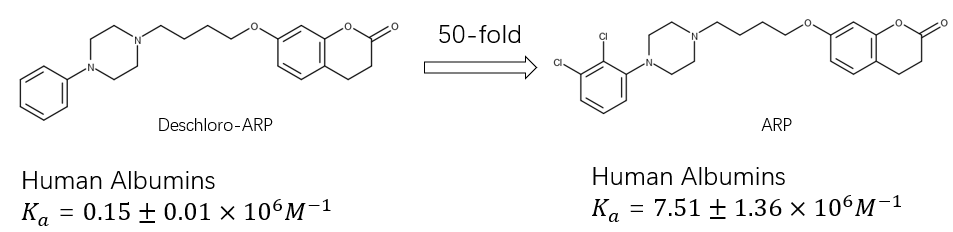
Figure 2. 比起Deschloro-ARP,ARP(Aripiprazole)在苯环上多了两个氯因而有更多的Cl…C_ali_apo相互作用,与人血白蛋白结合亲和力提高了50倍
如视频1所示,除了Cl…C_ali_apol相互作用之外,Cl还与极性的C-H以及C=O接触,因此很难结合亲和力的提高很难归结为单一的疏水相互作用贡献。
视屏1. PDB 6A7P的两个Cl,不仅有Cl…C_ali_apol相互作用, 而且还有Cl…C=O与Cl..C-H(polar)相互作用
分子对接的打分函数对这种相互作用估计不足。比如用AutoDock Vina 1.20在score only模式下预测化合物Deschloro-ARP与ARP的结合亲和力,计算的Ki分别为-9.831与-10.611kcal/mol。这个微小的差异几乎可以忽略不计,预测值不能解释化合物的结合亲和力差异,因此可视化检查相互作用是非常重要的。
也存在典型的反例:倾向性显著的相互作用越多,并不一定导致活性越强。分析Douglas等人[2]的Dihydroorotate dehydrogenase抑制剂数据(图3)发现,虽然Cl取代化合物(R=Cl,Ki=20nM)具有最多的Cl…C_ali_apol相互作用:与VAL134、VAL143、ILE360的侧链-CH3紧密接触,还与PRO52的-CH2-紧密接触,Cl处于典型的疏水环境中,但是比未取代的化合物(R=H,Ki=1.7nM)的结合亲和力却意外地降低了17倍。
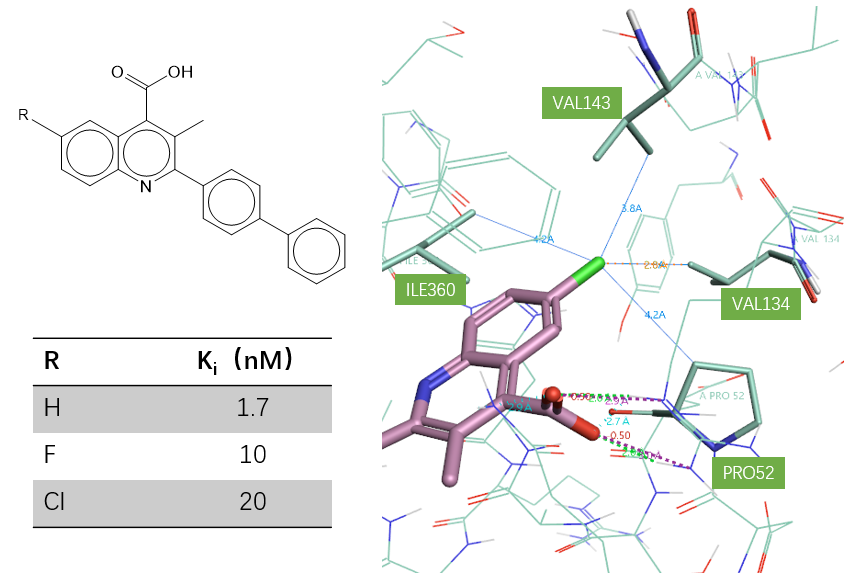
图3. 左边:三个同系物对Dihydroorotate dehydrogenase抑制剂的结构及其Ki;右边:R=Cl化合物预测的结合模式
其中图3的结合模式为预测得到:从PDB 1UUO出发,将对应原子替换为所需的原子、然后对替换后的原子进行力场优化,获得R=H,F,Cl化合物的结合模式用于相互作用分析。
2.2 Cl…C_ali_don相互作用算例
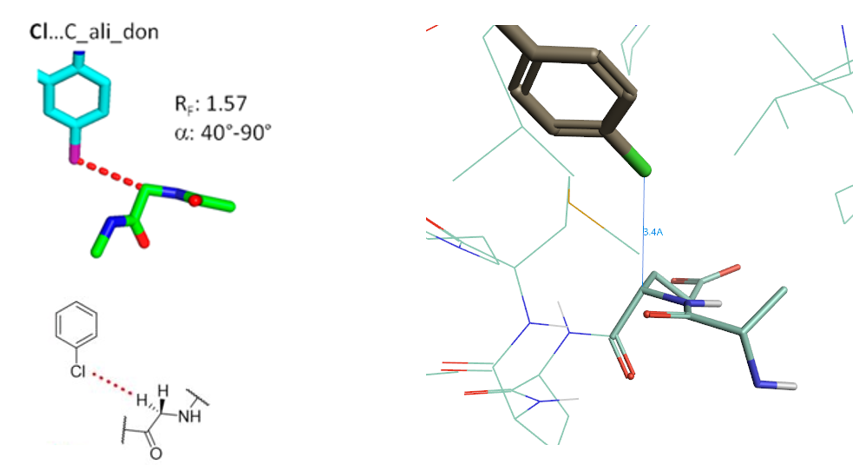
Figure 2. Cl…C_ali_don相互作用, PDB 1UUM
2.2 Cl…C_pi_carbonyl相互作用算例
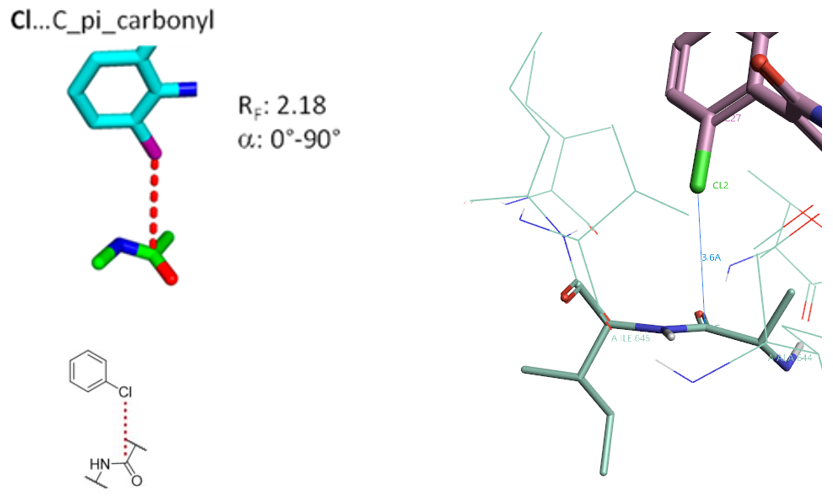
Figure 3. Cl…C_pi_carbonyl相互作用, PDB 5IA3
2.3 Cl…C_pi_phenyl相互作用算例
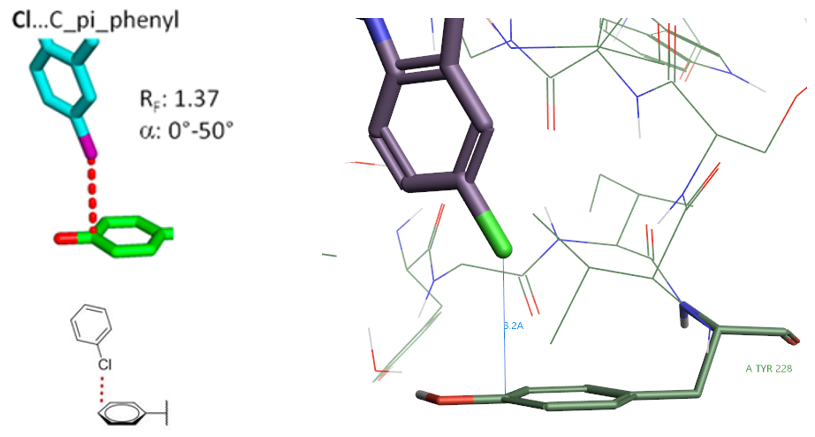
Figure 4. Cl…C_pi_phenyl相互作用, PDB 3Q3K
2.4 Cl…C_pi_pos相互作用算例
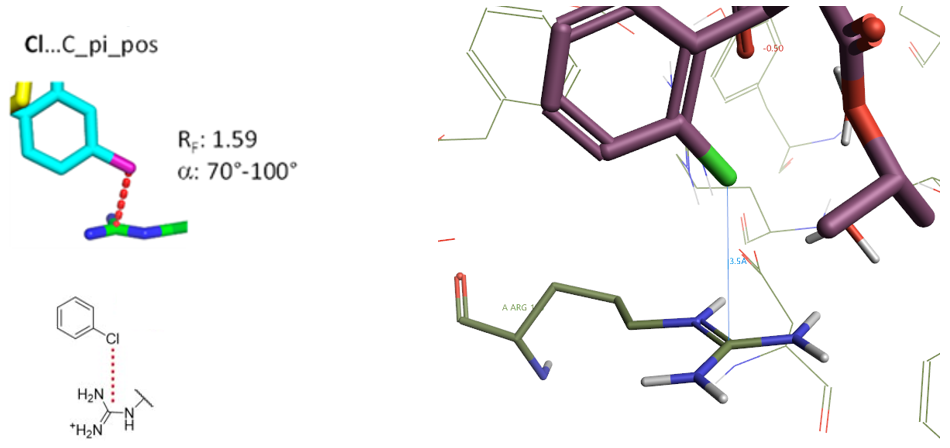
Figure 5. Cl…C_pi_pos相互作用, PDB 2AMV
2.5 Cl…N_pi_don相互作用算例
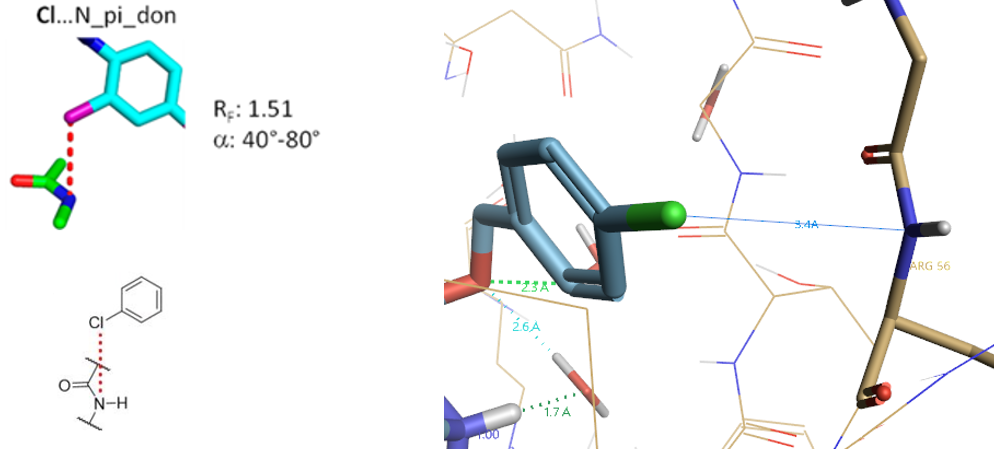
Figure 6. Cl…N_pi_don相互作用, PDB 2C1B
2.6 Cl…O_pi_acc相互作用算例
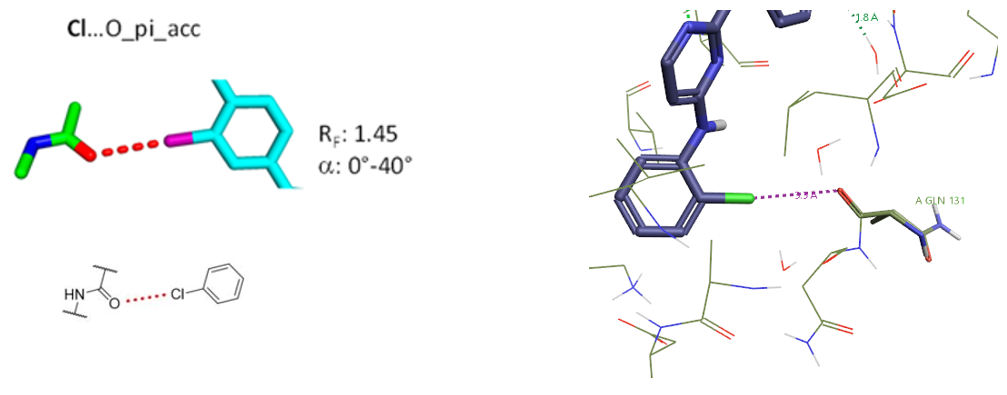
Figure 7. Cl…O_pi_acc相互作用, PDB 3UNK
3. 芳香碳键连的氟与蛋白受体相互作用
3.1 F…C_ali_don相互作用算例
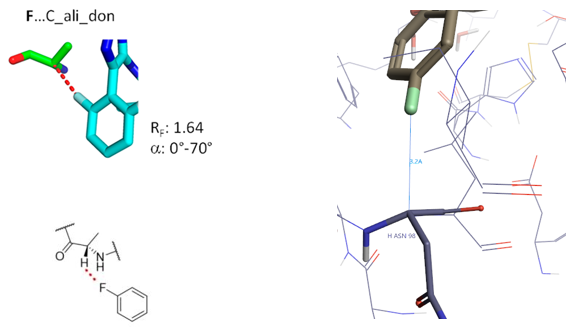
Figure 8. F…C_ali_don相互作用, PDB 1OYT
Olsen等人(2003)对Thrombin进行的氟扫描研究是C-F⋅⋅⋅C=O相互作用的强有力证据。分析抑制剂与Thrombin的复合物结构(PDB 1OYT)可知:苯环4位的氟与ANS98有非常紧密的接触,发生了C-F⋅⋅⋅H-C与C-F⋅⋅⋅C=O相互作用,它们的F⋅⋅⋅C距离分别为3.5与3.2 Å,详见图8与10。
在分子设计中引入这类相互作用,可以显著提高化合物的活性。在Thrombin抑制剂的算例中,使得化合物活性提高5.4倍(图9):F-取代前后的化合物的Ki值分别为310nM与57nM。
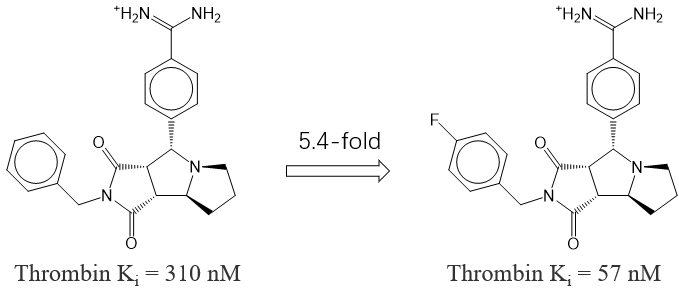
Figure 9.Thrombin抑制剂苯环4-位的氟化使得活性提高了5.4倍
与2.1的算例类似,分子对接方法也不能充分估计C-F⋅⋅⋅C=O相互作用。用AutoDock Vina 1.20在score only模式下预测化合物F原子去除前后的结合亲和力,计算的Ki分别为-10.287与-10.056kcal/mol。F原子的去除与否几乎对Ki的预测值没有影响,预测值不能解释化合物的结合亲和力差异。再一次,对这相互作用的可视化检查不能用分子对接计算所代替。
3.2 F…C_pi_carbonyl相互作用算例
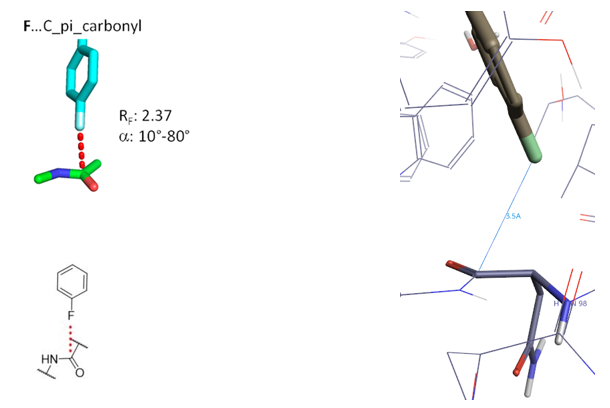
Figure 9. F…C_pi_carbonyl相互作用, PDB 1OYT
3.3 F…C_pi_pos相互作用算例
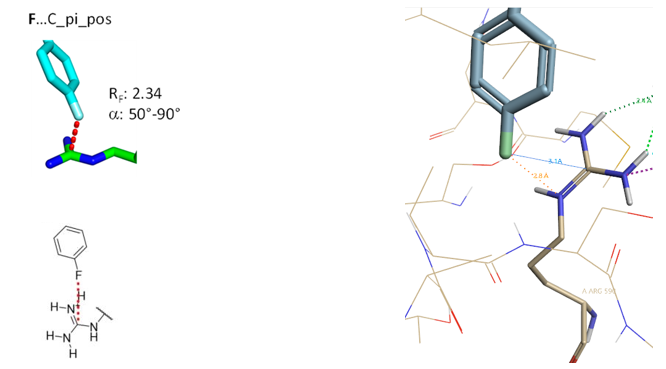
Figure 10. F…C_pi_pos相互作用, PDB 1HWK
3.4 F…N_pi_don相互作用算例
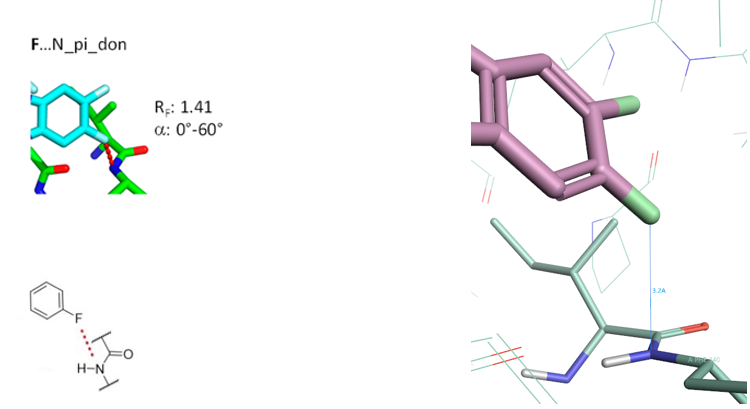
Figure 11. F…N_pi_don相互作用, PDB 5Q14
4. 腈基与蛋白受体相互作用
4.1 CN…C_ali_don相互作用算例

Figure 12. CN…C_ali_don相互作用, PDB 2I0Y
4.2 CN…N_pi_don相互作用算例
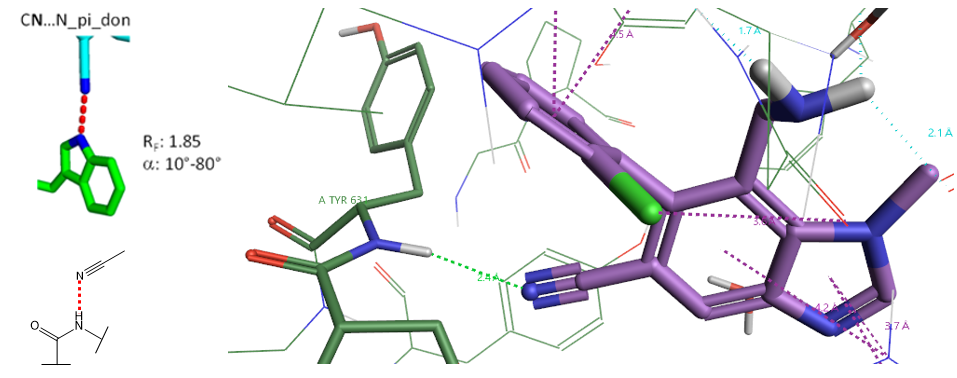
Figure 13. CN…N_pi_don相互作用, PDB 3CCC
4.3 CN…N_pi_don_pos相互作用算例
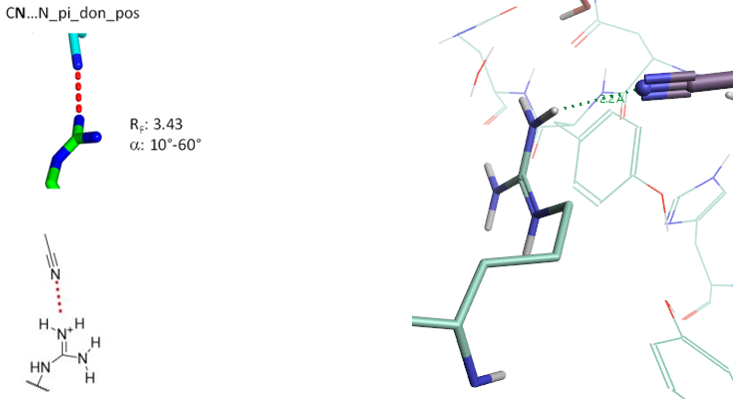
Figure 14. CN…N_pi_don_pos相互作用, PDB 1LD7
5. 磺酰基氧与蛋白受体相互作用
5.1 SO2…N_don_pos相互作用算例
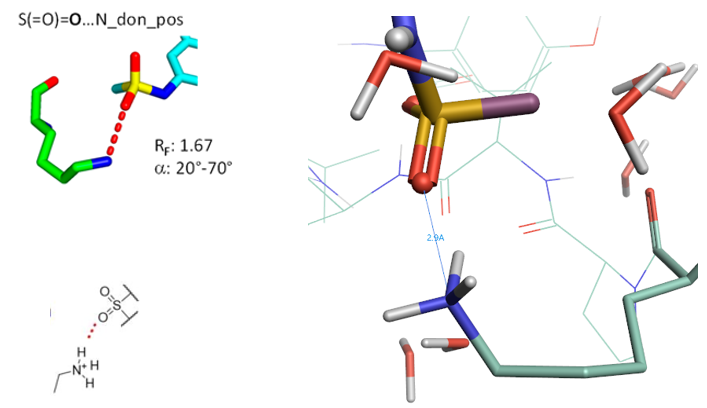
Figure 15. SO2…N_don_pos相互作用, PDB 5UER
5.2 SO2…C_ali_don相互作用算例
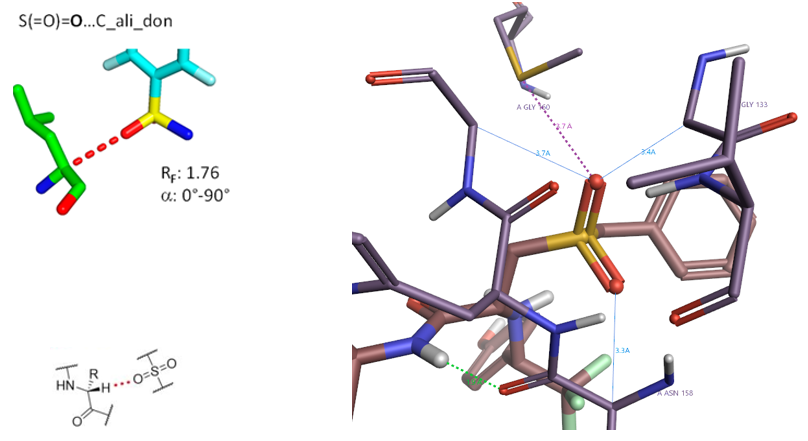
Figure 16. SO2…C_ali_don相互作用, PDB 2FRA
5.3 SO2…C_pi_don相互作用算例
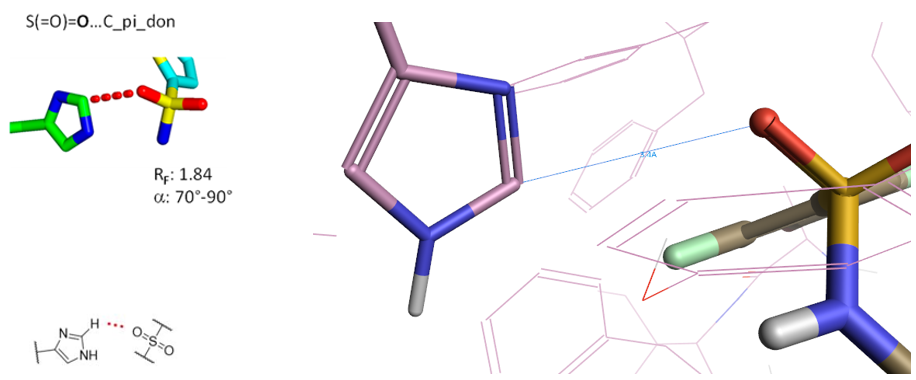
Figure 17. SO2…C_pi_don相互作用, PDB 5TTD
5.4 SO2…N_pi_don相互作用算例
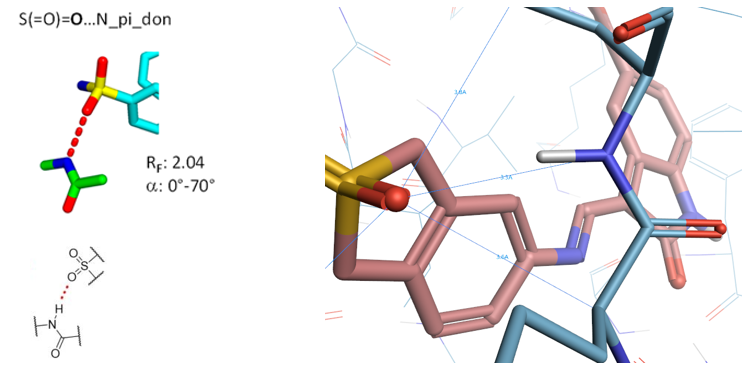
Figure 18. SO2…N_pi_don相互作用, PDB 1KE7
5.5 SO2…N_pi_don_pos相互作用算例
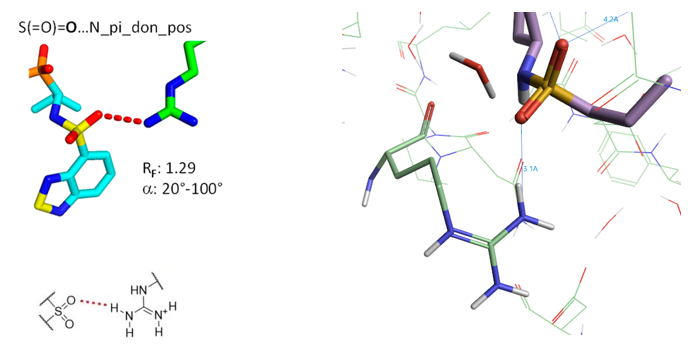
Figure 19. SO2…N_pi_don_pos相互作用, PDB 6BBV
6. 芳香硫原子与蛋白受体相互作用
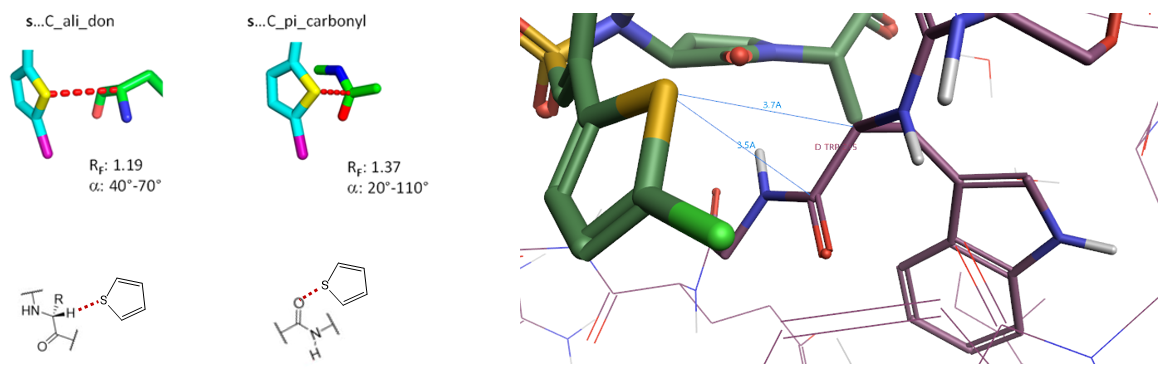
Figure 20. S…C_pi_carbonyl以及S…C_ali_don相互作用, PDB 2JH0
文献
- Sakurama, K.; Nishi, K.; Imoto, S.; Hashimoto, M.; Komatsu, T.; Morita, Y.; Taguchi, K.; Otagiri, M.; Yamasaki, K. Further Evidence Regarding the Important Role of Chlorine Atoms of Aripiprazole on Binding to the Site II Area of Human Albumin. J. Pharm. Sci. 2019, 108 (5), 1890–1895. https://doi.org/10.1016/j.xphs.2018.11.045.
- Batt, D. G.; Copeland, R. A.; Dowling, R. L.; Gardner, T. L.; Jones, E. A.; Orwat, M. J.; Pinto, D. J.; Pitts, W. J.; Magolda, R. L.; Jaffee, B. D. Immunosuppressive Structure-Activity Relationships of Brequinar and Related Cinchoninic Acid Derivatives. Bioorganic Med. Chem. Lett. 1995, 5 (14), 1549–1554. https://doi.org/10.1016/0960-894X(95)00252-O.
- Olsen, J. A.; Banner, D. W.; Seiler, P.; Obst Sander, U.; D’Arcy, A.; Stihle, M.; Müller, K.; Diederich, F. A Fluorine Scan of Thrombin Inhibitors to Map the Fluorophilicity/Fluorophobicity of an Enzyme Active Site: Evidence for C-F⋅⋅⋅C=O Interactions. Angew. Chemie Int. Ed. 2003, 42 (22), 2507–2511. https://doi.org/10.1002/anie.200351268
索要Flare Viewer的Story Board
首先您需要下载一个免费的Flare Viewer才能浏览已经准备好的相互作用分析。下载Story board,请联系我们。



