在基于结构的设计中用Flare Viewer来提高视觉分析的效率
摘要:很多重要的非经典相互作用不能被传统的分子对接、分子力场等计算方法正确评估,因此视觉分析在基于结构的药物设计中是非常必要且重要的。本文介绍了Flare的Pick group的主要功能,并演示了如何用Flare的Pick group在基于结构设计中提高视觉分析的效率。
问题
之前我们讨论过人工视觉分析在分子对接后处理中的重要性[1],还分享了Kuhn等人[2]总结的各种非经典蛋白-配体相互作用模式的重要性。我们列举了多个活性悬崖的例子,其中配体化学结构的微小差异可导致配体-蛋白在相互作用上巨大的差异。这种活性差异经常难以用分子对接等计算方法进行预测,比如在可视化算例2.1与3.1中[3],发现分子对接软件AutoDock Vina(Version 1.2)的打分值就不能区分化合物在活性上的较大差异,这进一步说明基于经验(知识)的视觉分析不能完全被分子对接计算所代替。
虽然Kuhn等人对非经典相互作用的重要性分析(即基于统计学的相互作用分析,RF interaction)给出了很多启示,但是面对复杂的蛋白-配体复合物结构,单纯通过肉眼考察此类相互作用是非常耗时且效率低下的。Kuhn视线法的第一步是找出配体上一个感兴趣原子一定距离被的蛋白原子,虽然已经有Chimera、Pymol等图形化软件可以做到,但我认为Flare Viewer更加便利。因此,本文的主要目的是演示如何用Flare Viewer的Pick group来提高视觉分析的效率。
Pick Group的描述与使用
用Pick group按钮可以完成全面、综合的原子挑拣任务,图1总结了其功能。
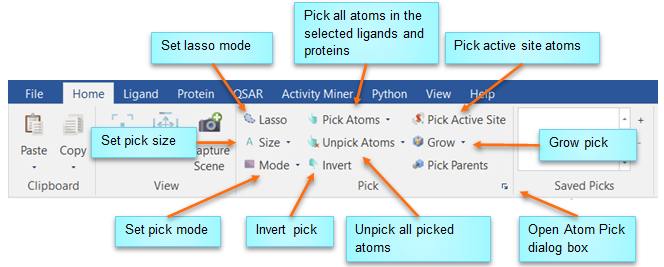
图1. Pick group按钮的主要功能汇总
主要的功能简述如下:
Size
设置点击选中对象的类型:
- Atom: 单击选中一个原子
- Residue: 单击选中一个氨基酸残基
- Fragment: 单击选中整个片段
- Chain: 单击选中整条链pick entire chains
- Molecule: 单击选中整个分子
Mode
设置点击挑选其它对象时对当前已被选中对象的影响方式:
- Replace: 当前的单击选中替换掉原先的
- Add: 新的单击选中与之前选中累加起来
- Subtract: 从之前的选中对象中扣除掉现在选中的对象
- Toggle: 根据新的单击选择进行反向选择
- Intersect: 新、旧选中对象的交叉对象
Pick Atoms
- Pick Atoms: 选中所有可视化呈现的原子(配体以及蛋白) (快捷键: Ctrl + A)
- Pick Ligands: 选中可视化呈现的配体原子
- Pick Proteins: 选中可视化呈现的蛋白原子
Unpick Atoms
Invert
反向选中当前的原子(对象)。
Pick Active Site
在蛋白或配体表单里至少选中一个配体或蛋白,或者视窗内至少一个配体或蛋白可见,这时点击Pick Active Site按钮,将以配体定义蛋白结合位点的方式将距离配体一定距离内的蛋白残基选中。
Grow
在当前选中原子的基础上在指定的半径内扩大选中范围。
Pick Parents
在当前选中对象的基础上,拓展至对象所在的“父”结构,比如:
- 如果你有个配体原子被选中了,Pick Parents将选中拓展至该原子所在的整个配体。
- 如果你有个蛋白残基原子被选中,Pick Parents将选中拓展至该蛋白原子所在的整个残基。
- 如果你有整个残基被选中,Pick Parents将选中拓展至该残基所在整个链(Chain)。
- 如果你有整个链被选中,Pick Parents将选中拓展至包含该链的整个蛋白。
高级的操作
点击Pick group右下角的小箭头,有更多高级的操作。
用Python进行自动操作
如果需要自动地从PDB下载蛋白结构、准备结构、选择原子、居中放大等操作,需要使用Flare的Python来实现。比如,Paolo[4]就演示了如何用Python来展示结合到同一蛋白结合位点的两个配体的公共接触蛋白原子。
用Pick group快速找到与特定配体原子接触的蛋白原子
以PDB 6A7P为例,下载蛋白结构并准备好之后,如图2所示,先选中一个氯原子(图2操作1),然后点击grow(图2操作2),则选中了与该氯原子4Å距离内的所有原子(图2阴影高亮的原子),包括配体原子。接着在unpick atom下撤销对配体的选中,则仅保留下蛋白原子被选中。这些原子正式接下来要进行重点分析的对象。
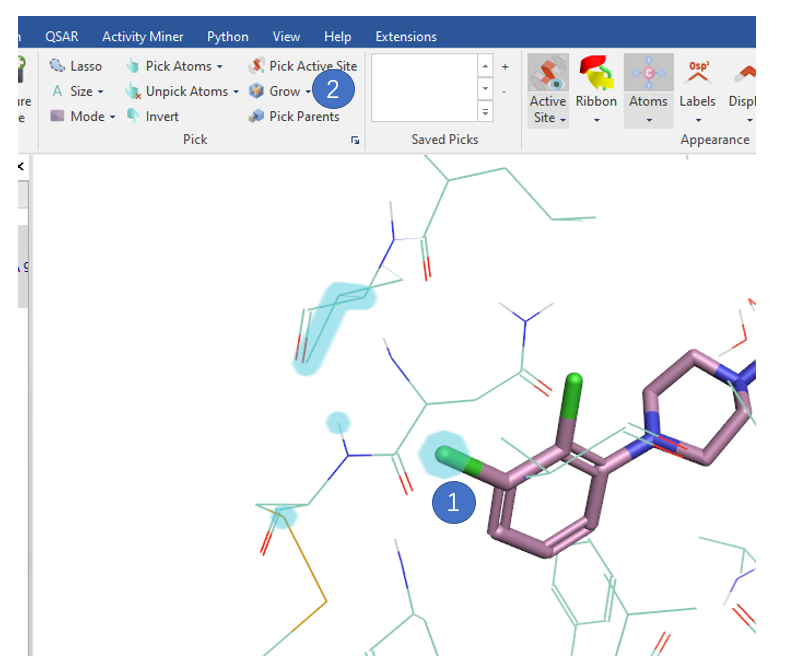
图2. 选中与氯原子距离4Å的蛋白原子(PDB 6A7P)
比如,接下来我们可以测量氯原子与蛋白原子间的距离以及相关的角度等等,如图3所示。
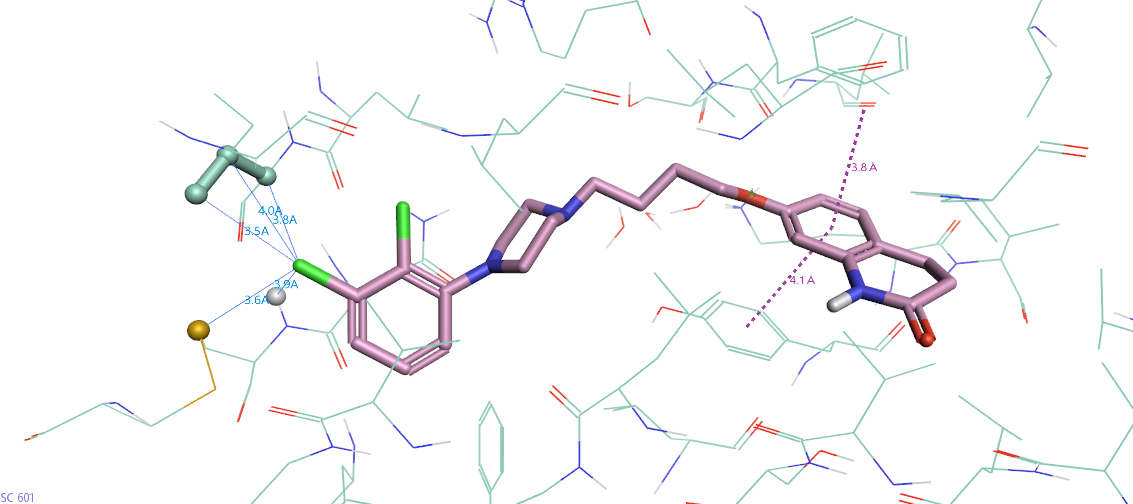
图3. 氯原子与接触蛋白原子间的距离
下载免费的Flare Viewer
注册下载软件:https://www.cresset-group.com/software/download-flare-viewer
视频:使用storyboard
视频:用Flare Viewer分析蛋白-配体相互作用
文献
- 李凯丽,张丽君,柏川(2021-06-04). 墨灵格的博客. 基于结构药物发现中的决策:对接结果的手工检查
- 肖高铿(2021-05-01).墨灵格的博客. 数据库挖掘的启示:不同寻常的蛋白-配体相互作用有多重要?
- 肖高铿(2021-05-09). 墨灵格的博客. 不同寻常的蛋白配体相互作用-PDB可视化算例
- Paolo Tosco(2018-09-27).Using Python in Flare to find common contacts. Retrieved from https://www.cresset-group.com/about/news/python-flare-common-contacts



