摘要:Cresset技术的核心在于其专有的XED力场,该力场通过扩展电子分布(XED)来更准确地模拟分子内的静电和轨道相互作用,无需过多参数。XED力场可简化为“场点”,用于描述分子,并应用于多种药物设计工具。Cresset利用场点技术开发了包括Spark、Blaze和3D Field QSAR等工具。Spark用于骨架跃迁和生物等排体替换,例如在PROTAC AK-2292连接臂设计中,成功生成了活性连接臂,并能识别出活性更高的结构。Blaze是一种3D虚拟筛选技术,通过识别相似的静电和形状性质,发现新的苗头化合物。3D Field QSAR则用于分析SARS-CoV-2 Mpro抑制剂,识别关键活性区域,并构建预测模型,其性能优于传统的2D方法。此外,Cresset的静电互补性 (EC) 技术通过比较蛋白质和配体的静电场,突出吸引和排斥区域,为化合物优先级排序提供依据,并可视化蛋白质-配体间的静电相互作用。这些技术共同为药物发现提供了一种基于分子静电和形状的有效方法。
Ryan Hubball. et al. (2025). https://www.cresset-group.com
Cresset技术简介
Cresset核心技术利用了我们专有的XED力场的强大功能,该力场最初由Cresset创始人Andy Vinter开发,旨在更好地模拟极化率1。
与以原子为中心的电荷力场不同,扩展电子分布(XED)被添加到原子上,从而能够模拟分子内的静电和轨道相互作用,而无需引入更多的参数。
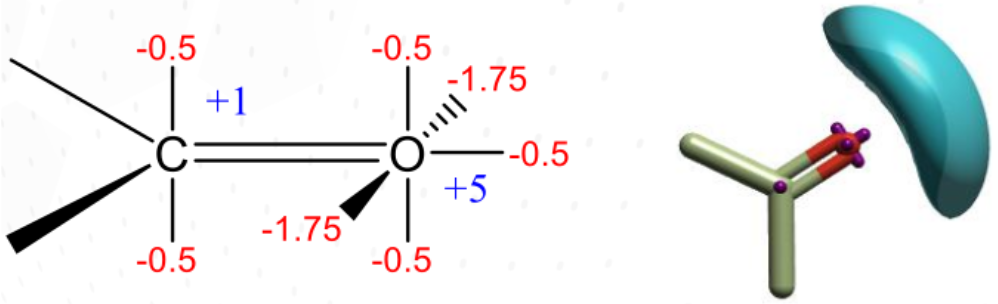
图1. 左图:丙酮,显示了XED值。右图:以3D形式显示的丙酮,并在原子上添加了XED。
XED力场可以表示分子、分子间相互作用势或“分子场”。这些都可以简化为它们的极大值和极小值,即“场点”,并用作描述符2。
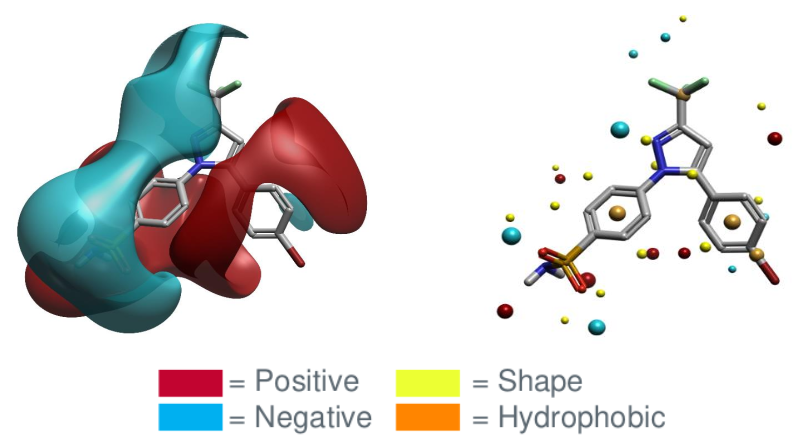
图2. 左图:3D分子静电相互作用势。右图:场点。
场点用于许多Cresset工具中,以辅助基于结构和基于配体的药物设计。
接下来我们会介绍Cresset科学家如何利用基于场点的分子描述方法来帮助药物发现。
Spark骨架跃迁
Spark™是一种骨架跃迁和生物等排体替换工具,它能够利用输入的片段库替换参比分子中选定的部分结构。随后,将新设计的分子与起始结构进行形状和场点相似性比较。
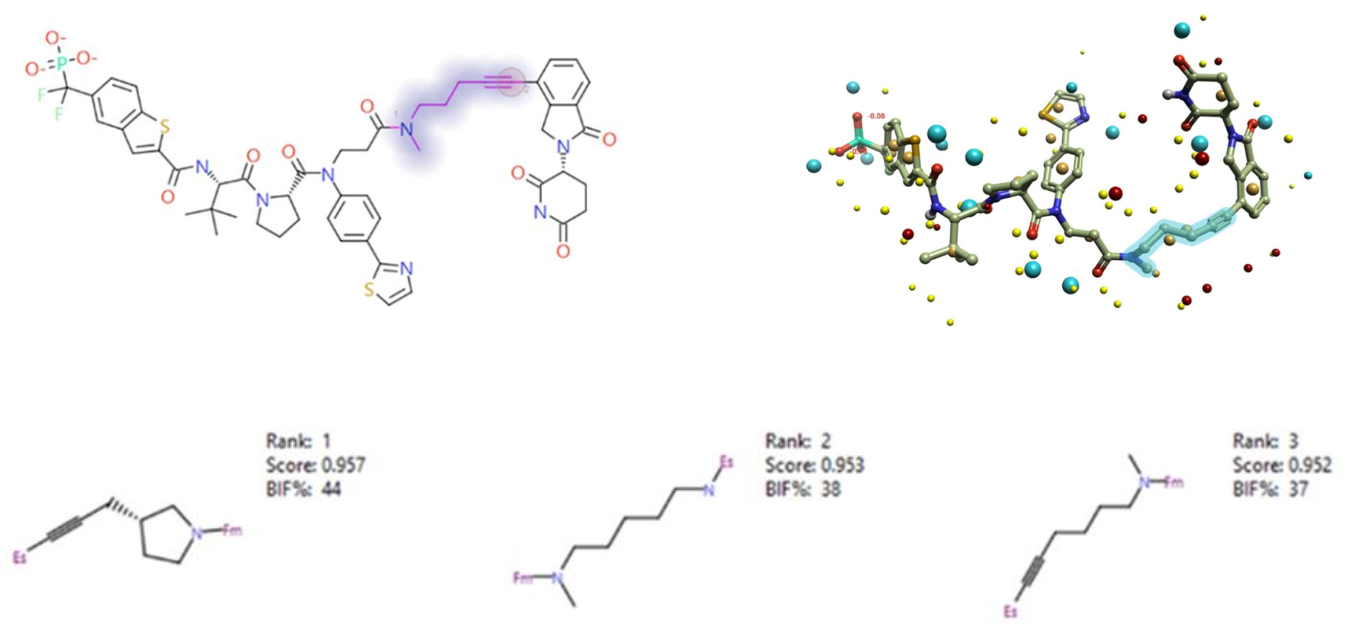
图3. 左上:Spark的输入选择界面. 右上:高亮显示了连接臂的分子及其3D场点。图下:打分最高的8个Spark结果。
此类方法可用于建议新型连接臂片段,用于构建异双功能降解分子:AK-2292由STAT5结合基团、CRBN结合基团和连接它们的连接臂组成。该分子被报道为一种有效的异双功能降解分子,具有强大的抗肿瘤活性3。
在骨架跃迁实验中搜索新的连接臂,生成了该研究报告的十个连接臂中的八个。其中包含第三个活性最强的降解分子,在Spark产生的1000个结果中排名第七。
该研究中未被Spark发现的唯一连接臂是两个环状酰胺,它们表现出非常低的降解活性。
这些结果表明Spark能够生成新颖的结构,同时保持输入结构期望的形状和静电性质。
BLAZE虚拟筛选
Blaze™是一种3D虚拟筛选技术,它利用场点识别与已知生物活性分子具有相似静电和形状性质的分子,从而为发现额外的苗头化合物提供新的思路。该方法被用于以DG025过渡态抑制剂4作为已知生物活性的参比分子,筛选跨膜IRAP蛋白的新苗头化合物。尽管起始的查询分子为伪肽,Blaze仍能够识别满足活性位点静电和形状要求的活性小分子。
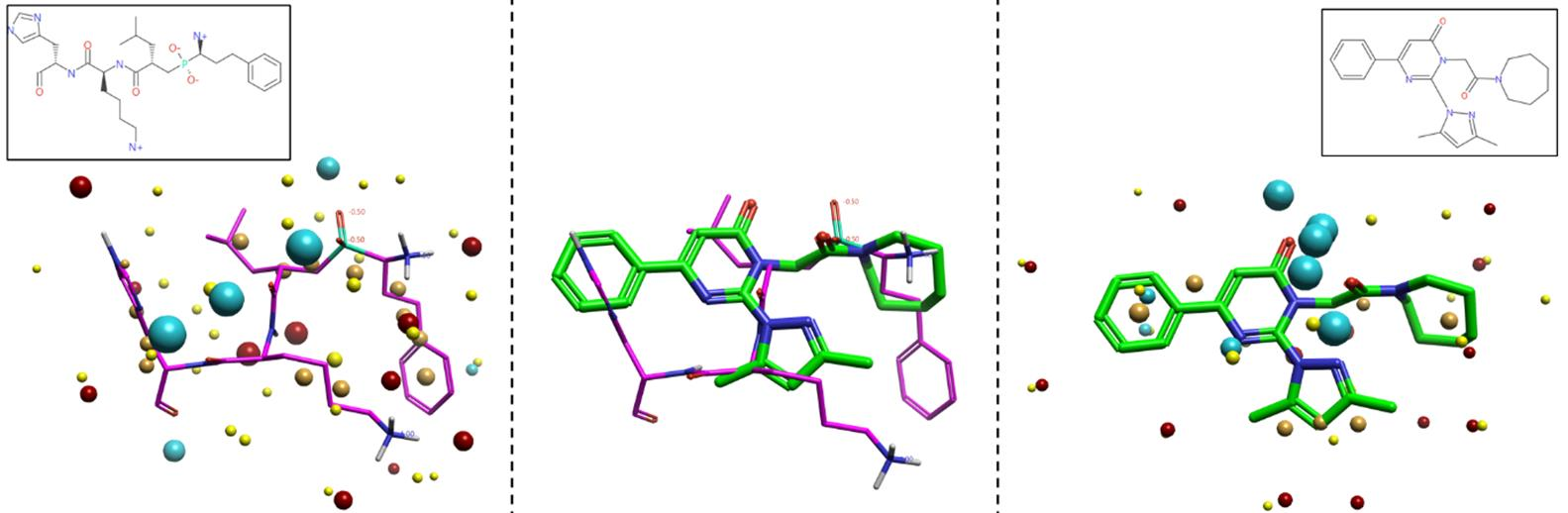
图4. 左:伪肽输入分子及其场点;中:分子叠合;右:推荐的苗头化合物及其场点。
3D-Field QSAR:阐明抑制剂活性
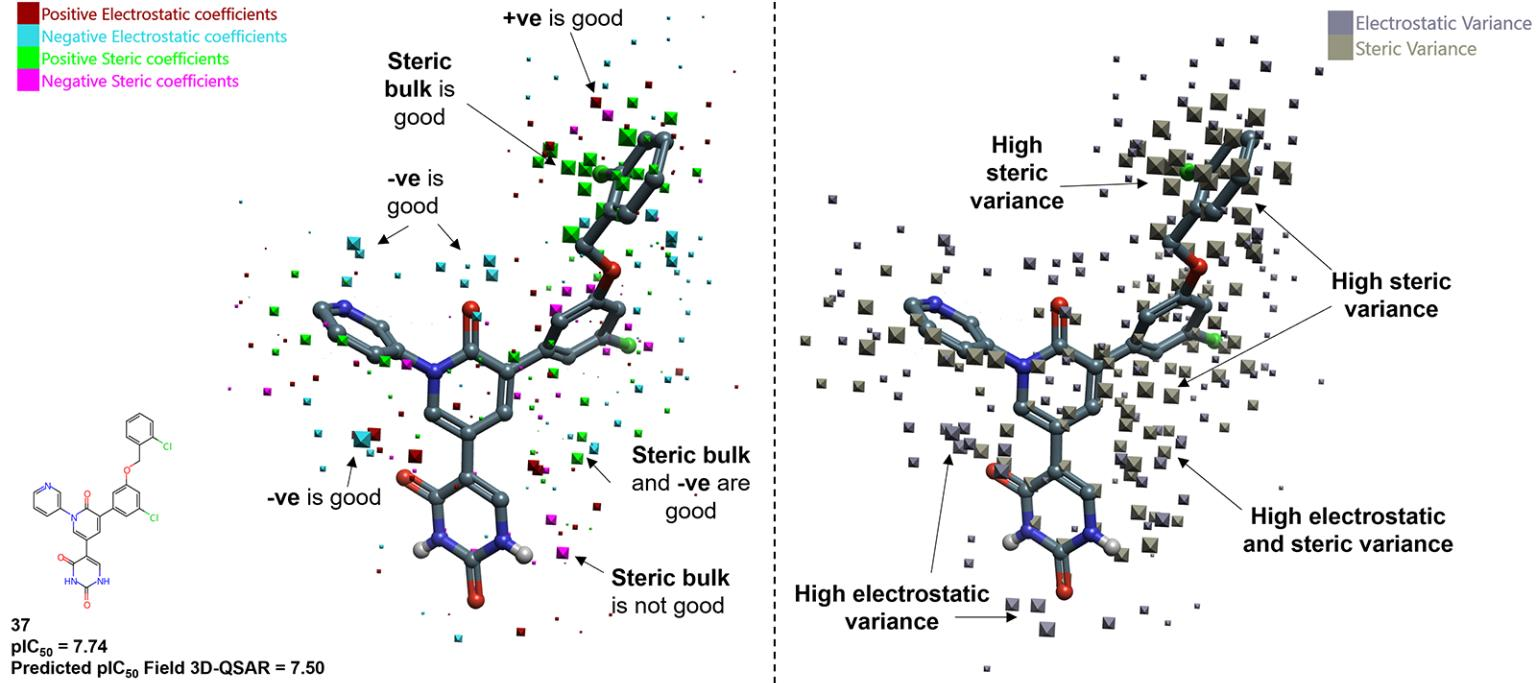
图5. 数据集中活性最强的分子及其3D Field QSAR信息。左侧,高亮显示了正负静电和立体系数的空间分布位置。右侧,显示了3D Field QSAR中静电和立体系数的变异区域。
3D Field QSAR利用场点识别对生物活性重要的特征,并将这些相关性特征关联起来形成假设。已对一组SARS-CoV-2 Mpro抑制剂进行了研究,以构建预测模型。
将76种不同化学类型的非共价抑制剂叠合到4个共晶结构组成的参比化合物上5。通过使用3D Field QSAR而非2D方法,可以识别潜在的关键区域,用于进一步建模,从而提供优化机会。
该案例研究表明,2D QSAR模型和3D QSAR模型之间存在一致性,其中3D Field QSAR模型的性能优于机器学习方法。
通过3D Field QSAR实现的可视化,利用静电和空间系数来阐明活性,并说明如何指导新型治疗药的设计,并用来进行优先级排序。
静电互补性(Electrostatic Complementarity™)
静电互补性 (Electrostatic Complementarity™,EC) 比较蛋白质和配体的静电场,以突出吸引和排斥区域。这既产生了一个打分,在研究中用作优先级排序的指标,也可以在Flare™中显示为分子表面,以显示蛋白质和配体之间静电有利(绿色)或存在静电冲突(红色)的区域。
在最近的一项研究中,利用Spark骨架跃迁技术为METTL3抑制剂生成了新的设计思路6。为了对生成的设计进行优先级排序,使用了静电互补性(EC)以及其他指标。
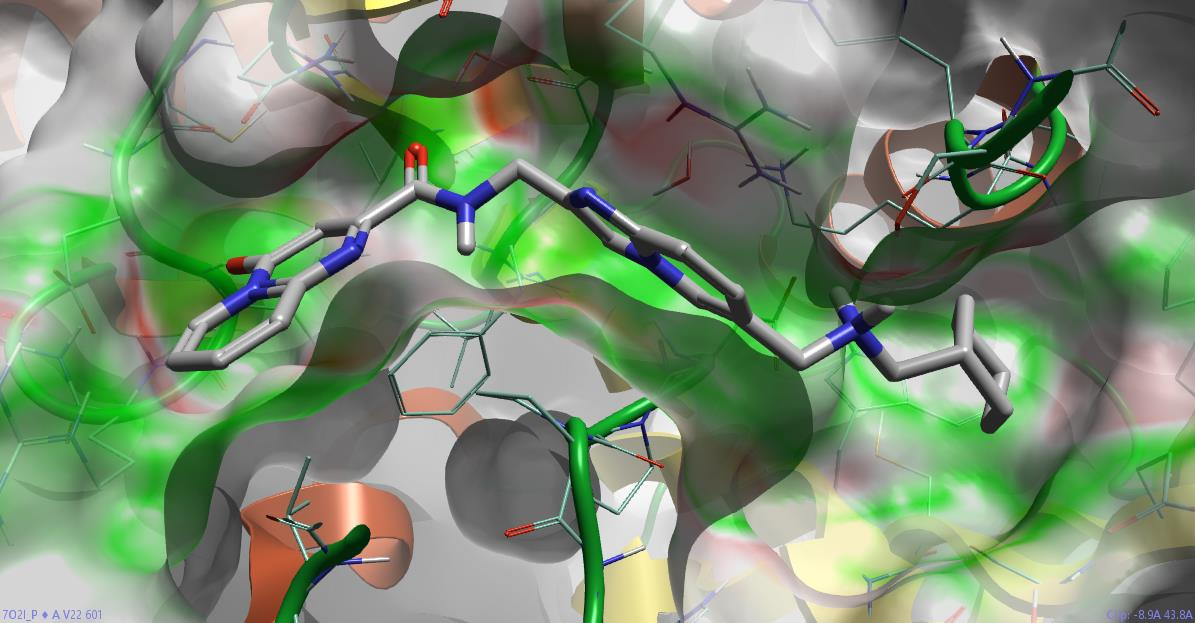
图6. METTL3与配体共晶结构(PDB 7O2I)结合位点周围的静电互补表面
联系我们
Blaze, Spark, Flare是Cresset三个旗舰药物设计软件。可以通过软件安装或技术服务的方式使用这些软件。请现在就联系我们安排试用,我们的专业团队随时准备通过安装和设置为您提供支持,而我们全面的教程库——涵盖从常见工作流程到高级方法和功能的所有内容将帮助您开始使用。我们在这里帮助您更快地实现目标,让您设计出感兴趣的分子。
电邮:info@molcalx.com
电话:020 – 38261356
参考文献
- Vinter, J.G. (1994) “Extended electron distributions applied to the molecular mechanics of some intermolecular interactions,” Journal of Computer-Aided Molecular Design, 8(6), pp. 653–668. Available at: https://doi.org/10.1007/BF00124013.
- Cheeseright, T. et al. (2006) “Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation,” Journal of Chemical Information and Modeling, 46(2), pp. 665–676. Available at: https://doi.org/10.1021/ci050357s.
- Kaneshige, A. et al. (2023) “Discovery of a Potent and Selective STAT5 PROTAC Degrader with Strong Antitumor Activity In Vivo in Acute Myeloid Leukemia,” Journal of Medicinal Chemistry, 66(4). Available at: https://doi.org/10.1021/acs.jmedchem.2c01665.
- Mpakali, A. et al. (2024) “Stabilization of the open conformation οf insulin‐regulated aminopeptidase by a novel substrate‐selective small‐molecule inhibitor,” Protein Science, 33(9), p. e5151. Available at: https://doi.org/10.1002/pro.5151.
- https://cresset-group.com/discovery/specific/prioritization-
new-molecule-design-qsar-models - Dutheuil, G. et al. (2025) “Discovery, Optimization, and Preclinical Pharmacology of EP652, a METTL3 Inhibitor with Efficacy in Liquid and Solid Tumor Models,” Journal of Medicinal Chemistry, 68(3), pp. 2981–3003. Available at: https://doi.org/10.1021/acs.jmedchem.4c02225.



