摘要:本教程展示了如何用Ligandscout生成配体-蛋白相互作用2D图,并用鼠标编辑氨基酸的位置,保存为图片,调整图片的分辨率。
一. 背景
常有人问:如何用2D图展示配体-受体的相互作用?这里,我推荐用Liganscout来绘制相互作用图2D图。
二. 操作步骤
根据不同的情况:1)从PDB数据库获取复合物结构;2)配体分子的pose是用分子对接、分子叠合等获得的结果。
1. 从PDB数据库获得复合物结构
- 生成基于结构的药效团
- 编辑、生成特定分辨率的2D相互作用图
- 保存2D相互作用图
同基于结构的药效团一样,直接输入PDB code,准备基于结构的药效团,会自动的生成相互作用2D图,具体操作见:
Ligandscout基于结构的药效团建模:http://blog.molcalx.com.cn/2016/09/03/ligandscout-tutorial-structure-based.html
Ligandscout的2D相互作用图是可以用鼠标编辑的:在2D图上,用右键按住一个氨基酸,可以移动该氨基酸残基。你可以调整残基的布局直到满意为止。
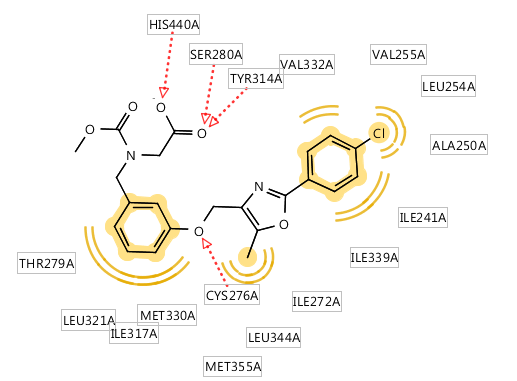
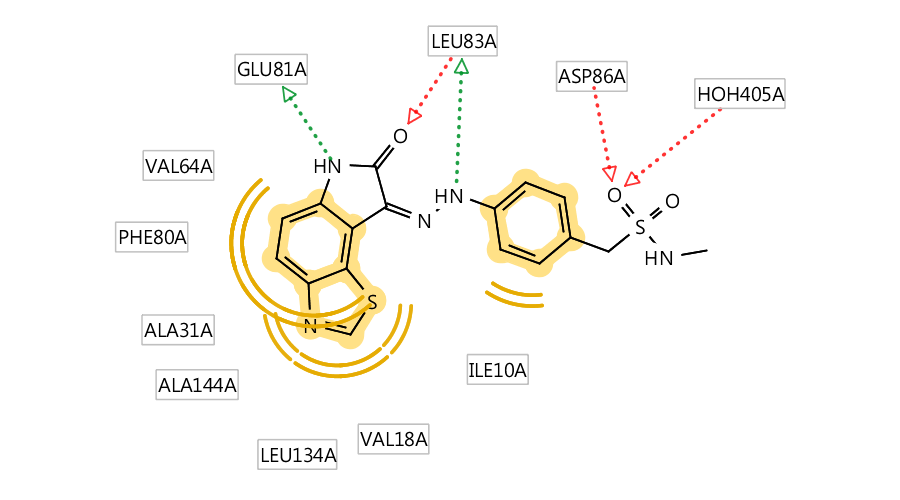
图1. Ligandscout生成的2D相互作用图,用鼠标右键按住残基,可以调整位置
菜单:File>Save as file,设置文件类型为Image File(见图2),并选择一个文件类型(见图3),比如PNG(见图4)。
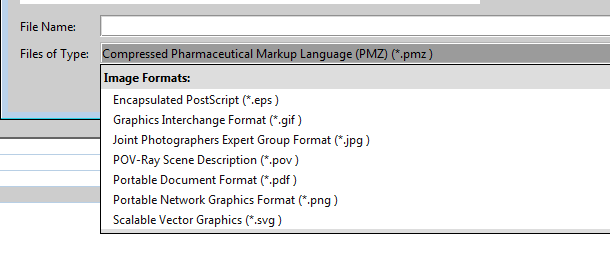
图2. 选择文件类型为Image File
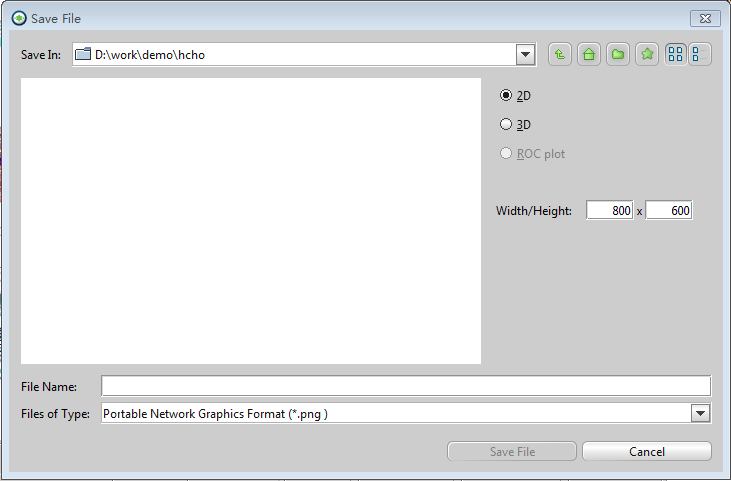
图3. 选择图像格式,比如PNG,并设定图像的分辨率
2. 用分子对接等获得配体的pose
操作步骤如下:
- 下载PDB,定义结合位点
- 插入pose
- 生成、保存2D-相互作用图
同上一个方法,总的来说,需要定义结合位点。
File | Insert 插入对接生成的pose,并生成基于结构的药效团
同上一种情况的操作。
三. 分享几张2D图

图4. PDB code 1KE6
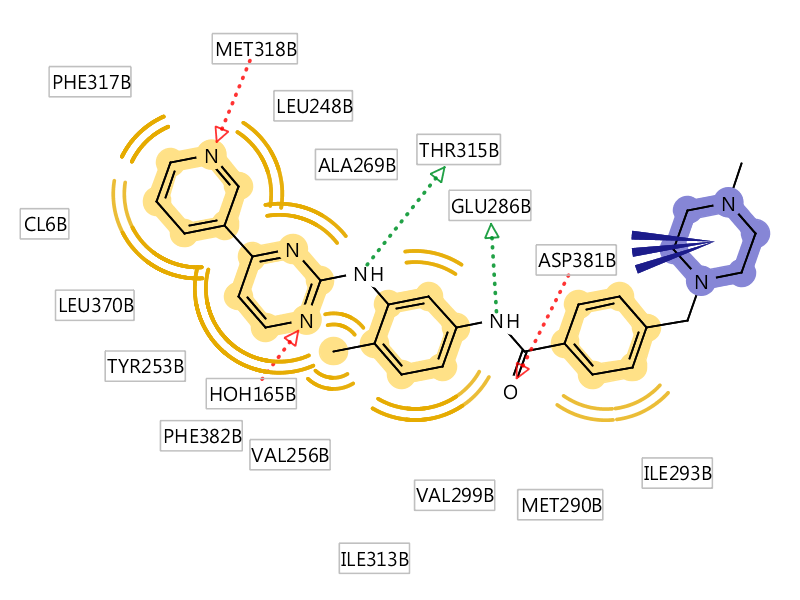
图5. PDB code 1IEP



