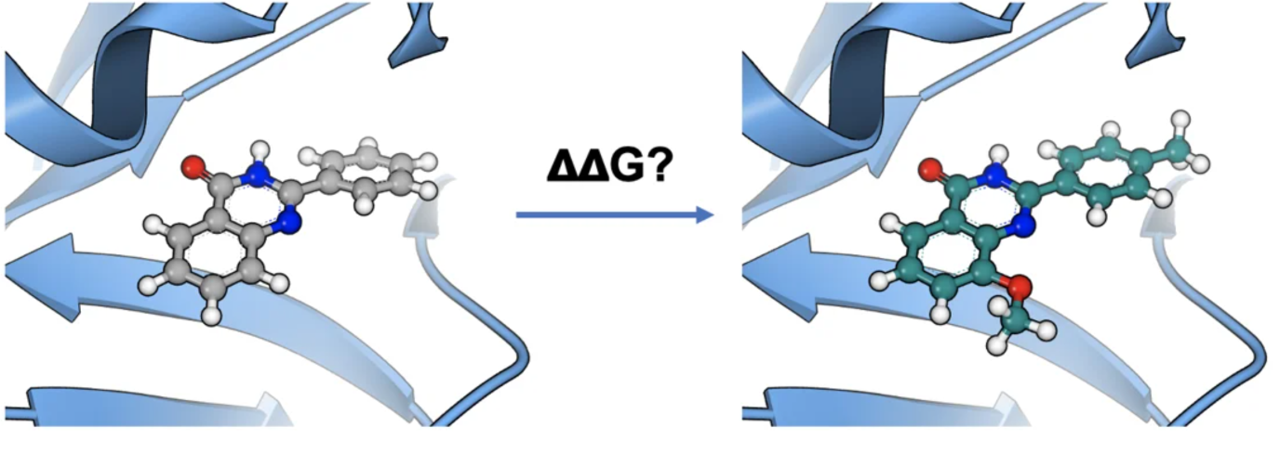
摘要:OE Affinity是OpenEye云计算平台ORION的一个模块,OE Affinity NES是为蛋白质-配体复合物的结合自由能计算的高效解决方案,通过其非平衡切换(Non-Equilibrium Switching)方法,提供快速且经济高效的相对结合自由能计算。
简介
OpenEye为蛋白质-配体复合物的结合自由能估算提供了高效且有效的解决方案——OE Affinity NES,其采用由马普研究所(Max Planck Institute)开发的非平衡切换(Non-Equilibrium Switching,NES)技术进行结合自由能计算。与需要更接近平衡态采样的自由能微扰(FEP)或λ动力学等传统平衡方法相比,NES方法在速度和成本效益方面具有显著优势。
OpenEye进一步优化了NES技术,使其在Orion云平台上实现自动化计算。即使面对大量配体,该平台也能在数小时内完成计算,使OE Affinity NES特别适用于迭代式先导化合物优化流程。
我们的自动化解决方案在确保速度、性能和精度的同时,可显著节省时间和成本。
一位大型制药公司的CADD科学家评论到:
“对于40个配体,传统FEP+方法需要24-36小时,而基于Orion平台的NES方法仅需2-3小时。“
OE Affinity NES的特点
- 已获验证——基于马·普研究所DeGroot实验室发表的研究成果
- 准确——已在内部和外部数据集上验证
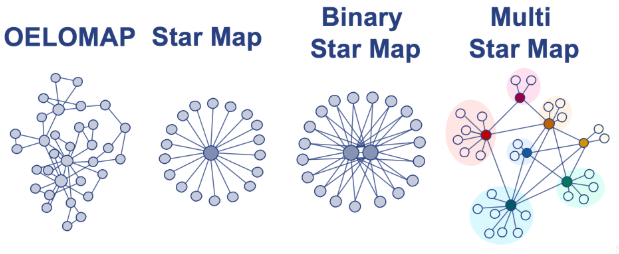
- 灵活——用于连接配体的边映射器选项包括OELOMAP、Star Map、Binary Star Map和Multi-Star Map
- 易于使用——在熟悉的Orion网络环境中快速启动和运行
- 自动化——系统设置、映射、端点平衡和控制的简单流程
- 性能——极快且高度并行化;可在数小时内运行整个配体数据集
- 控制——高度可定制,适用于初学者使用和专家控制
- 集成——可轻松与其他配体和结构方法结合使用
根植于科学,优化性能,为您而制
依托前沿科学与技术,OE Affinity通过Orion云建模平台将强大工具直接交付至您的网络浏览器,从而简化药物发现流程。无需安装软件,您只需一个网络浏览器即可登录并立即开始!
我们的自动化解决方案专为速度、性能和准确性而设计,可帮助您节省时间与成本。
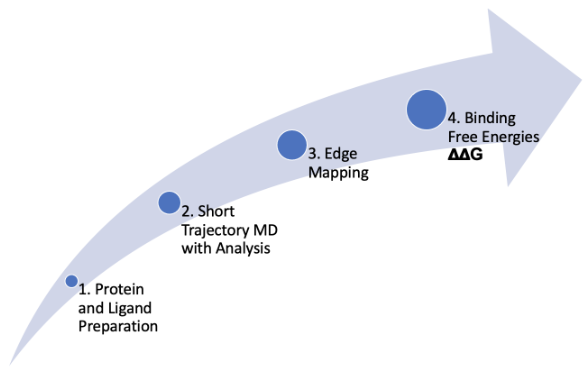
OpenEye提供全自动工作流程,可系统化优化您的计算流程,从蛋白-配体预处理到自由能分析均实现无缝衔接。该工作流程支持可定制化设计,为专业研究人员提供更精细的参数调控与操作灵活性。
便捷、高效、自动化映射
OE Affinity使得用户可以便捷地创建映射器,并提供多种选项灵活选择。在结合自由能计算中用于连接配体的自动化边映射器包括:OELOMAP、Star Map、Binary Star Map及Multi-Star Map。
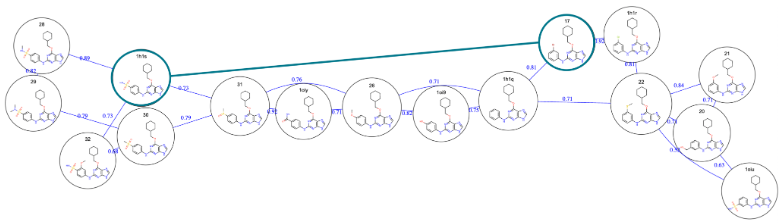
OE Affinity Mapper通过选择高效的”连接路径”(ligand transformations)优化非平衡切换(Non-Equilibrium Switching, NES)过程。这些配体转换路径可实现整个配体集合的快速结合自由能计算。连接路径的选择方法结合使用最大公共子结构(Maximum Common Substructure, MCS)相似性分析与OpenEye的ROCS(形状与色散)打分体系,从而显著提升计算精度。
交互式边映射器提供灵活的操作
通过交互式边映射器(interactive Edge Mapper),用户可无缝添加或移除连接路径,并以选定配体为中心枢纽生成新的星型图(Star Map)。更新后的边映射可保存为数据集用于下游自由能计算,使得用户能够通过人工专业知识优化边的网络构建。
通过浏览器直接操作的拖放功能,可便捷地自定义连接路径
针对具有柔性结合位点的蛋白质
为提升对柔性结合位点蛋白质的配体结合亲和力预测能力,建议协同应用诱导契合结合模式(Induced-Fit Posing)与OE相对结合亲和力分析(OE Affinity Relative Binding)。这两种工具的联合使用可更精确地模拟配体-蛋白质相互作用,显著提高预测准确性。
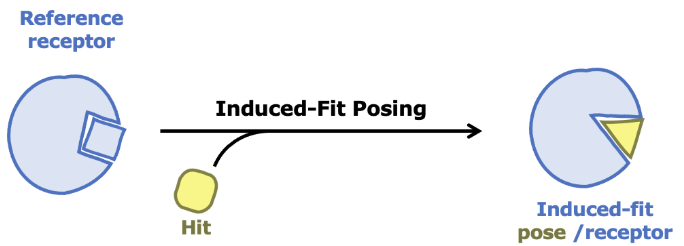
OpenEye诱导契合结合模式的示意图说明
FAQ
OE Affinity打分的工作原理
OE Affinity打分通过计算系综平均MM-PBSA能量值及基于知识的蛋白-配体相互作用打分(knowledge-based scoring)来实现。其中,MMPBSA值代表所有主要构象簇的玻尔兹曼加权平均,而基于知识的打分则通过系综平均表征配体初始结合模式下蛋白-配体结合相互作用的稳定性。
该打分方法通过整合基于物理(MMPBSA)与基于知识(基于经验的相互作用评分)的信息,可快速评估配体并进行优先级排序,从而在药物发现过程中实现高效且经济的决策支持,显著提升候选分子筛选效率。
OE Affinity打分是否可以作为OE Affinity相对结合亲合力计算的前处理步骤?
是的,您可以通过OE Affinity打分来优化计算资源分配。该方法结合基于物理(MMPBSA)与基于知识(基于经验的相互作用打分)的双重打分体系,可作为完整OE Affinity相对结合自由能计算的高效预筛选工具。为OE Affinity打分进行的系统设置(system setup)与短轨迹分子动力学平衡(short-trajectory MD equilibration)可直接复用于OE Affinity相对结合自由能计算,从而为您节省宝贵的计算时间。
是否支持使用带有不同净电荷的配体?
是的,现在已支持不同形式电荷配体之间的热力学循环计算(自2022年12月版本起)。此外,系统还会针对用户提供的映射文件(mapper files)计算边的打分(edge scores)。
是否支持个性化设置与微调优化?
OE Affinty模块提供模块化工作流(Floes)以实现个性化操作体验,支持初学者和专家用户的差异化需求。用户可选择以下两种模式:(1)预配置自动化流程(Pre-configured Automated Floes);(2)独立流程(Individual Floes)提供专家级控制(Expert Control)。
OE Affinity相对结合自由能计算提供哪些分析报告?
用户将获得结合自由能计算的全面且交互式报告。此外,用户还可无缝比较实验亲和力与计算所得的终点分析结果(预测的相对结合自由能)。
OE Affinity相对结合自由能计算的时间和成本如何?
OE Affinity相对结合自由能通过其非平衡切换(Non-Equilibrium Switching)方法,提供快速且经济高效的自由能计算。测试案例表明,该方法可在数小时内处理数十个配体,成本仅为数百美元。
OE Affinity相对结合自由能方法与传统方法的差异
OE Affinity相对结合方法(OE Affinity Relative Binding)是一种高效且经济的自由能计算方法,其技术框架源自马·普生物物理化学研究所Bert L. DeGroot实验室Vytautas Gapsys团队开发的非平衡系综(NES)方法(Gapsys et. al.,Chem. Sci. 2020, 11, 1140-1152)。
通过整合OpenEye专有的系统预平衡技术与嵌合分子计算方法,该方法实现了中间态的快速过渡和短轨迹的并行采样。相较于传统基于平衡态的计算方法,该方法在保持同等计算精度的前提下显著提升了计算效率。
OE Affinity相对结合方法的准确性如何?
该方法已在多个数据集(包括CDK2、PTP1B、Jnk1、p38、凝血酶、MCL1、Bace和Tyk2)上经过严格测试与验证。开放力场(OpenFF)和GAFF小分子力场可供使用。
Kandall τ相关系数显示,OpenEye的方法在使用GAPSYS(2020)和FEP(JACS 15)文献数据集时具有可比的准确性。这些测试结果表明,OpenEye方法在整体性能上无显著差异,是自由能计算的可靠解决方案。
是否可以使用自定义力场?
是的,从2023.2版本开始,分子动力学(Molecular Dynamics)和非平衡切换(Non-Equilibrium Switching)模块现已支持自定义力场选项。
能否从相对结合亲和力预测绝对结合亲和力?
OE Affinity模块为用户提供最大似然估计法(Maximum Likelihood Estimator),可通过相对结合亲和力数据和一组参比实验结合自由能(reference experimental binding free energies)来预测绝对结合亲和力。该估计值仅在满足以下条件时提供:图的边(graph edges)连接良好且提供部分结合自由能实验数据。
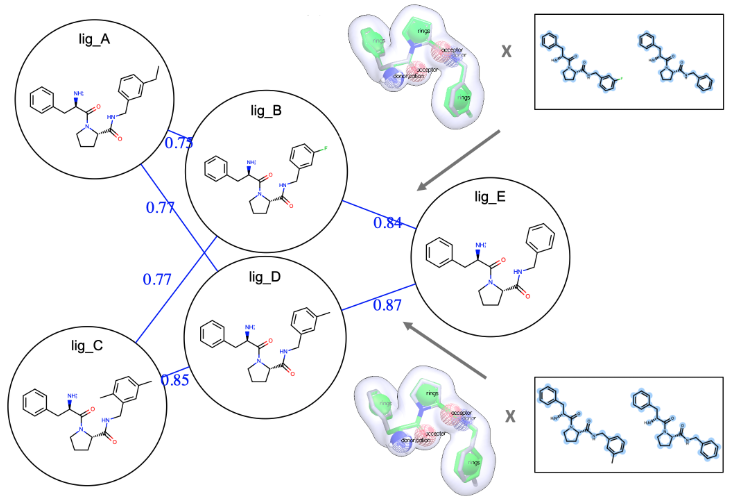
使用OpenEye的非平衡切换计算结合自由能:推动更有效的分子设计
相关文章
- Gapsys, V. et al. (2020) “Large scale relative protein ligand binding affinities using non-equilibrium alchemy,” Chemical Science, 11(4), pp. 1140–1152. Available at: https://doi.org/10.1039/C9SC03754C.
- Wagle, S., Bayly, C.I. and Mobley, D.L. (2025) “Advancing Binding Affinity Calculations: A Non-Equilibrium Simulations Approach for Calculation of Relative Binding Free Energies in Systems with Trapped Waters,” Journal of Chemical Theory and Computation, 21(15), pp. 7593–7604. Available at: https://doi.org/10.1021/acs.jctc.5c00758.
如何获取OE Affinity NES
OE Affinity NES是OpenEye云计算平台ORION的一个模块,由于美国出口政策合规性的原因,目前在大中华地区只能以计算服务(FTE)的方式来使用。



