在骨架跃迁中整合理论环系数据库:RIPK1抑制剂GDC-8264发现路径的回溯性研究
摘要:骨架跃迁是先导化合物优化中的核心策略,其成功在很大程度上依赖于高质量、结构多样的替换片段库。本文将 Peter Ertl 团队于 2024 年发布的包含近 400 万药物化学合理环系的理论数据库整合至 Cresset Spark 的静电–形状驱动骨架跃迁流程中,并以 RIPK1 抑制剂 GSK2982772 为起点分子,对其三氮唑甲基片段进行回溯性替换实验。结果表明,在仅筛选 4,311 个理化性质匹配的环系子集后,Spark 不仅高置信度地重现了后续优化中关键分子——包括工具化合物 GNE684 与临床 II 期候选药物 GDC-8264——所采用的关键双环骨架,还生成了数百个结构新颖、三维形状与静电场高度相似、且能维持与 Asp156 残基关键氢键相互作用的替代方案。本研究证实,基于生物活性分子统计规律生成的理论环系库可显著拓展骨架跃迁的化学探索边界,在保持药效特征的同时注入结构新颖性,为突破现有专利限制和加速先导化合物优化提供有力支持。
肖高铿 2025-11-03
前言
从苗头化合物(hit)到先导化合物(lead)的优化(hit-to-lead)是药物发现的关键阶段,需在生物活性、安全性、理化性质及知识产权等多个维度之间实现精细平衡。其中,生物等排体替换(bioisosteric replacement)作为一种广泛应用且行之有效的策略,常被用于从起始分子衍生出具有先导化合物特征的新结构1。Cresset开发的生物等排体与R-基团替换软件 Spark2 基于其独特的静电与分子形状相似性算法3,可针对苗头化合物的特定片段生成数百种结构多样且理化性质合理的替换建议。Spark 的片段数据库4整合了来自文献、专利、商业化合物库、合成试剂、小分子晶体结构以及理论生成环系的数百万高质量化学片段。这些经过筛选与验证的片段,构成了 Spark 生物等排体替换能力的核心基础,显著提升了其在先导优化中的效率与准确性。
环系结构是生物活性分子的核心骨架,不仅决定分子的整体三维构型,还深刻影响其理化性质、药代动力学行为及靶标结合能力。Peter Ertl 等人5通过对 ChEMBL 数据库中数十万种生物活性分子的系统分析发现,尽管药物化学中常用的分子骨架高度集中(例如,仅32种核心框架即可描述约半数已知药物),但潜在的合理化学空间仍远未被充分探索。基于此,作者从 ChEMBL 中提取出36种最常见的环拓扑结构——涵盖2至3个相连的简单环(环大小 \(≤\) 7),包括经典的稠合环与螺环体系;同时定义了16种常见原子类型(8种脂肪族、8种芳香族)。通过将这些原子类型系统地填充至上述拓扑模板,并结合一套源自生物活性分子统计规律的过滤规则(如排除不稳定缩醛、酯类、反芳香体系等),研究团队构建了一个包含约近400万(3,931,782)个结构新颖、合成可行且药物化学合理的环系数据库。值得注意的是,其中99.2%的环系在 ChEMBL 与 PubChem 等主流化学数据库中均未见报道5。这些通过计算理性设计生成的环结构,代表了尚未被实验化学充分覆盖的“暗物质”化学空间,若将其整合至 Spark 的片段库中,有望显著拓展骨架跃迁的探索边界,为先导化合物优化注入新的结构多样性。
受体相互作用蛋白激酶1(Receptor-Interacting Serine/Threonine-Protein Kinase 1, RIPK1)是调控炎症反应与程序性细胞死亡的关键节点,已成为治疗自身免疫病、慢性炎症、神经退行性疾病及某些癌症的重要靶标。目前,多个 RIPK1 抑制剂已进入临床开发阶段,适应症涵盖银屑病、类风湿性关节炎、溃疡性结肠炎等炎症性疾病,以及肌萎缩侧索硬化症(ALS)、阿尔茨海默病等中枢神经系统疾病,甚至包括胰腺癌。代表性化合物包括:GSK2982772(图1-左),首个进入临床试验的 RIPK1 抑制剂,用于炎症性疾病的治疗6;GNE684(图1-中),作为高选择性工具分子,有力验证了 RIPK1 在炎症通路中的核心作用7;以及 GDC-8264(图1-右),目前处于II期临床研究阶段,用于预防心脏手术相关急性肾损伤及主要不良肾脏事件8。鉴于该靶标已有丰富且结构多样的临床及临床前抑制剂数据,RIPK1 成为评估新环系数据库对 Spark 骨架跃迁工作流影响的理想模型体系。
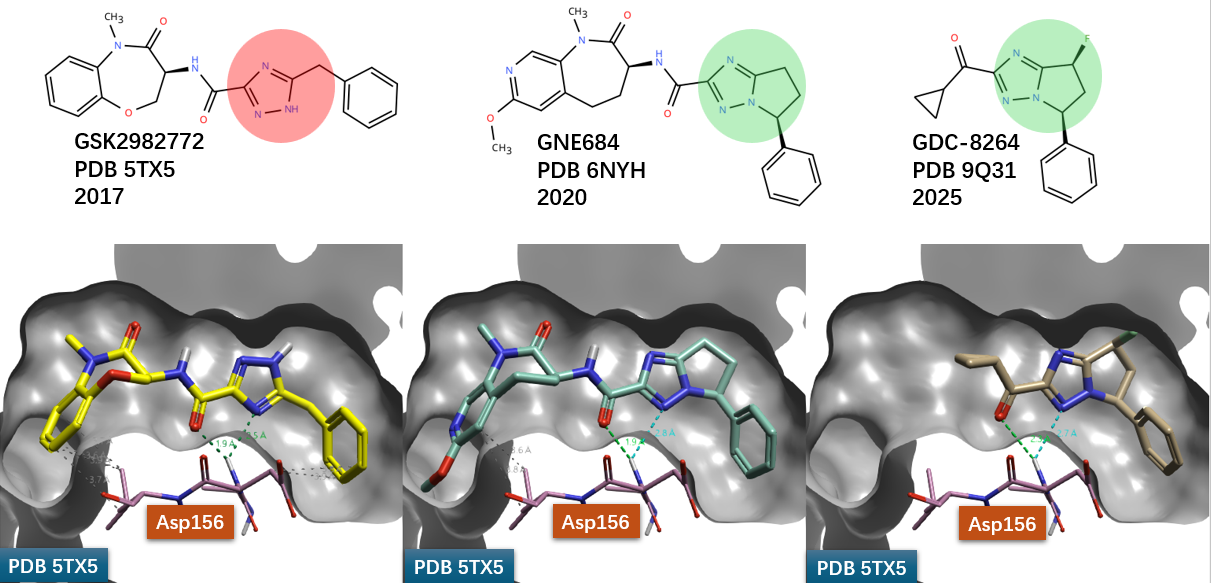
图1. RIPK1抑制剂GSK2982772、GNE684与GDC-8264的化学结构及其结合模式
考虑到 GSK2982772 的结构于2017年首次公开,而 GNE684(2020年)与 GDC-8264(2025年)相继披露,本文以 GSK2982772 为起点分子,聚焦其三氮唑甲基片段(图1中红色高亮区域),利用 Spark 对该片段进行系统性生物等排体替换。研究旨在回溯性验证:若将 Peter Ertl 的理论环系数据库整合至 Spark 片段库,是否能高效地将 GNE684 与 GDC-8264 所采用的关键三氮唑双环骨架(图1中、右绿色高亮部分)以高相似性评分优先推荐出来,从而在计算层面重现后续优化路径中的关键跃迁。
在本研究中,我们首先从 Protein Data Bank下载了 RIPK1 与 GSK2982772 的共晶结构(PDB ID: 5XT5),并利用 Flare 软件完成结构准备。随后,在 Spark 软件中启动 Scaffold Hopping or R-group Replacement 模块,以共晶结构中的配体作为起点分子,选定其三氮唑甲基片段(图2中高亮区域)作为替换目标区域,并将两个连接点(attachment points,图2红色标记)的原子类型约束为 Csp³, Csp², Car 和 Ring Only,以确保生成的生物等排体在化学合理性与合成可行性方面符合药物化学要求。
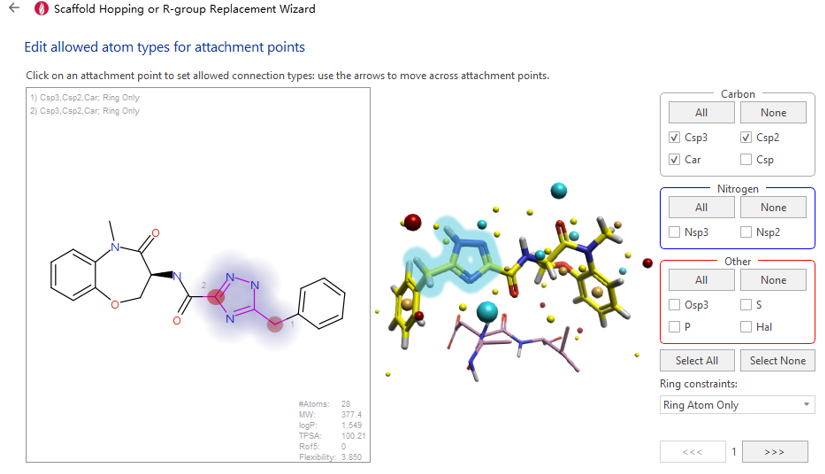
图2. Spark骨架跃迁查询结构的定义
为提升计算效率并聚焦于相关化学空间,我们未对 Peter Ertl 提供的近四百万环系数据库进行全库搜索,而是依据查询片段的理化特征设定筛选条件,仅保留结构特征相近的子集用于替换。具体过滤标准如下:
- 环数量:包含1-2个环,允许共享边、原子
- 氢键受体(HBA)数量:1-3
- 氢键供体(HBD)数量:0-2
- 非氢原子数(heavy atom): 6-10
经过上述筛选,共获得4,311个结构唯一的环系化合物。由于Peter Ertl的环系并没有取代基,因此没有必要进行分子碎片化操作,直接使用碘原子(I)对环系数据库进行连接点(attachment point)标记。在本实验中,仅对环系进行两个连接点的标记,并设定两个I原子之间至少间隔一个化学键。随后,利用Spark平台的Create Database模块,在Pre-labeled fragments模式下进行数据库构建9,10。最终,共获得86,783个具有不同双取代位点的结构片段(图3)。
图3. 数据库库的选择与选项
所有计算均采用 Spark 的“标准模式”(Calculation Method: Normal),其核心参数配置如下:最大保留结果数为 500;相似性评分基于配体整体相似性(Ligand Similarity);首轮打分引入蛋白结构作为排除体积(excluded volume),但不参与氢键或静电相互作用的显式计算;第二轮打分前对片段进行构象最小化(梯度截断值 0.700),并采用“软”蛋白排斥场(soft hardness);形状与静电场相似性权重各占 50%(Shape: 0.50;Field contributions: Positive = 1.00, Negative = 1.00, Surface = 1.00, Hydrophobic = 1.00);最终相似性度量采用 Dice 系数。
其中需要注意的是,在这次计算中,蛋白结构仅作为排除体积参与打分,未参与显式的氢键或静电相互作用打分。
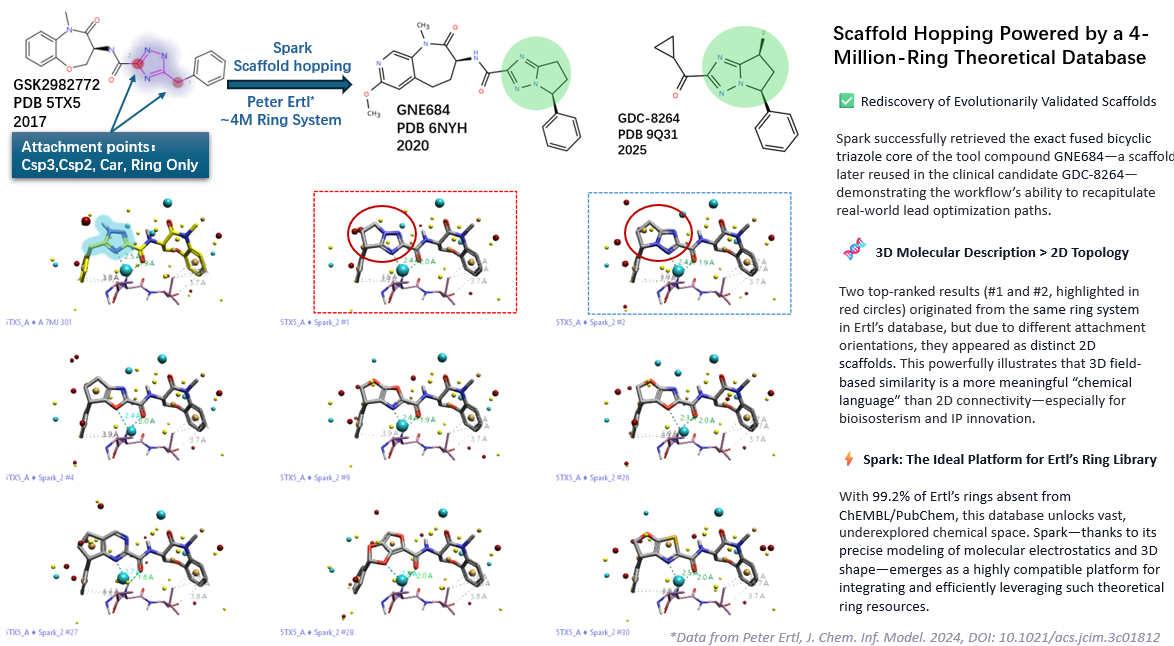
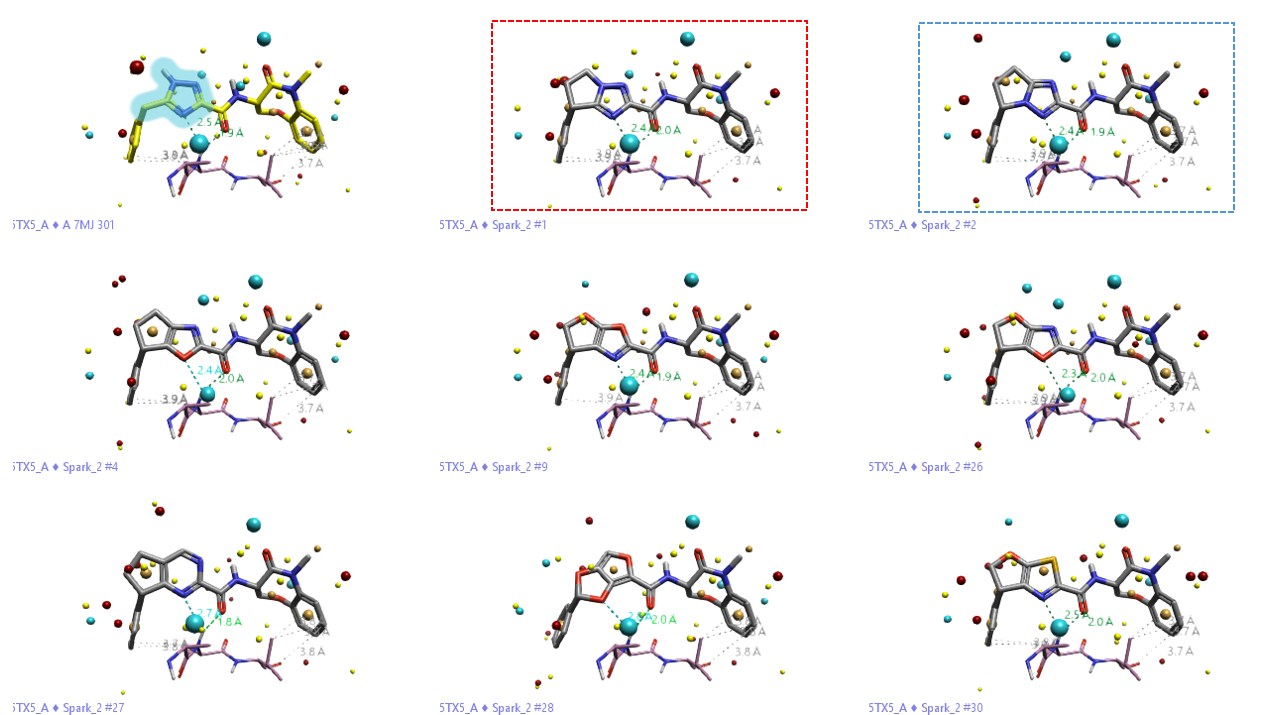
图4. 部分骨架跃迁结果示例。黄色:查询配体 GSK2982772(高亮部分为替换片段);其余为 Spark 推荐的生物等排体。
Spark 共生成 500 个替换结果,其总体相似性(Sim)、场相似性(FSim)与形状相似性(SSim)得分分别分布于以下区间:
- Sim:0.8470-0.9390
- FSim:0.7470-0.9160
- SSim:0.8660-0.9680
尽管所有结果在整体三维相似性上表现优异,仍需警惕局部关键药效特征的缺失——例如与 RIPK1 活性口袋中 Asp156 酰胺 NH 形成氢键的能力。图4展示了部分高分(黄色结构为参考配体 GSK2982772)且与Asp156 酰胺-NH 形成氢键相互作用的结果。
其中,结果 #1(红色框)与结果 #2(蓝色框)在排序中位列前两位。值得注意的是,结果 #2 成功复现了 GNE684 与 GDC-8264 所共有的三氮唑稠合双环骨架结构。尽管结果 #1 的母核在二维拓扑结构上与结果 #2 完全一致,但其取代基连接点存在差异,导致二者在整体分子骨架分类上被归为不同类型。然而,如图4所示,二者在三维空间构象及关键药效团特征方面高度相似。
这一现象揭示了“连接异构性”(connectivity isomerism)在骨架跃迁策略中的潜在价值:即使保持相同的环系与侧链组成,仅通过调整连接位点,亦可生成结构新颖且三维性质高度保守的候选分子。换言之,新骨架的发现未必依赖于引入全新化学片段,而可通过现有片段的拓扑重排实现——这为基于结构的药物设计提供了更具效率与创新性的探索路径。
除成功回溯已知临床候选物的骨架外,本研究还获得了数百个结构新颖且高分的替代方案。如图4所示,结果 #1 不仅与 GNE684/GDC-8264 共享相同的环拓扑,亦能与 Asp156 形成关键氢键;其余六类代表性骨架虽在连接方式或环组成上与已知抑制剂显著不同,但仍保持与原始配体高度的三维形状与静电场相似性,并同样具备与 Asp156 酰胺 NH 相互作用的能力。这些结果充分表明,整合 Peter Ertl 的理论环系数据库可显著拓展 Spark 骨架跃迁的化学探索边界,在维持靶标结合特征的同时,有效引入结构新颖性,为先导化合物优化提供丰富的设计选项。
讨论与结论
本研究通过将 Peter Ertl 等人构建的包含近 400 万环系的理论数据库整合至 Cresset Spark 的静电–形状驱动骨架跃迁工作流,对 RIPK1 抑制剂 GSK2982772 的三氮唑甲基片段开展了回溯性替换实验。结果表明,该策略不仅成功重现了后续优化中关键分子——包括工具化合物 GNE684 与处于 II 期临床研究的 GDC-8264——所采用的三氮唑双环骨架,还生成了数百个结构新颖、三维相似性高且理化性质合理的替代方案,充分验证了理论环系数据库在先导化合物优化中的实用价值。
值得强调的是,Peter Ertl 数据库的构建并非基于穷举式枚举,而是以 ChEMBL 中数十万生物活性分子为基础,系统提炼出 36 种常见环拓扑结构与 16 种代表性原子类型,并结合芳香性规则、抗芳香体系排除、不稳定官能团过滤及热力学稳定态选择等多重约束条件。这种“以生物活性为导向”的生成逻辑,有效确保了所产环系在化学合理性、合成可行性与药物化学相关性之间取得良好平衡。正如本研究所示,即便仅对经理化性质筛选后的 4,311 个环系子集进行搜索,Spark 仍能高效识别出与 RIPK1 活性口袋高度兼容的新颖骨架,且多数高分结果能够维持与关键残基 Asp156 的氢键相互作用——这一特征正是多数 RIPK1 抑制剂共有的核心药效要素6–8。
尤为引人注目的是,结果 #1 与 #2 虽源自同一核心环系,没有因连接取向不同而呈现出显著差异的3D场点分布,这正是生物等排体的意义所在。这种“连接异构性”(connectivity isomerism)现象揭示了骨架跃迁中一个常被忽视的设计维度:即便核心环结构保持不变,仅通过调整母核取向,即可在保留关键药效团空间排布的同时,生成在传统二维子结构检索中被视为“全新骨架”的候选分子。这一发现提示,在评估结构新颖性或制定专利规避策略时,应超越二维拓扑层面,更多关注三维药效特征的保守性与演化潜力。
此外,本研究所用蛋白结构(PDB: 5XT5)仅作为排除体积参与计算,未显式建模氢键或静电相互作用。尽管如此,高分结果仍普遍具备与 Asp156 形成氢键的能力,这侧面反映了 Spark 场相似性方法在隐式捕捉关键相互作用方面的鲁棒性。在后处理时可引入显式蛋白-配体相互作用过滤,可以进一步提升对局部药效特征的识别精度,尤其在区分相似骨架的细微信号时。
综上所述,本工作证实:将基于生物活性统计规律生成的理论环系数据库融入静电–形状驱动的骨架跃迁流程,可在维持靶标结合能力的前提下,显著拓展先导化合物的结构多样性边界。Peter Ertl 数据库中高达 99.2% 的环系在 ChEMBL 与 PubChem 中未见报道5,表明当前药物化学对合理环系空间的探索仍极为有限。Spark 凭借其对静电场与三维形状的精细刻画能力,成为整合并高效利用此类理论环系资源的高度适配平台。
因此,我们建议在 Spark 的生物等排体替换与骨架跃迁实验中积极引入 Peter Ertl 等理论环系数据库。此举不仅有助于突破现有专利壁垒,更可系统性地拓展可成药化学空间,为结构创新驱动的药物发现提供丰富且高质量的候选骨架。
致谢
本文使用了通义千问(Qwen)大语言模型辅助优化语句通顺性与学术表达。所有研究设计、计算实验、结果分析及科学结论均由作者独立完成,AI 未参与任何科学判断或内容生成。
文献
- Meanwell, N.A. (2013) “The Influence of Bioisosteres in Drug Design: Tactical Applications to Address Developability Problems,” in N.A. Meanwell (ed.) Tactics in Contemporary Drug Design. Berlin, Heidelberg: Springer Berlin Heidelberg, pp. 283–381. Available at: https://doi.org/10.1007/7355_2013_29.
- Spark. https://www.cresset-group.com/software/spark/
- Cheeseright, T. et al. (2006) “Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation,” Journal of Chemical Information and Modeling, 46(2), pp. 665–676. Available at: https://doi.org/10.1021/ci050357s.
- Spark数据库. http://blog.molcalx.com.cn/2021/04/07/spark-database.html
- Ertl, P. (2024) “Database of 4 Million Medicinal Chemistry-Relevant Ring Systems,” Journal of Chemical Information and Modeling, 64(4), pp. 1245–1250. Available at: https://doi.org/10.1021/acs.jcim.3c01812.
- Harris, P.A. et al. (2017) “Discovery of a First-in-Class Receptor Interacting Protein 1 (RIP1) Kinase Specific Clinical Candidate (GSK2982772) for the Treatment of Inflammatory Diseases,” Journal of Medicinal Chemistry, 60(4), pp. 1247–1261. Available at: https://doi.org/10.1021/acs.jmedchem.6b01751.
- Patel, S. et al. (2020) “RIP1 inhibition blocks inflammatory diseases but not tumor growth or metastases,” Cell Death & Differentiation, 27(1), pp. 161–175. Available at: https://doi.org/10.1038/s41418-019-0347-0.
- Patel, S. et al. (2025) “Discovery of Clinical Candidate GDC-8264, a Novel, Potent and Selective RIP1 Inhibitor for Amelioration of Tissue Damage and the Treatment of Inflammatory Diseases,” Journal of Medicinal Chemistry [Preprint]. Available at: https://doi.org/10.1021/acs.jmedchem.5c01891.
- SPARK教程 | 如何导入试剂创建自己的可搜索数据库. 墨灵格的博客. 2021-03-26. http://blog.molcalx.com.cn/2021/03/26/reagent-import.html
- SPARK教程 | 分子碎片化创建自己的可搜索数据库. 墨灵格的博客. 2021-04-06. http://blog.molcalx.com.cn/2021/04/06/spark-molecule-fragmentation.html