摘要:已上市的或正在研发中的共价化合物数量表明药物化学重新引起了人们对靶向共价抑制剂的兴趣。 本文以Bruton酪氨酸激酶(BTK)共价抑制剂Evobrutinib[1]的发现为例,演示如何用Flare[2]中的共价对接工作流来帮助进行共价化合物的设计及其优先排序。
Nathan Kidley
Cresset, New Cambridge House, Bassingbourn Road, Litlington, Cambridgeshire, SG8 0SS, UK
前言
Evobrutinib是最近用于治疗多发性硬化症的BTK共价抑制剂。 它显示出良好的激酶选择性(尤其是针对EGFR和Tec家族激酶),以及有发展前景的总体ADME特性。
默克公司以PDB 3GEN结合位点内的竞争性BTK抑制剂B43(见图1)作为发现Evobrutinib的起点。 在本案例研究中,我们也用该结构来演示Flare的共价对接工作流如何用于简化化合物的设计和优先级排序。

图1. B43在BTK结合位点里的结合模式(PDB: 3GEN), 绿色虚线为氢键
Flare的共价对接(Covalent docking)
共价对接(Covalent docking)是Flare中的一个模块,使用Lead Finder对接算法[3]来预测共价抑制剂的结合模式和相互作用。为了在Flare中进行共价对接计算实验,待对接的配体必须绘制为具有适当反应性的亲电形式(也称为共价弹头,covalent warhead)。一旦配体与靶标蛋白非共价结合, 这些低化学反应性的亲电基团就位于活性位点中特定的亲核残基附近,它们会迅速反应形成一个键。Flare支持的共价弹头如图2左显示。通过自定义,可以轻松扩展共价弹头列表。

图2. 左:Flare支持的共价弹头列表;右:Evobutrinib共价对接于Cys481的化学转化
在开始进行计算实验的时候,必须指定活性结合位点里的一个亲核氨基酸(选自Cys、Lys、Ser或Tyr任意之一)。然后,共价对接工作流将输入的化合物进行化学转化:修饰共价弹头以生成伪共价配体。 它没有与蛋白形成共价键,而是与所选氨基酸的质子化亲核原子成键(图2-右)。
蛋白、配体以及分子对接的准备
在Flare中下载PDB:3GEN蛋白-配体晶体结构,并充分准备以添加氢原子、优化氢键、消除原子碰撞并设定最佳质子化状态。对于准备好的体系,通过手动旋转Thr474的羟基来进一步优化以产生更有利的配体-蛋白氢键相互作用,同时也与其它BTK蛋白质结构保持一致。
由于PDB:3GEN蛋白结构(在Caldwell等人[1]的研究中作为发现Evobrutinib的起点)未与共价配体结合,因此目标亲核氨基酸Cys481的侧链可能不具有合适的构象以形成共价键。为了探索这个潜在的问题,可以复制一个蛋白结构,并为Cys481选择一个可替代的旋转异构体,然后对Cys481进行能量最小化。两种蛋白都用于探索目标亲核残基的替代旋转异构体以考察对分子对接计算的影响。
使用基于规则的质子化方式将Caldwell等人[1]的化合物A3-26导入到Flare(图3)。仔细地检查导入的配体结构的质子化状态,并手动地中和化合物A5(一种氨基吡啶类铰链结合片段)吡啶环的氮,以确保一致的结合模式并保持其与骨架Met477-NH形成氢键的能力。本数据集中的所有配体(包括A18,Evobrutinib)都带有一个迈克尔受体(Michael acceptor)作为共价弹头。
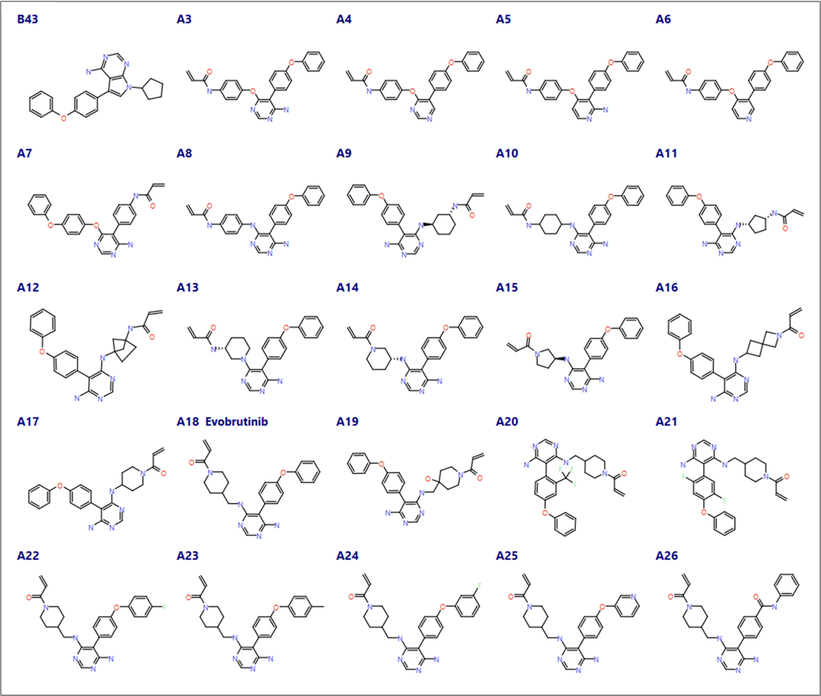
图3. Caldwell等人[1]用不同母核的化合物探索了:铰链结合区(A3-A8), 弹头与母核之间可选的连接臂(A9-A19)以及选择性的结合口袋(A20-A26)
初步的对接实验
使用Flare中的“Normal”共价对接方法,在3GEN活性位点中进行铰链结合设计(A3-A8)的探索性对接,对接时将结晶水排除在外,并将Cys481定义为亲核氨基酸残基,如图4所示。进行该初始实验是为了快速验证是否需要其它限制条件,或者需要对配体或蛋白质进行更改,以获得良好的对接结果。
在半经验分子力学函数与优化的基础上,用Lead Finder的“rank score”对每个对接实验的结合模式(pose)进行优先排序,以识别晶体学结合模式。
由于结果令人鼓舞,因此使用“Accurate but Slow”和“Very Accurate but slow”的方法进一步进行了初步共价对接实验,以优化构象采样并使得其它含更加柔性连接臂设计的结果具有最大的一致性。 “Accurate but slow”方法可以获得速度和准确性之间的最佳平衡。
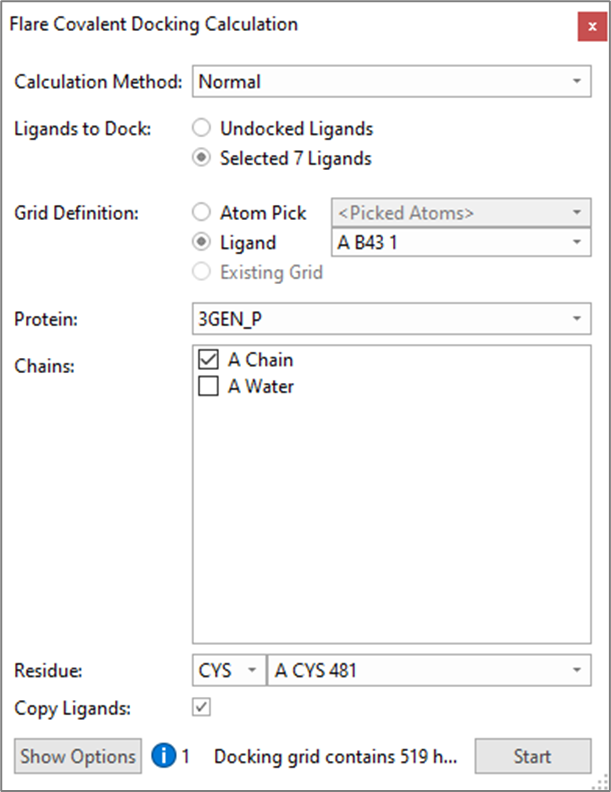
图4. Flare共价对接计算控制面板
结果与分析
对于铰接结合片段的设计(图3中的A3-A8),原始的Cys481旋转异构体获得最佳结果,这些设计是共晶配体B43类似物。Flare共价对接工作流将化合物A3和A5打分为最佳,而将A7打分为最差。
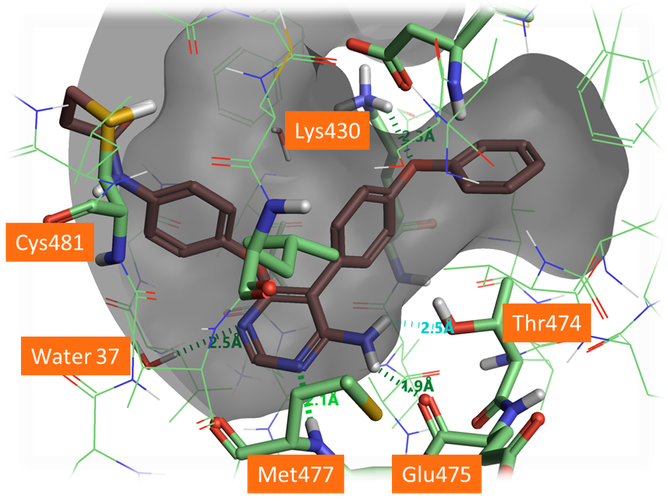
图5. A3的对接结合模式:A3与Met477的蛋白主链-NH、GLU475的蛋白主链羰基以及守门残基(gate keeper)Thr474的侧链羟基形成氢键。5位氮杂氮在活性位点与结晶水形成氢键相互作用,而该水分子在对接计算并为考虑在内。在该系列化合物的对接计算中将该结晶水分子包含在内是有益的。
如图5所示,A3和A5与Met477的主链-NH、Glu475的主链羰基以及Thr474的侧链羟基(所谓的守门残基)形成良好的氢键。由于EGFR激酶的守门残基是Met790,因此与Thr474形成氢键提供了获得比EGFR激酶对BTK固有选择性的机会。苯氧基苯基片段填充选择性口袋,与Lys430形成氢键。有趣的是,嘧啶的5-氮杂氮原子并不直接与蛋白相互作用,而是与原有B43共晶的结晶水发生氢键相互作用。在以后的嘧啶铰链结合片段系列化合物对接实验中将该水分子包含在内将有利于最有前途结合模式的识别和打分。
化合物A4和A6缺少与Thr474形成氢键的2-氨基取代基,但其它相互作用与化合物A3和A5观察到的相同。由于该药物发现工作的目的是靶向良好的激酶选择性,所以与BTK Thr474不形成相互作用可能会影响BTK与EGFR的固有选择性。
A7带有一个反向的氨基嘧啶片段,它无法像其他铰链结构域的设计一样形成相同的相互作用,这与Caldwell等人[1]报道的对BTK和EGFR较低的活性一致。
连接臂(linker,图3中的A9-A19)设计的共价对接,无论是对原始的和还是建模的Cys481旋转异构体,都产生了良好的结合模式。大多数化合物在所有对接实验中均表现出相似的结合模式,仅在连接臂的构象上观察到差异。这些设计很难确定优先级,因为活性位点的该区域是开放的并且溶剂可及的,这限制了对接的见解。当使用原始的Cys481旋转异构体进行对接时,化合物A11和A13-15的打分很差;而用建模的可替代Cys481旋转异构体时,化合物A13和A16的打分很差。在不存在蛋白质的情况下,对结合模式的构象进行能量最小化可以帮助推断化合物内能。
选择性口袋的设计还聚焦于改善苯氧基苯基环的微粒体稳定性。对接打分和蛋白-配体接触分析可以识别出:A20和A21苯环上的取代基与两种Cys481旋转异构体都存在空间冲突。该组的其它化合物A22-A26的末端苯环有所变化,并且契合到选择性口袋里。
与常规的对接一样,准备蛋白并使之具有正确的构象对结果有重大影响。在本研究种,柔性较低的配体倾向于使用起始的Cys481旋转异构体,例如在母核和弹头之间包含芳族连接臂的铰链结合片段设计。当使用替代的Cys481旋转异构体时,连接臂设计中更柔性的配体会产生更好的对接模式。
比较对接计算的与X-ray晶体结构的Evobrutinib结合模式
将Evobrutinib共价结合到BTK活性位点的晶体结构(PDB:6OMU)与Evobrutinib在PDB 3GEN活性位点内的对接位结合模式进行比较,可以突显两者之间的差异(图6)。特别是,6OMU中Cys481侧链的构象确实与3GEN的构象不同:6OMU活性位点中Lys430的构象和一些结晶水也存在细微的差异。
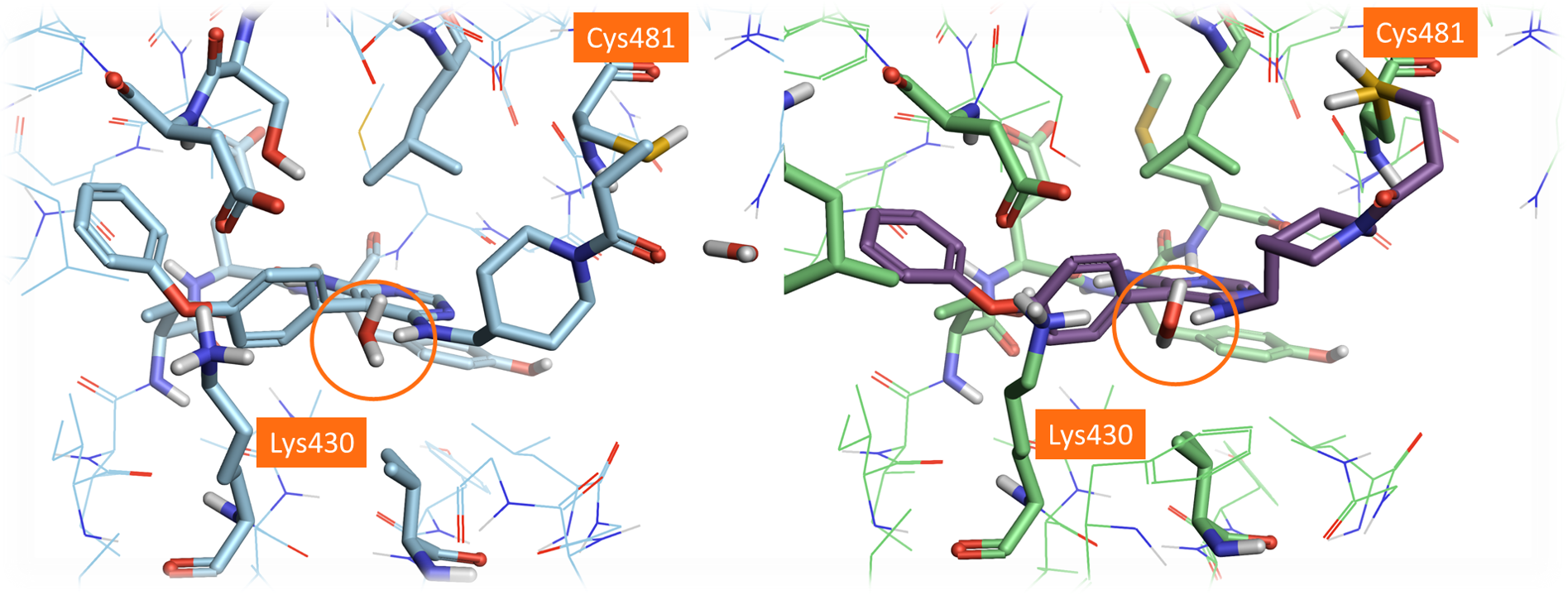
图6. 左:Evobrutinib共晶结构(PDB:6OMU);右:Evobrutinib在PDB 3GEN干结合位点里以“Accurate but slow”模式对接计算的最佳打分结合模式。通过比较两者,可以发现Cys481的侧链存在差异。
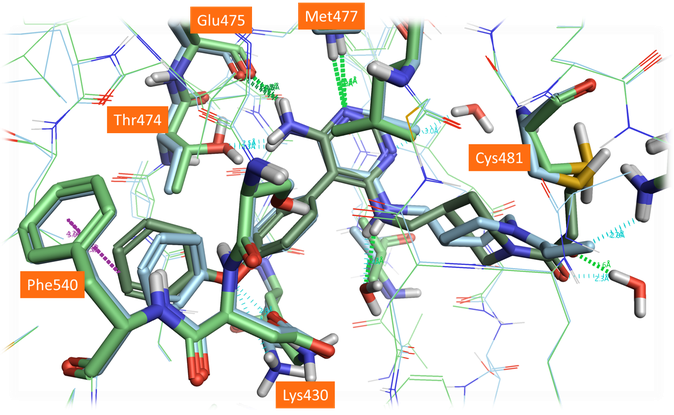
图7. Evobrutinib共晶结构(PDB: 6OMU,左蓝色)以及用可选的Cys481旋转异构体进行对接计算的最佳结合模式(绿色)、结合位点里的结合水
建模的Cys481侧链构象更接近其在6OMU中的实验位置(图7)。毫不奇怪,为亲核残基选择不同的旋转异构体会影响化合物的整个结合模式:使用Cys481替代的旋转异构体会显着改善结果,但连接臂构象仍与晶体结构中观察到的构象略有不同。通过在3GEN蛋白结构的活性位点中包含选定的水分子,特别是与连接臂胺临近的“1024”水分子,可以进一步改善这一点(图7)。最终的结合模式非常接近于在晶体结构中所观察到的,仅是在饱和环构象上有微小的差异。
结论
Flare提供了一个用户友好的界面,可用于准备、设计和对接您的共价配体。 对接工作流会生成伪共价配体,就像常规对接一样可以将结果保存在Flare项目中,从而有助于探索不同的对接结合模式,并进一步对结果进行后处理,从而为药物发现项目的进展选择最佳候选化合物。
文献
- Caldwell, R. D.; Qiu, H.; Askew, B. C.; Bender, A. T.; Brugger, N.; Camps, M.; Dhanabal, M.; Dutt, V.; Eichhorn, T.; Gardberg, A. S.; Goutopoulos, A.; Grenningloh, R.; Head, J.; Healey, B.; Hodous, B. L.; Huck, B. R.; Johnson, T. L.; Jones, C.; Jones, R. C.; Mochalkin, I.; Morandi, F.; Nguyen, N.; Meyring, M.; Potnick, J. R.; Santos, D. C.; Schmidt, R.; Sherer, B.; Shutes, A.; Urbahns, K.; Follis, A. V.; Wegener, A. A.; Zimmerli, S. C.; Liu-Bujalski, L. Discovery of Evobrutinib: An Oral, Potent, and Highly Selective, Covalent Bruton’s Tyrosine Kinase (BTK) Inhibitor for the Treatment of Immunological Diseases. J. Med. Chem. 2019, 62 (17), 7643–7655. https://doi.org/10.1021/acs.jmedchem.9b00794.
- Flare. https://www.cresset-group.com/software/flare
- Lead Finder. https://www.cresset-group.com/software/lead-finder



