摘要:Flare V6是我们基于配体和基于结构的药物设计平台。本次先睹为快介绍了即将发布的Flare™中几个计算化学相关的新功能:Hit expander、新增机器学习方法(多层感知机)以及本地与远程计算新增了控件。
Flare V6是我们基于配体和基于结构的药物设计平台,此次先睹为快探索了即将发布的Flare™中的新功能。
新:Hit Expander
Hit Expander是Flare的一个新模块,使用户能够围绕单个苗头化合物或先导化合物进行快速探索。它通过在苗头化合物所有未取代位置上添加小的取代基(图 1)来起作用。 在适当的情况下也进行芳香族的单原子取代。
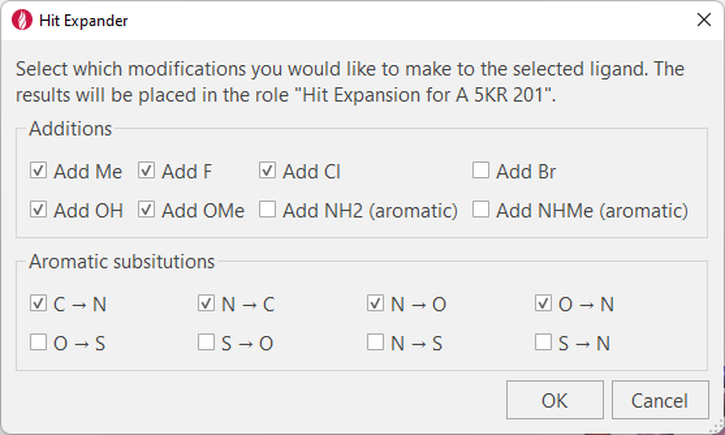
图1. Hit Expander面板使您可以在苗头化合物上进行单点加成和替换
枚举之后,对新配体稍稍进行最小化并放置在它们自己的角色中(图 2,右)。
然后可以通过使用最小生成树(图 2,右)、或使用现有 QSAR 模型、短的1ns Flare FEP计算对原始配体的枚举出的变体进行优先级排序。
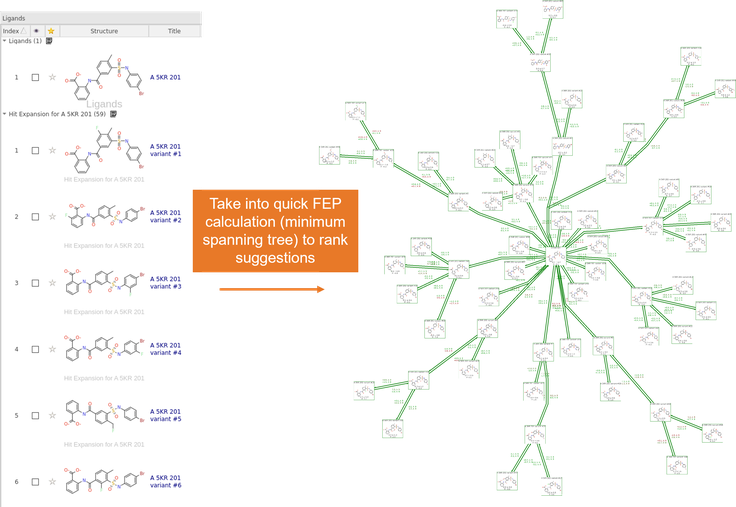
图2. Hit Expander枚举的变体可以通过短的1ns FEP计算来确定优先级
对于像Hit Expander之类的微小结构变化,较短的1ns Flare FEP 模拟产生的结果与更精确的4ns计算非常吻合(图 3)。这样可以快速对结果进行分类,以摆脱糟糕的替换,然后可以对最有希望的候选分子进行更精确的计算。
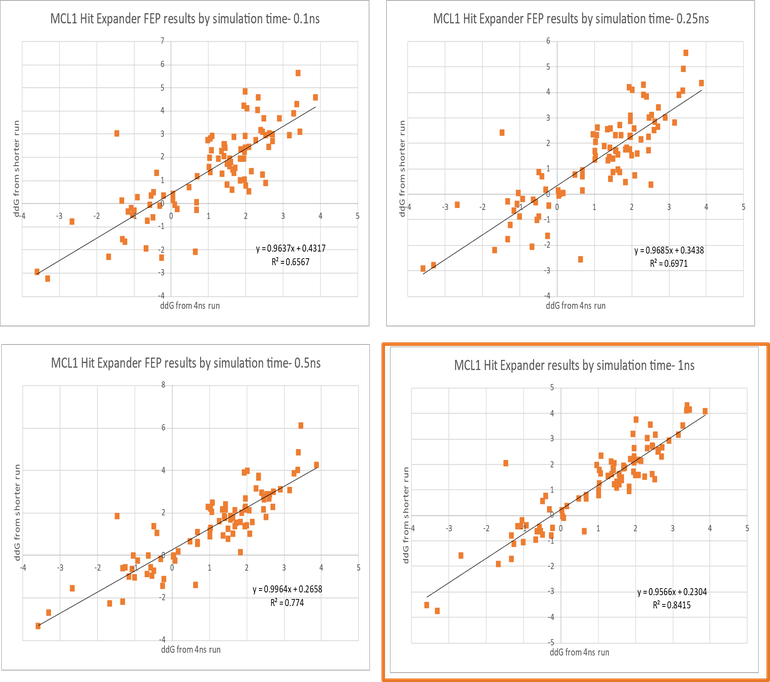
图3. 对于仅包含较小结构变化的数据集,快速的1ns模拟与更准确的4ns模拟结果非常一致
增强的QSAR
在Flare 中现有的强大且经过充分验证的机器学习 (ML) 方法基础上新增了多层感知器 (MLP) 回归和分类模型(图 4)。

图4. QSAR模型构建编辑器可以访问稳健且经过充分验证的机器学习方法
与以前版本的Flare一样,这些方法可使用Cresset 3D描述符模拟叠合好配体的静电和形状特性或自定义2D和3D描述符用于构建活性与ADMET性质的预测性QSAR模型。
在此版本中,列与活性编辑器(Column & Activity Editor) 使得自定义描述符变得更加容易,其中可以轻松选择性质组并将其设置为用于模型构建的 QSAR 描述符(图 5)。
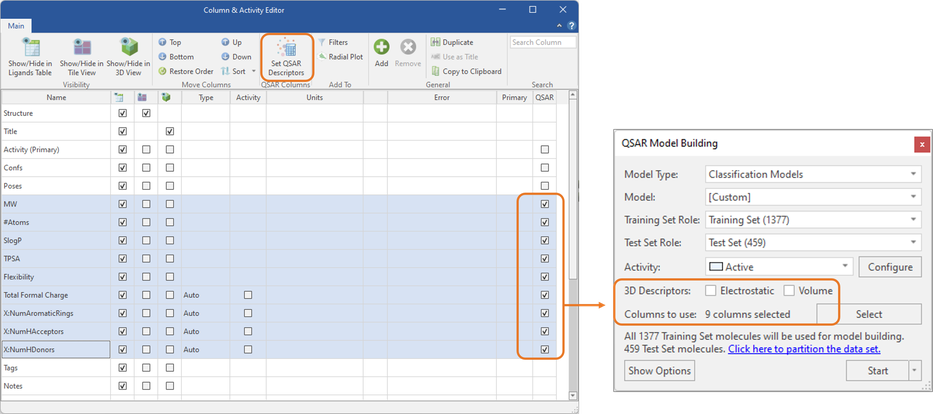
图5. 列和活性数据编辑器简化了QSAR模型构建选择自定义描述符
更多的本地与远程计算控件
如果您可以通过 Cresset Engine Broker™ 访问远程集群上的资源,按下“开始”按钮下拉菜单将使您能够控制计算开始运行的位置(图 6)。
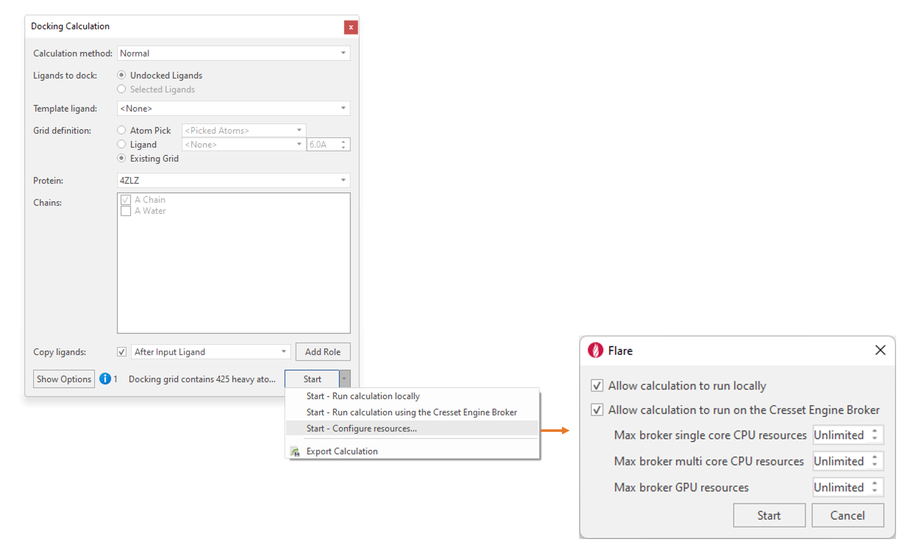
图6. 为Flare计算配置本地与远程计算资源
可选择的内容包括:
- 在本地运行计算,仅使用本地资源
- 使用Cresset Engine Broker运行计算(如果您无权访问,请联系我们)
- 配置资源:选择按下“开始”按钮时发生的情况(默认情况下,本地和远程计算都允许,Flare 将选择可用的最佳计算资源)
- 导出计算:可用于对接、构象搜索和叠合、分子动力学模拟和Flare FEP计算等等。此选项导出Flare Python脚本,用pyflare二进制文件从命令行运行所选实验。



