摘要:通过将基于结构的计算方法、结构生物学和化学知识与基于配体的计算方法相结合,可以优化虚拟筛选的起点,识别可能具有改善的受体亚型选择性的化合物。本文以选择性CB2配体的发现为例,演示了如何分析起点并正确对虚拟筛选进行分级挑选,从而对化合物进行识别、选择和优先级排序。然后,从虚拟筛选所使用的商业化合物库中购买或交给化学家或药物化学家进行合成,随后交给药理学家进行生物学测试。
原文:Keverne Louison; Scott Midgley. Virtual screening – Identifying leads with selectivity for Cannabinoid receptor 2 from Cannabinoid 1 binders
编译:肖高铿/2024-04-10
前言
大麻素(Cannabinoid,CB)受体是一类G蛋白偶联受体(GPCR),在多个治疗领域一直是活跃的研究领域。选择性靶向CB2受体是有效的镇痛剂,由于没有CB1结合的精神类作用,因此可能减少基于阿片类镇痛剂的使用和相关的成瘾风险。同时,CB2的免疫调节特性和能力可能有助于治疗肥胖症1,2。
虚拟筛选可为寻找新的潜在选择性苗头化合物提供起点。虚拟筛选可以在共晶配体上进行,而无需任何预先建模。然而,通过将基于结构的计算方法、结构生物学和化学知识与基于配体的计算方法相结合,我们可以优化虚拟筛选的起点,识别可能具有改善的受体亚型选择性的化合物。
本研究演示了如何分析起点并正确对虚拟筛选进行分级挑选,从而对化合物进行识别、选择和优先级排序。然后,从虚拟筛选所使用的商业化合物库中购买或交给化学家或药物化学家进行合成,随后交给药理学家进行生物学测试。
方法
在本研究中,我们从获得CB1和CB2受体的公开晶体结构开始。然后分析蛋白-配体复合物以发现选择性靶向CB2配体的机会。
从蛋白质数据库(PDB)下载临床前研究配体(图1)冷冻电镜复合物结构6KPC(CB2)和6KPG(CB1),并使用Flare™进行结构准备3,4。然后将两个蛋白结构的G蛋白偶联受体(GPCR)结构域进行比对,以清楚地显示两种受体的结合口袋。这样,可以识别CB2受体中潜在的亚型特异性残基和区域,以引发与新配体的靶向选择性相互作用。将每个受体PDB结构的天然配体对接到另一个受体的PDB结构中,以产生配体在两种受体结合位点中的结合模式。
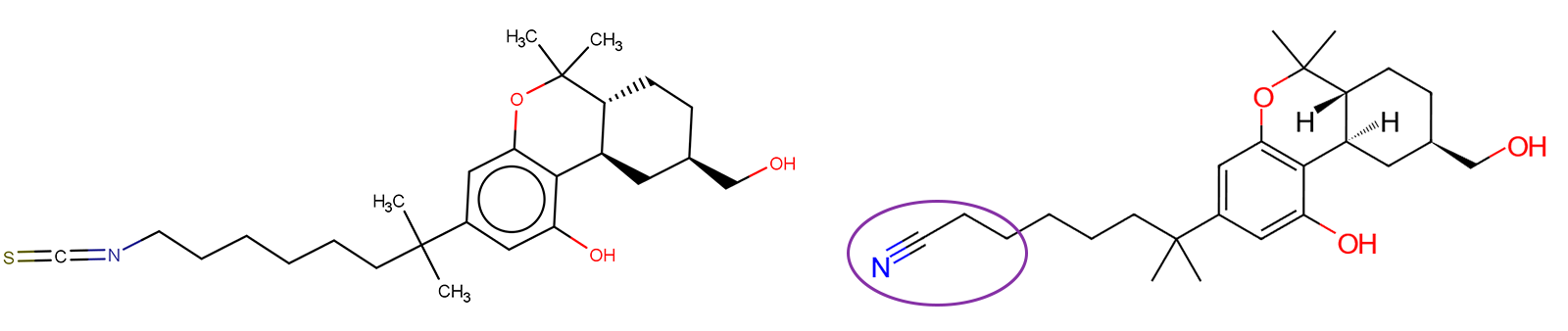
图1. CB1配体AM12033(PBD 6KPG,左)与CB2配体AM841(PDB 6KPC,右)的冷冻电镜结构
选择CB2 cryo-EM配体AM841中紫色圆圈高亮显示的原子(图1)来启动对受体结合口袋的评估。进行R基团替换有助于识别可以利用该口袋的新结构,其可以与CB2中存在、而在CB1中不存在的Ser165残基相互作用而可能对CB2具有选择性。这可使用Spark™ Docking工作流来实现5。对结合模式进行研究,可以观察到配体AM841中的氰基延伸到CB2结合口袋内的宽敞空腔中(图2)。
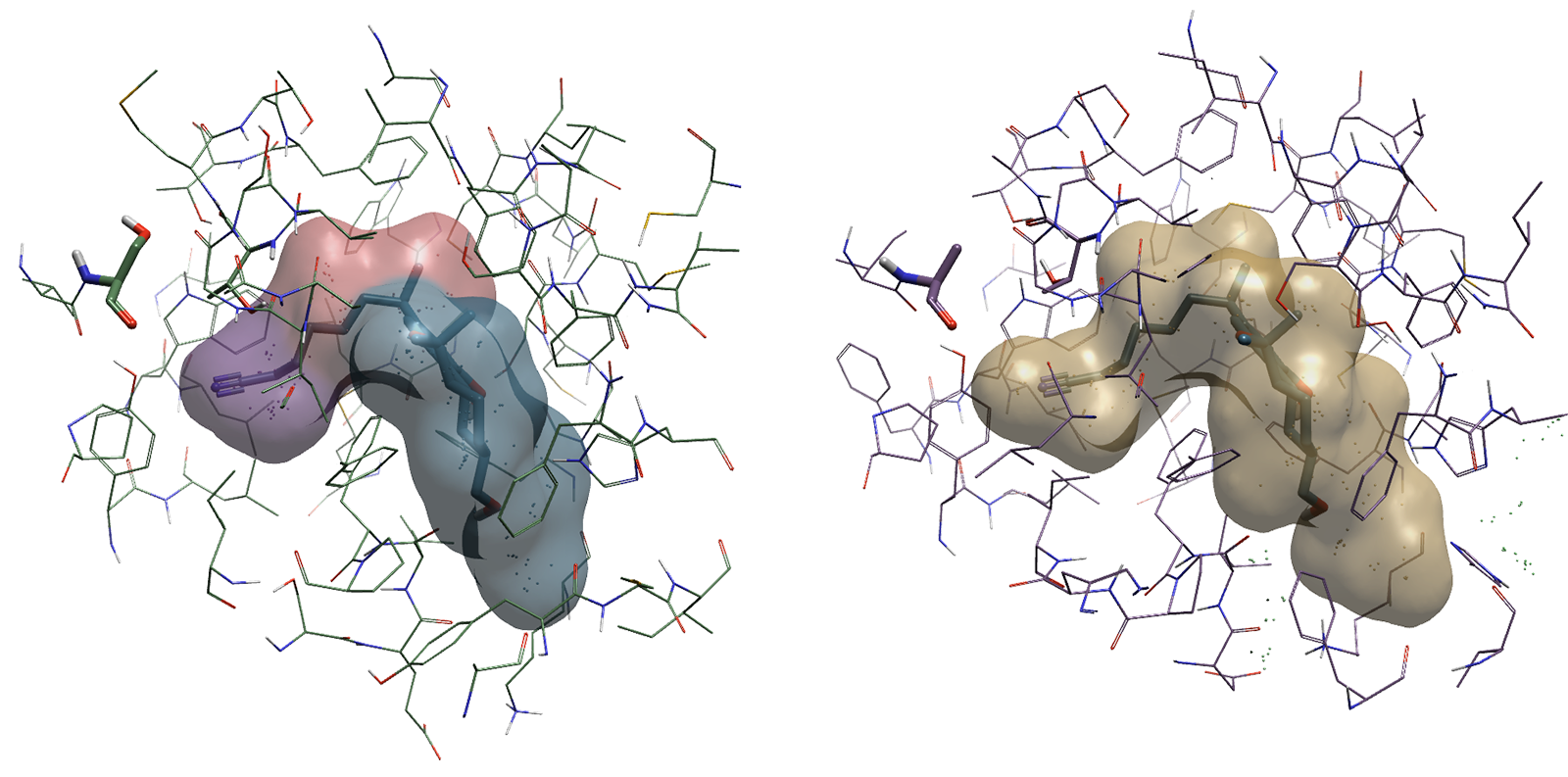
图2. CB2(左)与CB1(右)的结合口袋:在CB2结合腔附近存在一个残基Ser165,而在CB1对应的位置为Ala248.
对配体AM12033进行R基团替换实验,使用CB2的结合口袋作为排除体积,并对ChEMBL和SureChEMBL的very common与common片段库进行搜索。
对Spark结果的分析需要进行分级分类以去除不适合所研究项目的化合物。通过对药物化学和对靶标的生物学理解,仔细考虑分级分类步骤中使用的参数。首先,为了防止对结合熵惩罚产生的不利影响,将配体柔性限制为最多5个可旋转键。环的数量被认为是减轻这种情况的一种方法,但必须仔细平衡以防止分子变得太疏水,因此设置不超过4个环的阈值。最后,过滤掉含有化学上难处理基团的配体。对剩余的配体进行对接,以在CB1和CB2受体中产生所选配体的结合模式。在这个阶段,可以进行额外的检查,例如去除含有诸如PAINS之类等有问题基团的配体。
一旦获得了每个配体在两个受体中的结合模式,就可通过python脚本调用Flare API(pyFlare)对配体对接实验的结果进行分级分类。该脚本计算每个配体在两个受体中的静电互补性(Electrostatic ComplementarityTM,EC)6以及Lead Finder打分LF VSscore7,并计算配体在两个受体上的打分值的差值,其中diff = CB1 score – CB2 score。负值越大,表示配体对CB2相对于CB1的选择性越强,这使得能够过滤出那些对CB2具有更有利相互作用的配体。基于所有配体打分的数据范围,将LF打分差值的截止值设为3、EC打分差值的截止值设为0.3,对Spark结果进行过滤,这意味着仅保留与CB2和CB1之间的LF打分差值\(\ge 3\)以及EC打分差值\(\ge 0.3\)的配体。
在对结果进行分级分类筛选之后,Cresset Discovery科学家们利用他们对中枢神经系统活性配体的了解来筛除掉不太可能的候选物。去除了含有以下成分的配体8:阴离子、具有3个以上氢键供体的配体、7个以上氢键受体、8个以上可旋转键和高度疏水性的配体(预测LogP\(\ge 3\)的配体)。在此步骤结束时,选择满足所有所需条件的单一代表性化合物1(图3)作为Blaze™虚拟筛选9的新参考起点。
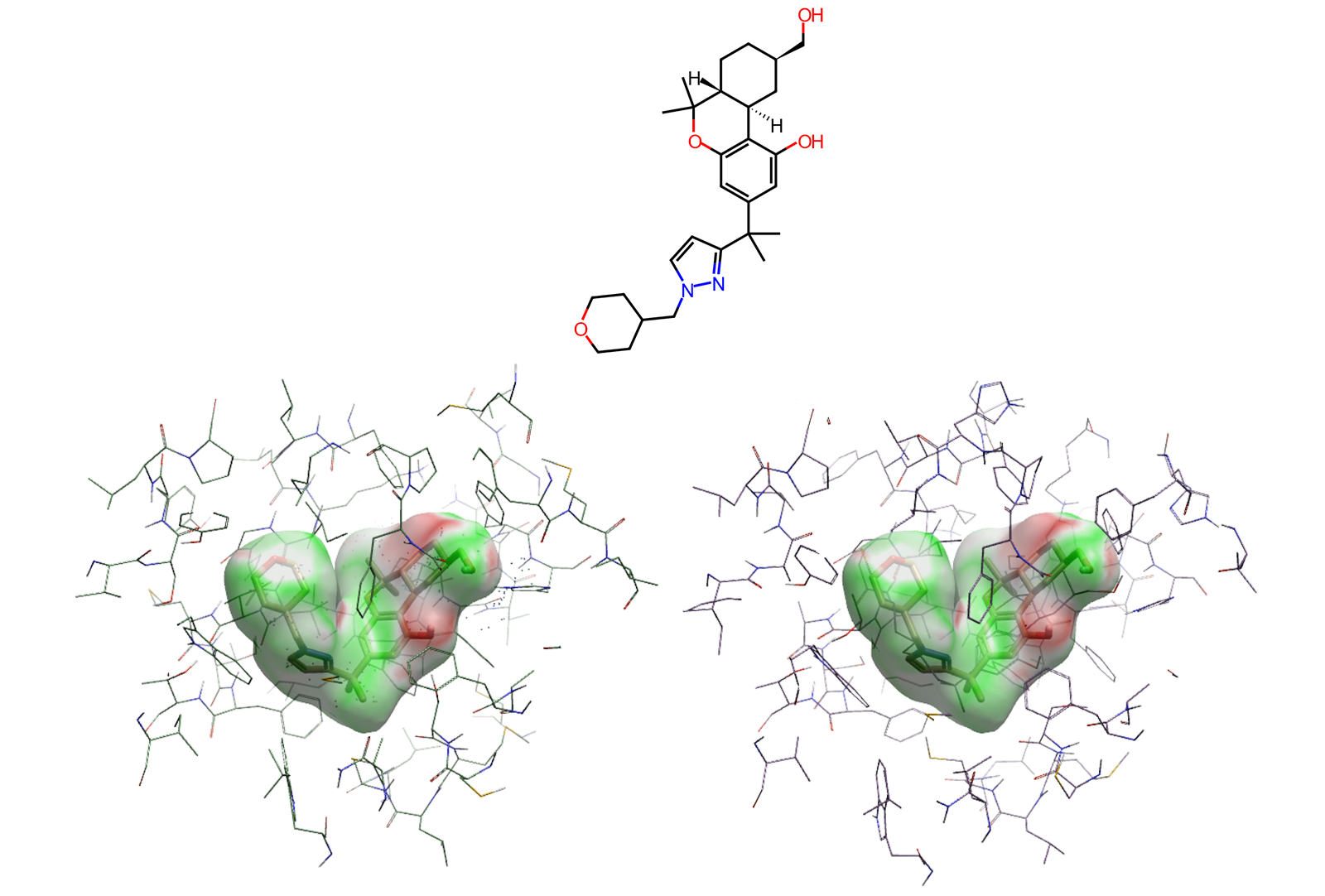
图3. 化合物1的2D结构(上)与3D结构,其表面分别以与CB2(左下)、CB1(右下)的静电互补性来着色。
将化合物1(图3)和AM12033作为参比模板分别进行独立的虚拟筛选,搜索ChEMBL、Enamine Advanced、Enamine HTS、Enamine Premium、Molport以及WuXi筛选数据库。CB2用作这些筛选的排除体积。
然后对结果过滤并除去阴离子。通过自定义pyFlare脚本,使用先前定义的标准对打分最高的10000个苗头化合物进行了分级处理。剩余的苗头化合物被重新对接到CB1和CB2的结合口袋,并计算EC和LF打分的差值。将这些结果绘制在直方图中,以可视化化合物1和AM12033的虚拟筛选指标。
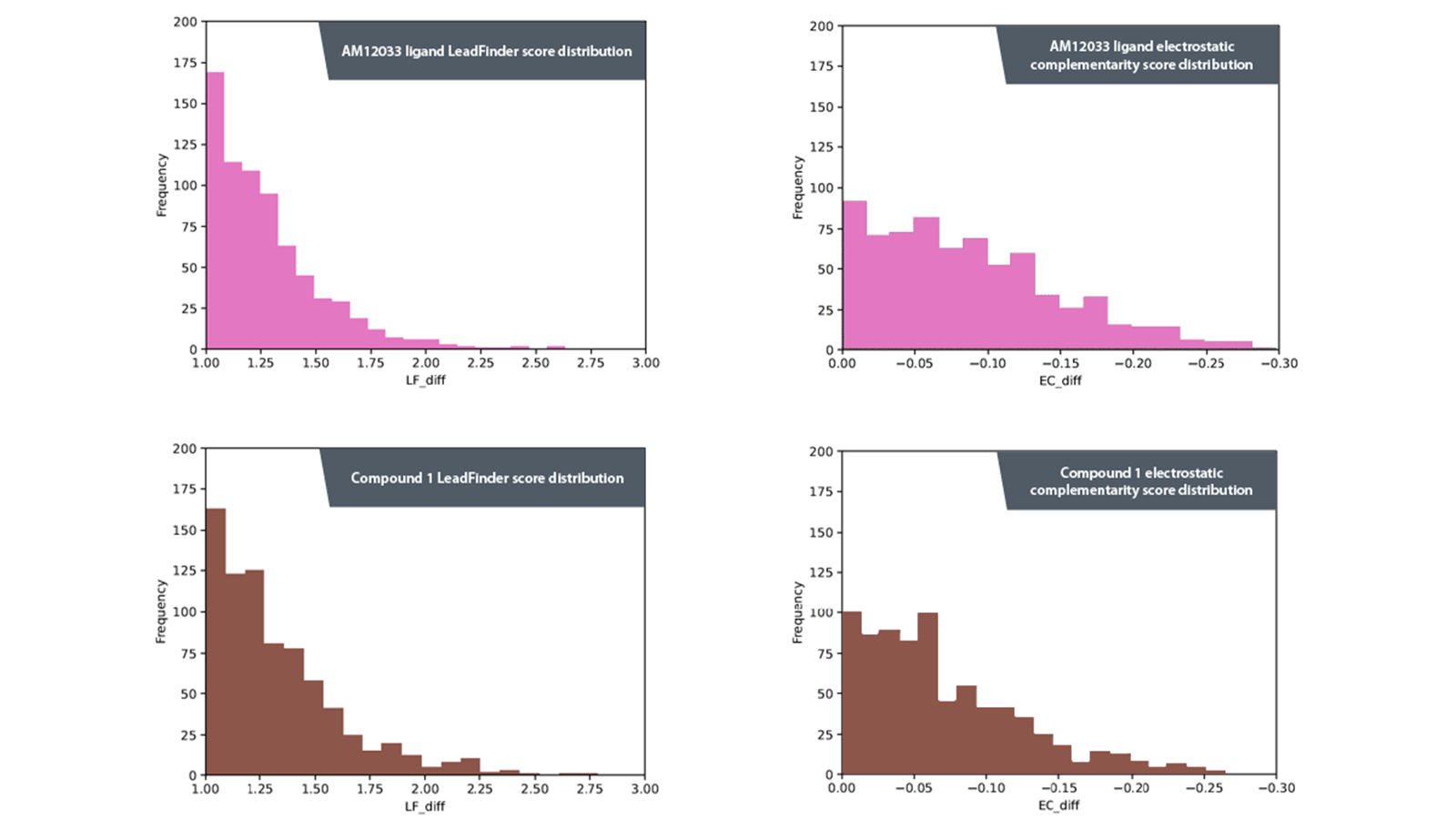
图4. 直方图显示了不同受体虚拟筛选结果的LeadFinder(LF)打分与静电互补性打分(EC)之间差异的分布。左上角:AM12033的LF打分分布;右上角:AM12033的EC打分分布;左下角:化合物1的LF打分分布;右下角:化合物1的EC打分分布。
如图4所示,化合物1比AM12033产生更加右偏的直方图,这表明在虚拟筛选之前的准备工作可能有利于为受体亚型选择性配体找到合适的起点。
分析图表可以发现,在化合物1产生的结果里面,从对接打分和EC打分上看,有更大比例的配体对CB2的选择性偏好高于对CB1。对接打分差值小于1或在负值范围内说明对CB1具有选择性,并且由于本研究的目的是识别对CB2具有选择性的化合物,因此将化合物1在负范围内的结果被去除,然后使用药物化学知识对剩余的化合物进行彻底的视觉分析,以确定可以购买或合成用于生物测试的化合物的最终列表。
图5显示了最终分级分类处理后排序靠前的四个例子。该实验提供了一系列结构多样的分子,这些分子都显示出对CB2的选择性,并且满足了使用靶标、位置以及合成化学知识的约束条件。
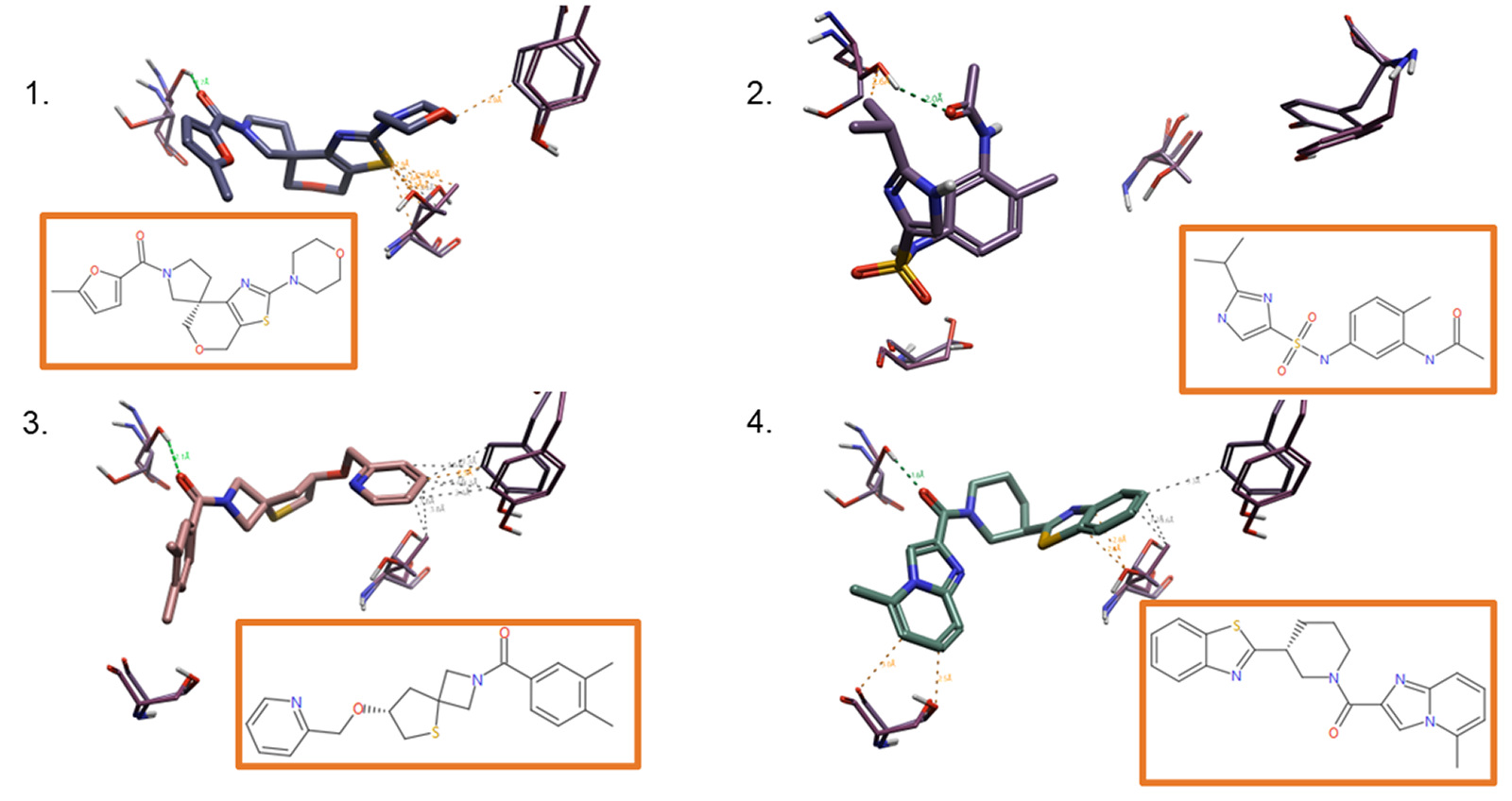
图5. 分级挑选之后,打分排序靠前的化合物示例(2D与3D)
观看Webinar录像了解更多
要直观演示此工作流程中执行的步骤,请观看我们的网络研讨会录像:通过虚拟筛选识别具有靶标选择性的先导化合物——优化起点以预测最有可能成功的分子,请点击这里申请播放录像。
参考文献
- Rossi, F.; Punzo, F.; Umano, G.R.; Argenziano, M.; Miraglia Del Giudice, E. Role of Cannabinoids in Obesity. Int. J. Mol. Sci. 2018, 19, 2690. https://doi.org/10.3390/ijms19092690
- Verty AN, Stefanidis A, McAinch AJ, Hryciw DH, Oldfield B. Anti-Obesity Effect of the CB2 Receptor Agonist JWH-015 in Diet-Induced Obese Mice. PLoS One. 2015 Nov 20;10(11):e0140592. https://doi.org/ 10.1371/journal.pone.0140592.
- Flare™, version 7.1, Cresset®, Litlington, Cambridgeshire, UK; https://www.cresset-group.com/flare/
- https://www.cresset-group.com/discovery/specific/protein-preparation/
- Hua T, Li X, Wu L, Iliopoulos-Tsoutsouvas C, Wang Y, Wu M, Shen L, Brust CA, Nikas SP, Song F, Song X, Yuan S, Sun Q, Wu Y, Jiang S, Grim TW, Benchama O, Stahl EL, Zvonok N, Zhao S, Bohn LM, Makriyannis A, Liu ZJ. Activation and Signaling Mechanism Revealed by Cannabinoid Receptor-Gi Complex Structures. Cell. 2020 Feb 20;180(4):655-665.e18. https://doi.org/10.1016/j.cell.2020.01.008.
- Spark™, version 10.7, Cresset®, Litlington, Cambridgeshire, UK; https://www.cresset-group.com/spark/
- Flare™, Cresset®, Litlington, Cambridgeshire, UK; https://www.cresset-group.com/flare/; Bauer M., Mackey M.; Electrostatic Complementarity as a Fast and Effective Tool to Optimize Binding and Selectivity of Protein–Ligand Complexes J. Med. Chem. 2019, 62, 6, 3036-3050
- Lead Finder™, version 7.1, BioMolTech®, Toronto, Ontario, Canada; https://www.cresset-group.com/lead-finder/
- Pajouhesh H, Lenz GR. Medicinal chemical properties of successful central nervous system drugs. NeuroRx. 2005 Oct;2(4):541-53. https://doi.org/10.1602/neurorx.2.4.541.
- Blaze™, version 10.3.2, Cresset®, Litlington, Cambridgeshire, UK; https://www.cresset-group.com/blaze/
请联系Cresset,使用我们的CADD软件或Discovery CRO加速您的项目
Cresset提供软件和发现服务,以加强您的研究工作。我们的专家团队在药物和计算化学以及生物学方面拥有丰富的经验,可以与您一起工作以实现您的项目目标,而我们的软件提供了前沿的方法,可以为您的项目提供有价值的见解。
想要了解更多有关我们如何支持您进行虚拟筛选项目的信息,请联系我们进行CADD软件评估或与我们的Discovery CRO团队进行保密讨论。



