摘要:本文演示了如何用SMARTS语言编码羧酸、伯胺以及它们之间的反应,并借助化学信息学软件包RDKit在KNIME界面实现基团识别、化合物过滤以及进行双组份的化学反应,最后生成组合库并保存为SDF格式的文件。
作者:肖高铿
发布:2018-07-25
一.前言
药物化学研究中通常需要根据母核或反应去枚举组合库,后者称为基于反应的组合库,可以帮助化学家了解自己所能掌握、合成的真实化学空间。比如,您想枚举各种取代苯甲酸与各种伯胺反应生成酰胺,你可以使用基于反应的组合库来实现。假设你有10个苯甲酸化合物、10个伯胺,其中一些反应如下图1所示:
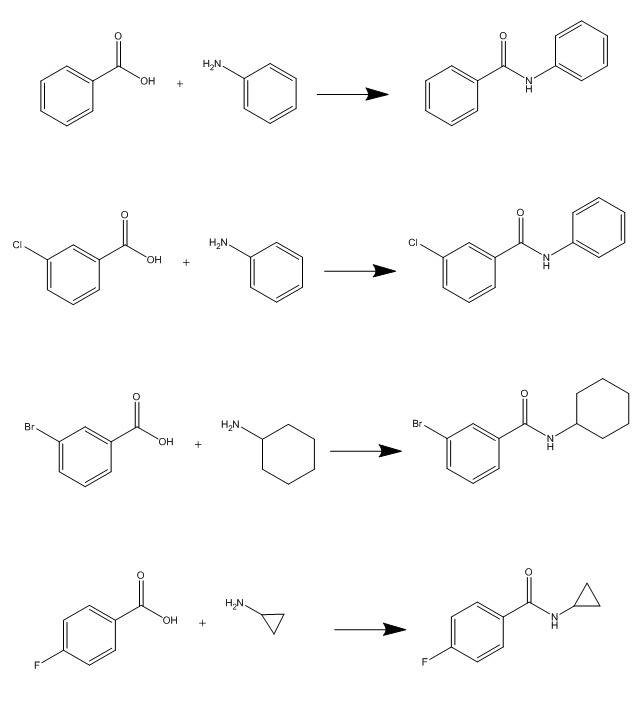
图1. 部分苯甲酸与伯胺反应的例子
我们可以通过绘制一个反应路线来合成一个化合物库,通过化学信息学技术将反应物枚举出来:每个反应物、产物可以用SMILES1来编码或其它的格式来表征,各种基团可以用SMARTS2来表征、查询、计数数量,反应可以用SMILES、SMARTS、SMIRKS3或RXN来表征。比如,乙醇与乙酸生成乙酸乙酯的反应可以写成下式反应(注意,在不同的浏览器观察反应效果会不同):
1 | CCO.CC(=O)O>>CCOC(C)=O |
你会发现用分子语言进行一个反应是非常简单的事情,与写SMILES没什么根本的区别。你可以用这些分子语言让各种试剂、中间体进行“反应”最后得到虚拟的产物库即组合库。组合库设计可以是如图1单步反应,也可以是如图2所示的多步反应。
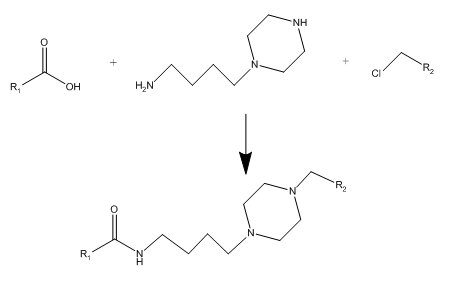
图2. 组合库支持多步骤反应
基于反应的组合库设计不仅支持上述的线型分子设计,还可以支持如图3所示成环反应步骤。
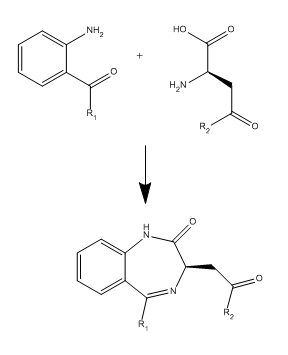
图3. 成环反应
这些复杂反应的组合库设计主要得益于SMILES、SMARTS与SMIRKS语言及其原子定位(Atom map)功能。利用这些分子语言,可以很容易构建自己的基于反应的组合库,比如用于从头设计5。
获得组合库之后,你可以进一步用各种虚拟筛选技术(比如基于分子对接、药效团、分子形状的虚拟筛选方法)、活性预测技术来研究组合库,从中发现潜在的、有价值的化合物,最后进一步去合成、生物活性测试。
二. 基于反应的组合库生成
本教程以苯甲酸与伯胺反应生成苯甲酰胺为例,举例如何建立苯甲酰胺组合库,包括下面流程:1)读入试剂化合物数据库;2)用SMARTS语言识别试剂数据库中的羧酸/伯胺类化合物、并计算每个化合物的羧酸/伯胺基团数量、保留仅含有一个羧酸/伯胺基团的化合物;3)用SMARTS语言建立羧酸与伯胺的反应、生成产物;4)产物的理化性质计算与过滤;5)保存产物为sdf格式。本教程是根据KNIME自带的组合库枚举(Chemical Library Enumeration 4)改编而来。
2.1 工具与材料
- KNIME: http://www.knime.org
- RDKit的KNIME组件:见KNIME
- 试剂库: reagent.sdf
2.2 预先知识
了解Daylight的SMILES、SMARTS与SMIRKS语言,以及初步的KNIME使用方法。
2.3 工作流

图4. 基于反应的工作流
工作流下载:reaction-based-library-demo.knwf
2.4 工作流中关键节点的解释
- 读入试剂库
- 设定使用的试剂数量
- 羧酸与伯胺的过滤
- 羧酸与伯胺反应生成酰胺
- 将结果保存为SDF文件
图4工作流的第(1)步是用SDF Reader读入试剂库reagent.sdf,这个试剂库里含有210个试剂,各种羧酸与伯胺化合物分别各含100多个。在本教程中,这些羧酸与伯胺要发生图1的反应,生成可能的酰胺。
图4的操作(2)是用来设定采用的羧酸、伯胺的数量,如果100个羧酸与100个伯胺反应会获得10000个化合物,这本教程中,我们不需要这么多化合物。操作(2)的目的是演示如何控制采用的试剂数量。
图4的操作(3)是用来提取试剂库中的羧酸与伯胺。SMARTS语言羧酸为“[CX3](=O)[OX2H1]”、伯胺为“[NX3;H2,H1;!$(NC=O)]”,用RDKIT的Substructure可以根据SMART语言将这两类化合物提取出来。
SMARTS语言结合其原子定位可以精确描述反应(图5)。比如,羧酸与伯胺反应生成酰胺可以描述为:
1 | [#7;!H0;!$([#7]-C=[O,N,S]);!$([#7]~[!#6]):1].[C:2](=[O:3])-[O;H,-]>>[#7:1]-[C:2]=[O:3] |
RDKIT的RDKit Two Component Reaction(图4步骤4)可以实现这个反应。

图5. SMARTS编码的反应
更多的反应类型编码可以参考Hartenfeller(2012)等人的工作5,在该文的SI中中分享了50种常见反应,采用Reaction-MQL编写。该文虽然不是用SMARTS语言编写反应,但是对如何精准地定义子结构很有帮助。比如,你会从中获得如何定义仲胺的启发,从而更快、更好地利用SMARTS语言。
最后,用SDF Writer节点(图4操作5)将结果保存为SDF文件。
三. 小结
本文演示了如何用SMARTS语言编码羧基、伯胺以及它们之间的反应,并借助化学信息学软件包RDKit在KNIME界面实现基团识别、化合物过滤以及进行双组份的化学反应,最后生成组合库并保存为SDF格式的文件。
四. 从这里开始可以做什么?
有了化合物库,可以接着做什么事?
- 用分子对接进行虚拟筛选
- 用药效团技术进行虚拟筛选
- 用分子形状、静电技术进行虚拟筛选
- 用QSAR模型或其它模型预测化合物的活性
五. 应用
1. 枚举组合库与活性预测、微流控辅助合成联用
组合库设计可以枚举产物获得组合库,结合活性预测与微流控辅助合成实现自动药物发现6。
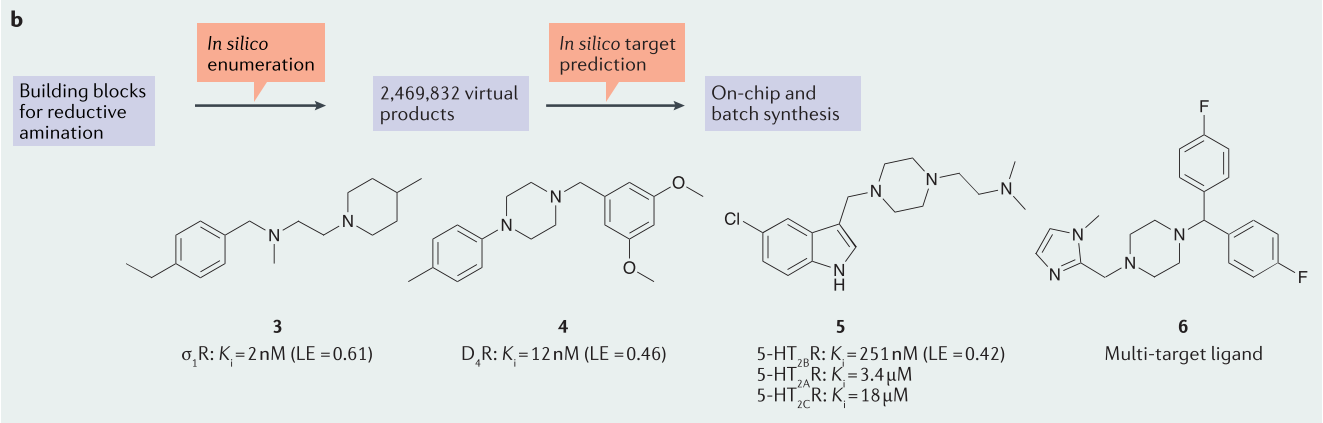
图6 集中库设计案例6:整合组合库设计、活性预测以及微流控辅助合成技术
2. 大规模枚举组合库发现高活性化合物7
Jiankun lyu等人7通过160多种反应枚举出大规模的组合库,通过虚拟筛选、合成、生物活性验证发现高活性化合物。这些化合物是原先ZINC里没有也购买不到的。
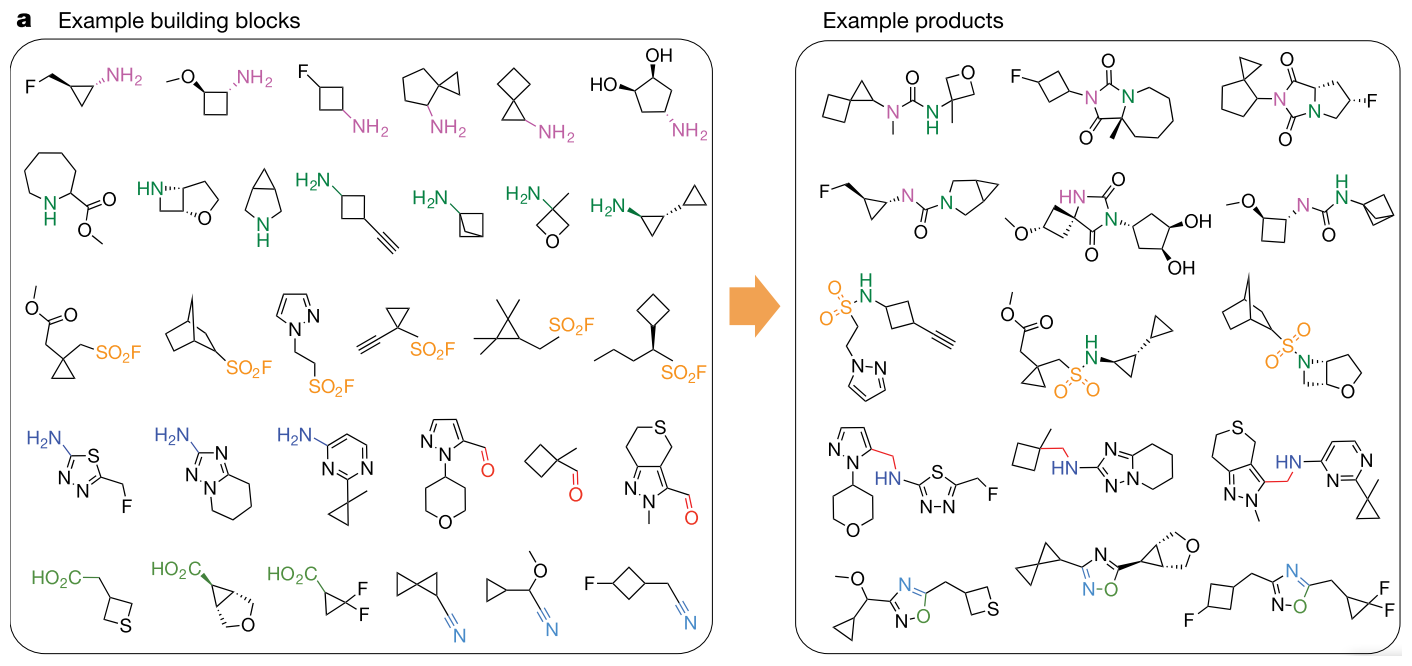
图7 超过规模的组合库枚举进行虚拟筛选发现高活性化合物
六. 从chem-space获得中间体(building block)创建JAK3共价抑制剂集中库
很多网站提供了类药化合物,而不提供building block等试剂,但是试剂对组合库的设计至关重要。我向大家推荐www.chem-space.com:不仅有类药、类先导化合物可以下载,而且有building block可以下载。
以Thorarensen等人8JAK3抑制剂PF-06651600的发现过程中合成路线为例,其中反应式如下图8:
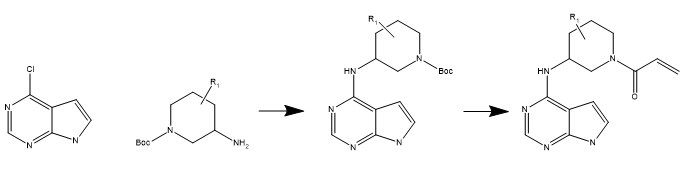
图8. 系列化合物的合成路线
Boc-保护哌啶衍生物是关键的中间体。我们在Chem-Space上试试看能有什么样的此类中间体可供购买,这可以用子结构检索(图9)。
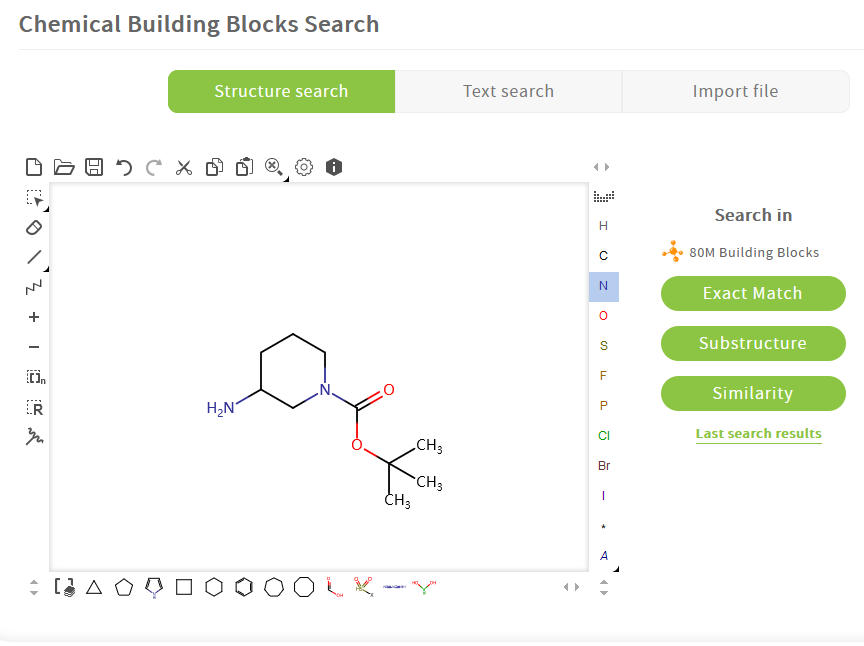
图9. 关键试剂的子结构检索
检索结果的首页如下图10。检索完成,我们得到198个中间体,先Mark All,再点击export to file,然后下载sdf格式文件。
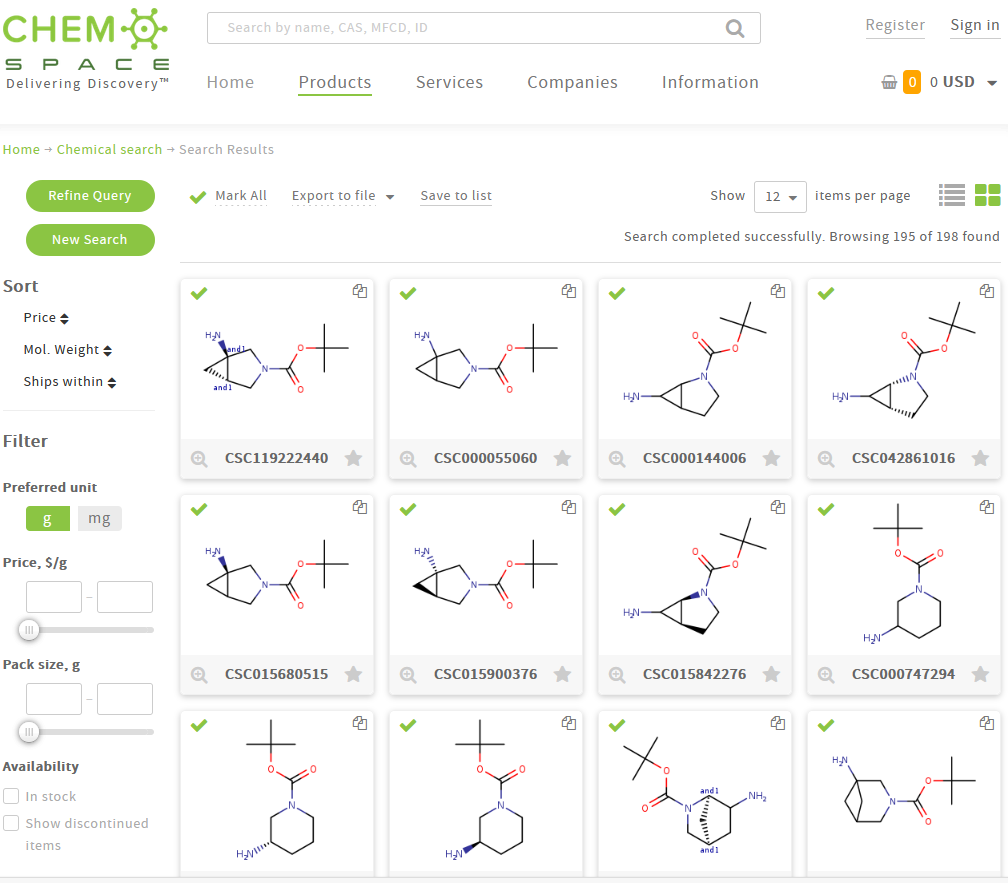
图10. 子结构检索的首页
检索到的结果文件:chem-space-jak3-linker.sdf
用knime对下载到的sdf进行去重复,得到185个不重复试剂(实际上有同一个试剂被枚举手性后得到多个对映异构体的情况)。根据图8反应,可以得到185个化合物:jak3-results.smi。
接下来用OpenEye的分子对接软件FRED对这185个化合物进行共价抑制剂虚拟筛选,详细过程见:用常规分子对接进行共价抑制剂的虚拟筛选,最后命中37个化合物(见表2)。其中部分化合物已有文献报道,但还有很大一部分打分好的化合物未见文献报道。
表1. 37个命中的化合物结构
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
七. 文献
- Dave. SMILES Tutorial – A full SMILES Language Tutorial. 2018-07-25访问:http://www.daylight.com/meetings/summerschool98/course/dave/smiles-intro.html
- SMARTS Examples. 2018-07-25访问:http://www.daylight.com/dayhtml_tutorials/languages/smarts/smarts_examples.html
- SMIRKS – A Reaction Transform Language. 2018-07-25访问:http://www.daylight.com/dayhtml/doc/theory/theory.smirks.html
- Chemical Library Enumeration. 2018-07-25访问:https://www.knime.com/knime-applications/chemical-library-enumeration
- Hartenfeller, M.; Zettl, H.; Walter, M.; Rupp, M.; Reisen, F.; Proschak, E.; Weggen, S.; Stark, H.; Schneider, G. DOGS: Reaction-Driven de Novo Design of Bioactive Compounds. PLOS Comput. Biol. 2012, 8 (2), e1002380.
- Schneider, G. Automating Drug Discovery. Nat. Rev. Drug Discov. 2017, 17:97.
- Lyu, J.; Wang, S.; Balius, T. E.; Singh, I.; Levit, A.; Moroz, Y. S.; O’Meara, M. J.; Che, T.; Algaa, E.; Tolmachova, K.; et al. Ultra-Large Library Docking for Discovering New Chemotypes. Nature 2019, 566 (7743), 224–229. https://doi.org/10.1038/s41586-019-0917-9.
- Thorarensen, A.; et al. Design of a Janus Kinase 3 (JAK3) Specific Inhibitor 1-((2 S ,5 R )-5-((7 H -Pyrrolo[2,3- d ]Pyrimidin-4-Yl)Amino)-2-Methylpiperidin-1-Yl)Prop-2-En-1-One (PF-06651600) Allowing for the Interrogation of JAK3 Signaling in Humans. J. Med. Chem. 2017, 60 (5), 1971–1993.
八. 联系我们
我们提供基于反应与基于母核的组合库设计服务,根据您对试剂的要求与反应路线为您枚举组合库,欢迎咨询。



