摘要:静电互补性打分(EC Score)的基本思想是:当配体和受体的静电势匹配时(即具有相同的量值和相反的符号),实现配体和受体之间的最大静电亲和力。本文详细解释了如何使用静电互补性打分进行分子设计。EC score的最大价值不在于给出分值,而在于EC方法的可视化分析帮你指出在配体哪些位置可以进行改进以提高结合亲和力。
Flare™V2引入了一种称为静电互补性打分(Electrostatic Complementarity score,EC score)的分析方法。基本思想非常简单:当配体和受体的静电势匹配(即具有相同的量值和相反的符号)时,实现配体和受体之间的最大静电亲和力。乍看之下,这似乎是显而易见的;再看之下,这似乎有点令人惊讶——让配体具有更大的潜力以使得相互作用能更好难道不是一个好主意吗?原因在于配体与蛋白质之间改进的静电相互作用能量抵消了配体去溶剂化惩罚的增加。
因此,我们所需要做的就是计算配体和蛋白在合适的接触表面上的静电势,然后计算某种相关度量来测量它们有多相似。在真空中,这种计算相当直接。不幸的是,像往常一样水使一切变得更加复杂。在没有运行长时间的分子动力学模拟情况下,我们不得不以某种方式近似处理溶剂效应。我不太相信连续介质溶剂近似可以达到这个目的,因为蛋白活性部位及其周围的水远远不是连续的电介质。然而,我们必须做些事情来解决水的问题。我们的答案混合使用将复杂的介电函数和对形式电荷的特殊处理,我们已经证明这对于可视化蛋白质活性位点内的静电势是很有效的(参见前面关于蛋白质相互作用电势的博客文章)。
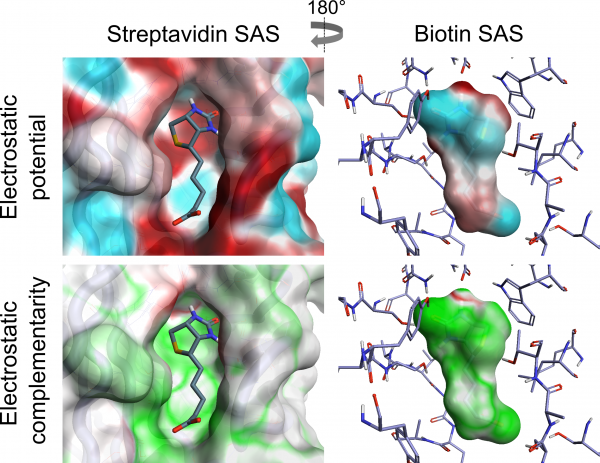
Figure 1. 生物素与链霉亲和素复合物的静电势与分子表面互补性
因此,我们计算静电势1, 通过互补性对表面着色来以可视化它们(Figure 1),并且我们可以计算总EC分数。现在的问题是:“它真的做了什么有用的事了吗?”
我们计算的是整体的EC分数,所以很明显要检查的是分数是否与活性相关。我们已经测试过很多数据集(参见Figure 2和前面提到的J Med Chem论文1),您可以得到从中等程度(RPA70N的r2=0.33)到非常好的(PERK的r2=0.79)相关性。问题解决了,那么:只要将您设计的配体对接到蛋白结合位点里,然后计算EC分数,并选择一个EC打分最高的配体,合成它!
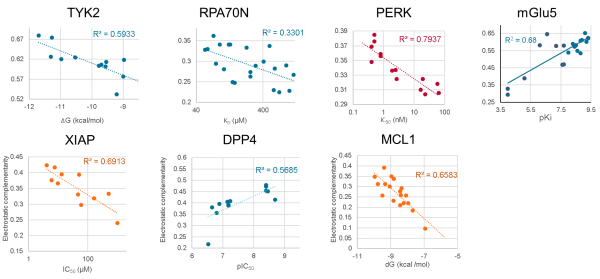
Figure 2: 一系列数据集的EC打分值与活性的相关性
不幸的是,其实并不是那么简单。虽然我们确实在多种数据集的多个靶标上证明EC分数与活性相关,但是这些数据集被非常仔细地准备过。原因是配体与蛋白的结合取决于许多不同的物理效应。静电是其中之一,也是非常重要的一个,但它不是唯一的一个,所以EC分数只会预测其他效应不变时的活性差异。
用于获得Figure 2中相关性的数据集非常保守:每个数据集中的配体都紧密相关,它们的大小近乎相同,具有大致相同数量的可旋转键,具有一致的结合模式,等等。此外,我们还发现为了获得强相关性,需要让叠合带来的噪音最小化(就像生成一个好的3d-qsar时一样),所以我们将所有的配体按公共子结构进行叠合,而不是依靠完全自动的分子对接。
如果所有其它的性质都一样的,那么EC分数更高的分子应该有更高的亲和力。不幸的是,在现实世界中,其他性质很少是相同的,因此除非你看到相当保守的变化(例如,问我:配体上哪里可以用氟取代以提高亲和力),否则EC分数很可能是一个糟糕的指南。回到最初的问题:”它真的做了什么有用的事了吗?”
幸运的是,虽然单个数值型EC分数对分子在活性位点中的放置非常敏感,但是EC值在表面上的分布要稳健得多。 EC方法的主要用途不是计算分数:它的价值在于可视化分子在哪里与蛋白的静电很好地匹配以及在哪里与之不匹配(Figure 3)。这为您提供了一些提示,提示您对分子的何处进行更改、以及您进行什么样的更改:添加卤素? 移动杂环中氮原子的位置?卤素原子或芳环边缘细小的静电相互作用很难用其它方式可视化。
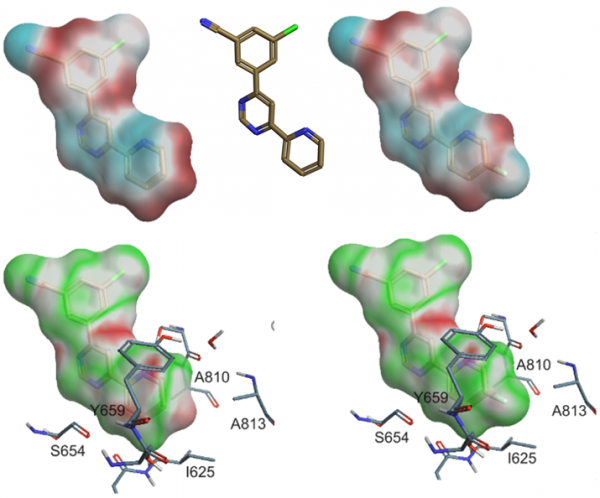
Figure 3.如图左侧的EC表面着色所示,MGLU5抑制剂在吡啶环上具有较小的静电冲突。如图右侧所示,在这个位置放上氟则可以消除静电冲突并提高亲和力。
然后,EC方法的主要用途是分析您的配体并指出哪里可以进行改进。您可以确信这些建议是合理的,因为我们已经在多个数据集中证明这一点,在配体之间的差异主要是静电的情况下,EC分数与亲和力相关。然而,EC分数本身并不是亲和力的一般预测因子,因为有许多因素不包括在分数中,这些因素可以使分子成为更好或更差的结合剂。
如果你想观察分子在活性位点的静电特征,并获得如何改进它们的建议,我们可以提供Flare的免费试用。
文献
- Bauer, M. R.; Mackey, M. D. Electrostatic Complementarity as a Fast and Effective Tool to Optimize Binding and Selectivity of Protein-Ligand Complexes. J. Med. Chem. 2019, acs.jmedchem.8b01925. https://doi.org/10.1021/acs.jmedchem.8b01925.



