摘要:小柱孢酮脱水酶(Scytalone Dehydratase,SD)抑制剂可以保护水稻免受稻瘟病菌引起的病害。本文以相差仅一个腈基的SD抑制剂6d与7d为例,综合利用蛋白相互作用势、配体场与GIST水热力学性质分析,来解释两者活性差异18000多倍的原因。
肖高铿/2021-09-10
1. 前言
小柱孢酮脱水酶(Scytalone Dehydratase,SD)是稻瘟病菌引起的水稻真菌病害的一个关键分子靶标,SD抑制剂可以保护水稻免受稻瘟病菌引起的病害。
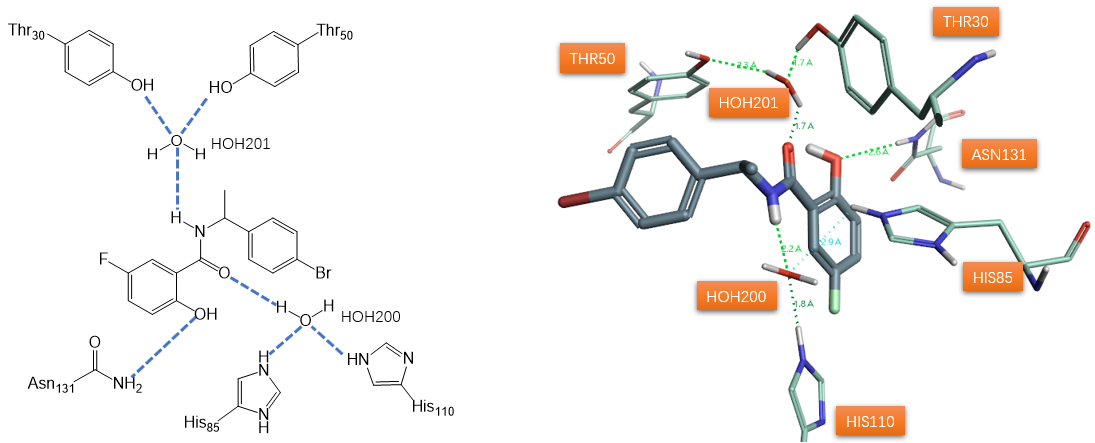
图1. 水杨酰胺类SD抑制剂1a及与其相互作用的关键水分子,PDB 1STD
杜邦公司报道了一个以水杨酰胺类抑制剂1a(图1)与SD共晶结构(PDB 1STD)为起点的SD抑制剂开发项目[1]。抑制剂1a除了与蛋白质侧链(未示出)发生许多疏水相互作用外,还与蛋白质、以及两个结合水分子形成了氢键网络(图1)。该氢键网络可用来解释抑制剂结合亲和力的强弱。然而,活性位点残基、水分子和抑制剂之间的氢键应被认为具有动态特性。其中一个结晶水分子(HOH200)与水杨酰胺羰基氧原子和酪氨酸30和50的酚羟基形成氢键。假设该水分子作为普通的酸与两个酪氨酸残基结合以诱导底物scytalone烯醇化。第二个水分子(HOH201)与抑制剂1a的NH 、HIS85 、HIS110 的epsilon位氮原子形成氢键,被认为是SD催化循环中脱水生成的水。
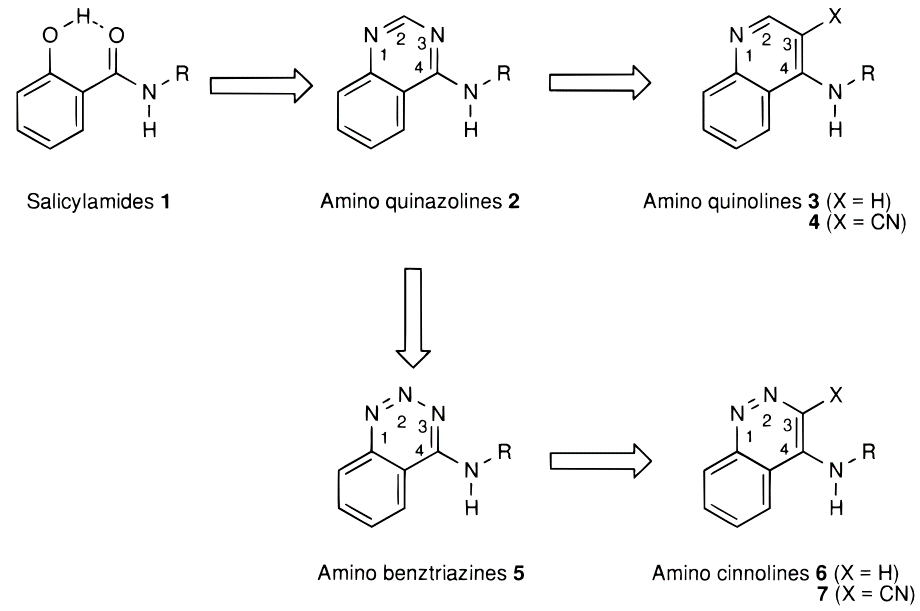
图2. SD抑制剂的设计与发展过程(图片来源于文献[1])
有理由相信,如果用生物等排体喹唑啉杂环替换1a的水杨酰胺部分,将保持与两个水分子的氢键网络。喹唑啉杂环是对1a水杨酰胺片段的分子内氢键的模拟环化而来,其对SD催化活性的抑制能力已经有文献公开。预期喹唑啉环的1位氮原子(序号见图2)将占据1a羟基的位置以从Asn131的侧链接收氢键。之后的喹唑啉类抑制剂与SD的共晶结构PDB 5STD的解释证实了这个猜测:这两个水分子(图3 HOH537与538)确实还保留着,通过氢键介导着抑制剂与蛋白之间的相互作用。重要的是,在氨基上需要合适的大体积疏水取代基来占据衬有许多疏水氨基酸侧链的空腔,以便实现有效的抑制。作者认为,稠合杂环可以作为合适的骨架,用于引入官能团以取代活性位点水分子之一。
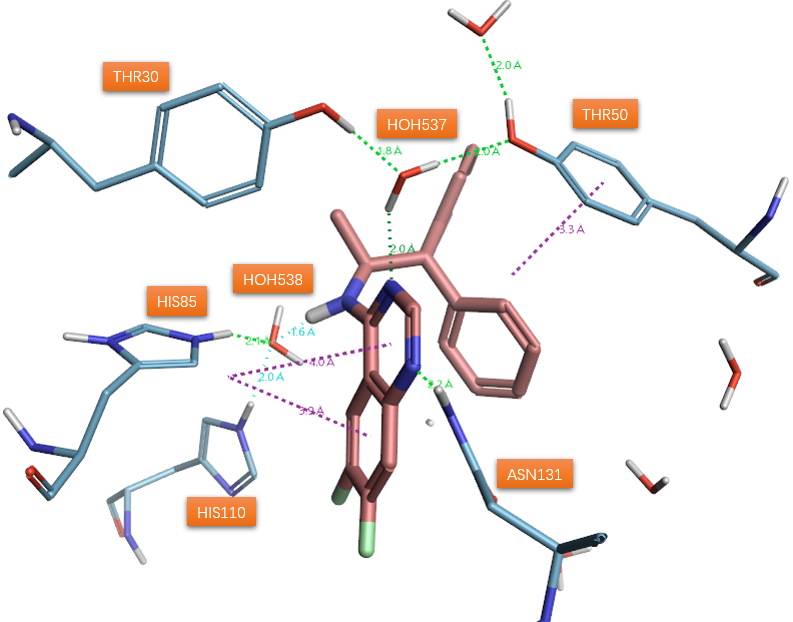
图3. 喹唑啉环抑制剂与SD共晶结构(PDB 5STD)揭示的相互作用模式
通过置换这种水分子来获得在结合亲和力上的增益已在文献中得到了充分认可。首先,将水分子从受限环境中释放到主体溶剂中,熵增加。其次,有可能与协调水分子的蛋白残基直接结合、相互作用。第二点是重要的,因为用于结合水分子的空间和静电环境必须对系统的总自由能提供比由于限制水分子而造成的熵损失更大的焓贡献。否则,水分子就不会受到这样的限制。如果抑制剂类似物被设计成取代结合的水分子,则由于水分子而导致的焓稳定性的损失应当部分地通过来自设计的抑制剂的焓贡献来重新获得。
作者设计的SD抑制剂的演变过程如图2所示,这里我重点以其中一对化合物6d与7d(图4)为例来说明结合水分子替换所代带的巨大结合亲和力(18182倍)收益。Michel等人[2]已经以6d、7d为算例对其中的水分子替换进行了详细的理论计算解释,而本文的主要目的是尝试从分子可视化、配体场、蛋白分子相互作用势、GIST溶剂热力学分析等角度来解释水分子替换所带来的结合亲和力增益。
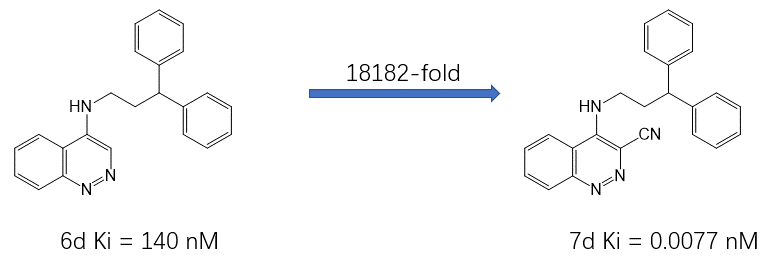
图4. 化合物6d与7d的化学结构式及其Ki值
2. 方法
2.1 蛋白的结构准备
将7d与SD的共晶结构(PDB 3STD)从蛋白质数据库下载到Flare中,并使用来自Protein Prep工具小心地准备,以添加氢原子、优化氢键、消除原子冲突并给蛋白结构分配最佳质子化状态。任何截短的蛋白质链被封端作为蛋白质准备的一部分。7d结构从共晶结构3STD中提取出来,而6d结构是根据7d的生物活性构象在结合位点里删除腈基得到。
2.2 GIST分析
了解蛋白活性位点内水分子的行为是药物设计的一个非常重要的方面。在蛋白质-配体复合物中,了解桥连水分子的稳定性是决定药物设计策略的关键。如果水分子不是特别稳定,可以设计配体以取代它,并与蛋白质活性位点直接相互作用:这通常会导致配体生物活性的增加。
GIST是一种水热力学性质分析方法[3],用于评估结合口袋的水合作用,并通过在分子动力学运行结束时对显式溶剂分布采样来计算相关的水热力学性质。GIST分析的结果显示为活性位点水合作用的三维等值面映射“happy”(绿色,与负∆G相关)和“unhappy”(红色,与正∆G相关)区域。unhappy区域映射的是蛋白质活性位点内更可能的可成药区域。happy区域映射的是蛋白结合位点内较低可能成药的区域,在那里水分子更稳定,因此更难置换。
在本文中,用Flare分别对结合位点的Apo结构(不包含配体、金属离子与结晶水)进行了GIST分析,使用了如下条件:
- Calculation method: Normal
- Ligand: None
- Grid spacing:0.5 Å
- Grid Definition:Ligand
- Chains: A
- Simulation length: 20ns
- Solvent Model: explicit TIP4Pew Water
2.3 蛋白相互作用势分析(PIP)
蛋白相互作用势(Protein Interaction Potential, PIP)是Cresset分子相互作用势对蛋白质的延伸,两者都是使用XED力场计算的。该方法在原理上类似于配体场的计算:蛋白质的活性位点被探针原子充满,并且计算每个格点上的相互作用势。该方法利用了Mehler等人[4]的距离依赖的介电函数来更好地处理蛋白质结构中的大量带电基团。仅计算和显示活性位点的蛋白质相互作用电势。
2.4 静电互补性分析
Flare引入了一种称为静电互补性打分(Electrostatic Complementarity score,EC score)的分析方法[5],它将配体静电的分析与蛋白静电的分析结合起来,以产生配体-蛋白质复合物的静电互补性(EC)的视觉评估和数值评估。基本思想非常简单:当配体和受体的静电势匹配(即具有相同的量值和相反的符号)时,实现配体和受体之间的最大静电亲和力。通过将该方法应用于一系列文献数据集,分析其视觉和数值分量,表明EC与报道的生物活性差异相关,并能够预测报道的生物活性差异[5]。我们用Flare的EC技术对化合物进行静电互补性比较。
2.5 配体场
Cresset XED力场[6,7]通过复杂的原子描述来模拟远离原子中心的电荷,从而改进了传统的分子力学。这使得能够更详细地描述静电并优异地重现分子间相互作用。XED力场由Andy Vinter博士开发,在Cresset改进,它正确地模拟了取代基对芳香族化合物的效应、复杂芳香族化合物中电荷密度的变化以及小分子、水和蛋白质之间的分子间相互作用。在本文中,我门用XED力场来描述配体的静电性质。
3. 理解6d与7d的活性差异
3.1 经典的蛋白-配体相互作用分析与分子对接分析
首先用Ligandscout 4.4分析共晶结构PDB 3STD中蛋白与配体7d的相互作用,如图5所示。可以发现,两个大体积亲脂性基团被疏水氨基酸包围,这正是作者想要的。模仿水杨酰胺片段的噌啉片段氮原子作为氢键受体与ASN131相互作用。噌啉环上的氨基做为氢键供体保留了与结合水HOH517的相互作用。最重要的是,噌啉环上的腈基作为氢键受体代替了原先的结合水与TYR30、TYR50发现氢键相互作用。
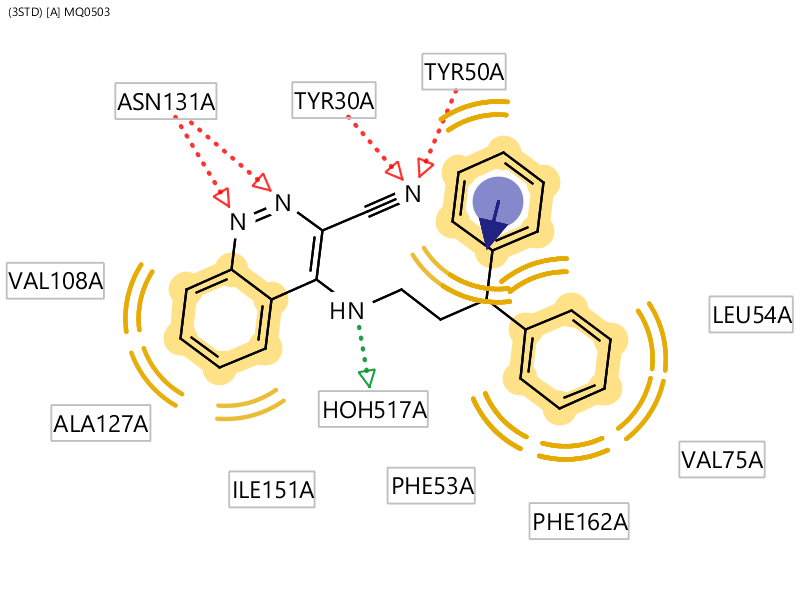
图5. 共晶结构PDB 3STD的蛋白-配体7d相互作用分析(没有呈现全部的π-π相互作用)。红色:氢键受体;绿色:氢键供体;黄色:疏水中心。
视频1则重点呈现了氢键与π-π相互作用,以及相关的氢键网络相互作用。注意到,噌啉环上的氨基与HOH517氢键相互作用的几何并不完美,应该以动态的观点去理解该水的实际作用方式:其氢的取向是可以变化的。但总的来说,HOH517介导的氢键网络还如1STD中的HOH201那样。可以看到7d的腈基做为氢键供体模拟了水分子直接与TRY30、TYR50的酚羟基发生氢键相互作用,这个相互作用是6d与7d之间在经典相互作用模式上的唯一差别。
视频1. 7d与SD共晶结构的相互作用分析(疏水相互作用未呈现)。绿色虚线:氢键;紫色虚线:π-π或阳离子-π。
接着在”干”蛋白结合位点(即将所有的结晶水都删除掉)里用AutoDock Vina打分函数评价6d、7d的结合亲和力,结果如表1所示。可以看到,两者的疏水贡献一样且都非常大,这与分子大体积疏水基团特征一致;6d比7d的氢键相互作用贡献少了1.8,这主要是由于6d少了腈基而不能与TRY30、50发生氢键相互作用。然而,Vina score打分并没有因为氢键作用的差异而将6d与7d区分开来。总的来说,Vina打分不能解释6d与7d在活性上高达18000多倍的差异。这再次提醒我们,分子对接打分函数并非为预测结合亲和力而设计,尤其是在先导化合物优化阶段应谨慎使用虚拟筛选打分函数来对分子进行优先性排序。
表1. 用AutoDock Vina打分函数预测化合物6d与7d的结合亲和力
| Comp ID | PDB ID | Vina Score(kcal/mol) | Hydrophobic | H-Bond |
|---|---|---|---|---|
| 6d | 3STD | -12.17 | 135.55 | 0.60 |
| 7d | 3STD | -13.09 | 135.55 | 2.41 |
Vina输入文件见附件1。
3.2 蛋白相互作用势与静电互补性分析
蛋白相互作用势可以快速地从蛋白角度定性地考察SAR、分析蛋白-配体的静电互补性。图6呈现了3STD结合位点的蛋白相互作用势,可以发现,7d的腈基被一个红色区块(正蛋白相互作用势)包围而凸显出来,这提示应该在配体上引入静电上互补的基团以提高蛋白-配体相互作用。还可以看到,噌啉环氮原子附近的蛋白相互作用势也为正,这是因为蛋白ASN131酰胺做为氢键供体朝向噌啉环而带来的。
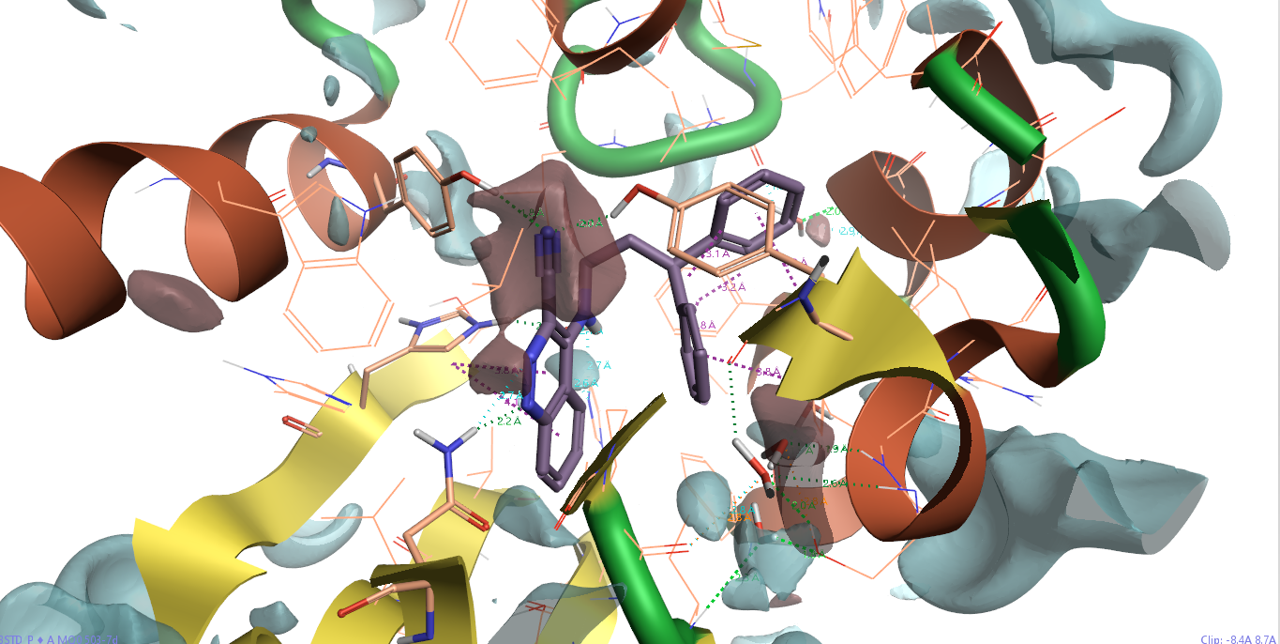
图6. 共晶结构PDB 3STD的蛋白相互作用势分析。红色区块:正蛋白相互作用势(eV=13等值图);蓝色:负蛋白相互作用势(eV=-8等值图)。
THR30、50酚羟基附近的正蛋白相互作用势与6d红色的正配体场(见图7左)发生冲突,而与7d蓝色的负配体场(见图7右)互补。6d与7d在噌啉环氮附近均为负配体场与正蛋白相互作用势互补。
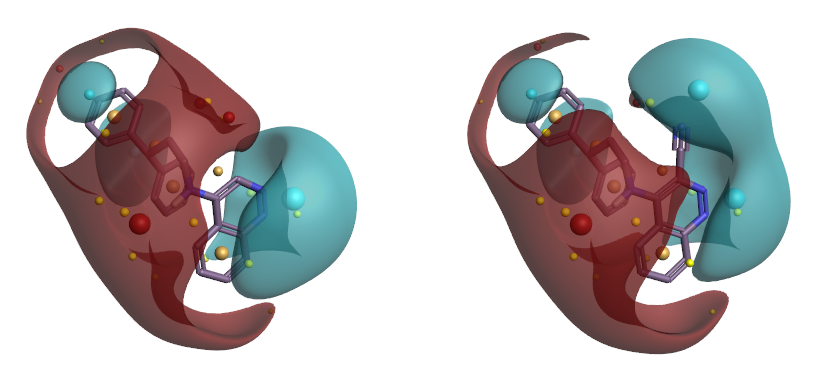
图7. 6d(左)与7d(右)的配体场。红色:正配体场;蓝色:负配体场
将分子表面用静电互补性(EC)打分值着色可以直观地考察EC值在表面上的分布,它的价值在于可视化分子在哪里与蛋白的静电很好地匹配以及在哪里与之不匹配。图8比较了化合物6d与7d分子表面的EC分布,鉴于两个分子仅在一处有差异,轻而易举地凸显出6d被腈基取代后的对应区域由红色转为绿色,这说明腈基取代是静电有利的,静电相互作用差异可是两者活性差异的主要原因之一。
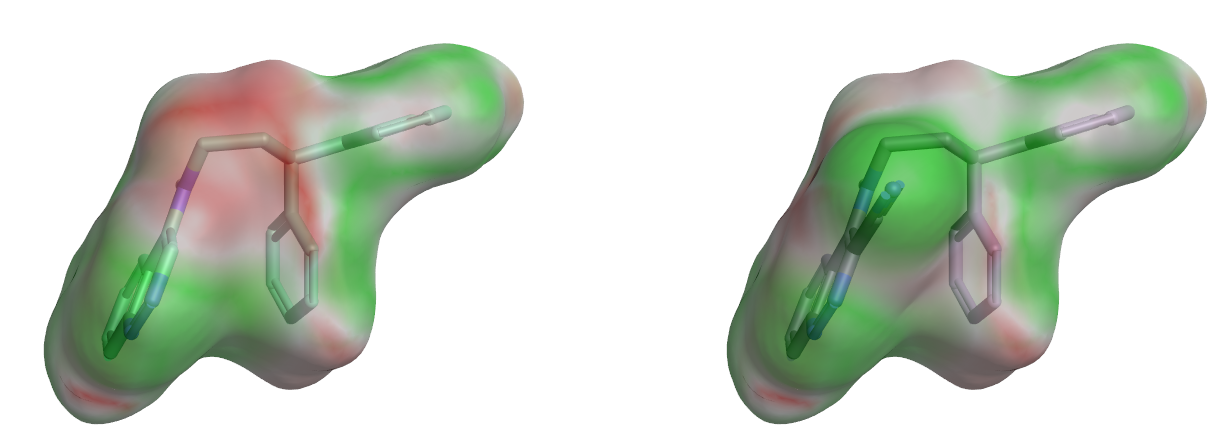
图8. 6d(左)与7d(右)分子表面静电互补性分析。红色:静电冲突;绿色:静电互补。
3.3 GIST分析
理想的配体,不仅要在形状、静电上与蛋白结合位点互补,而且还要与蛋白结合位点的溶剂热力学性质相匹配。用Flare/GIST分析PDB 3STD结合位点,结果如图9所示。可以发现结合位点基本被红色的区块占据,这说明该结合位点具有良好的成药性。同时我们发现,腈基所在的位置被一个红色的区块覆盖,说明对应位置的水是可置换的。
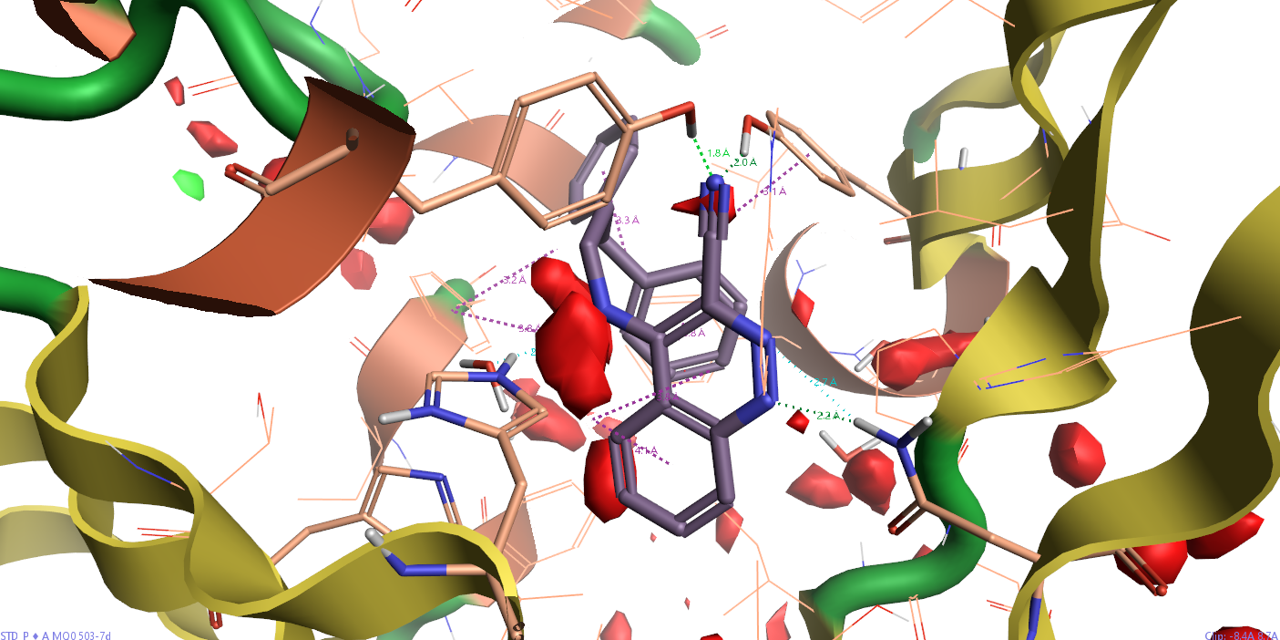
图9. PDB 3STD结合位点Apo结构(不包含溶剂、配体、金属等)的GIST分析结果。红色:ΔG=+0.5kcal/mol水平等值图;绿色:ΔG=-0.5kcal/mol水平等值图。
为了凸显能量高的重要的水,绘制了ΔG=+1.0kcal/mol与ΔG=+3.0kcal/mol水平网格状等值图,如图10所示,分别着色为粉红色与青灰色。可以发现腈基上没有对应的等值图出现,这说明腈基置换水而带来的结合亲和力增益(因为水置换相关的热力学增益)并不是很大,可能不是6d与7d活性差异的主要原因。
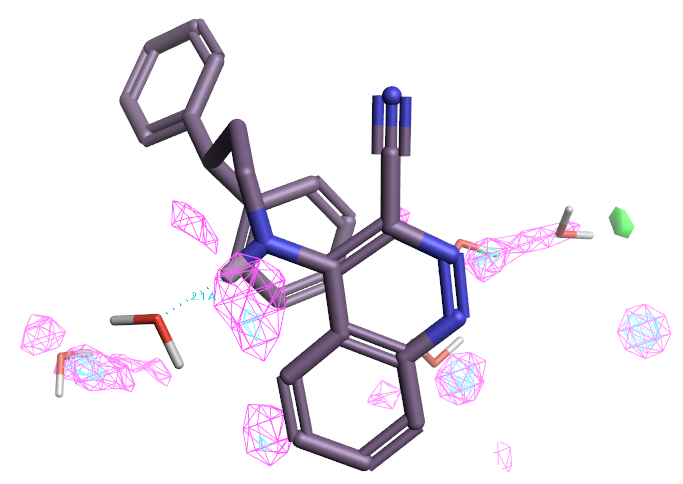
图10. PDB 3STD结合位点Apo结构(不包含溶剂、配体、金属等)的GIST分析结果。粉红色:ΔG=+1.0kcal/mol水平等值图;青灰色:ΔG=+3.0kcal/mol水平等值图。
有意思的是,用水分子替换策略来源的共晶结构PDB 5STD进行的GIST分析却给出略有差异的结果。视频2演示了PDB 5STD结合位点Apo结构的GIST分析结果。红色网格为ΔG=+0.5kcal/mol水平等值图,绿色网格为ΔG=-0.5kcal/mol水平等值图。
视频2. 共晶结构5STD结合位点Apo结构的GIST分析结果。红色:ΔG=+0.5kcal/mol水平等值图;绿色:ΔG=-0.5kcal/mol水平等值图。
可以发现,视频2呈现的ΔG=+0.5kcal/mol水平等值图与PDB 3STD分析的基本一致。所不同的是,若将等值图提高到ΔG=+3.0kcal/mol(图11粉红色等值图)与ΔG=+5.0kcal/mol(图11青灰色等值图)的时候,5STD的TYR30、50侧链酚羟基附近的HOH537(图11)依旧有等值图覆盖,在粉红色等值图里有一小点青灰色。也就是说,HOH537位置处的ΔG至少为+5.0kcal/mol,这是一个非常重要的水。
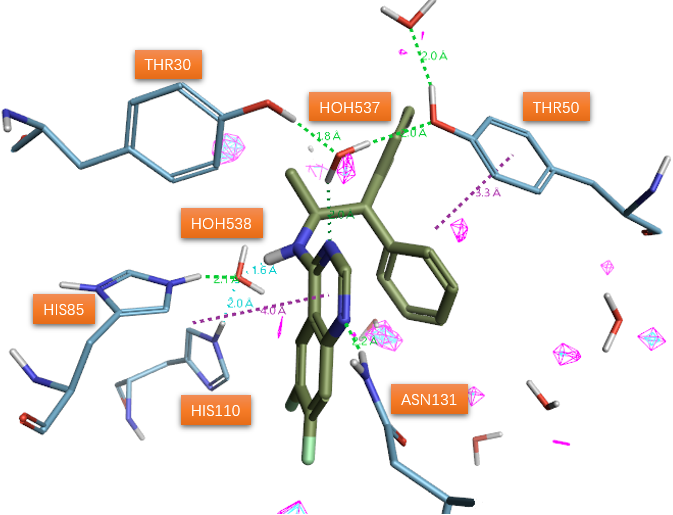
图11. PDB 5STD结合位点Apo结构的GIST分析结果。粉红色:ΔG=+3.0kcal/mol水平等值图;青灰色:ΔG=+5.0kcal/mol水平等值图。
虽然根据PDB 3STD与5STD对同一个位置计算的水合自由能存在差异,但无论如何,GIST分析都提示了在Tyr30、50侧链酚羟基附近重要的可替换的水合位点。综合之前的蛋白相互作用势分析,可以知道这个水合位点应该用极性的基团对水进行替换,并且该位点PIP呈强静电势,应该引入一个带负静电势的基团做为配体的一部分。对6d的配体场与表面互补性分析则给出了配体在静电上不匹配的位置。综合这些信息,有助于药化专家设计正确的配体,或者借助SPARK虚拟筛选而发现活性更高的化合物。
4.小结
总的来说,GIST水热力学分析可以确认可被替换水的位置;蛋白相互作用势(PIP)分析可以确认可替换水位置的蛋白静电特性,同时也定义对与之互补的配体在静电场性质方面的要求;PIP结合配体场可以进一步确定对用来替换该水的配体片段的性质要求;静电互补性(EC)分析可进一步考察蛋白与配体在静电上是否冲突确认修饰位置与方向。综合这些关键的SAR信息,可以凸显出配体上重要的药化修饰位置、指导新分子的设计。
根据初步的SAR信息,可以初步判断通过引入腈基来引入新的相互作用是7d相比6d活性得以提高18000多倍的主要原因,水分子替换的热力学贡献是次要原因。用杜邦公司作者的原话来总结就是:首先,将水分子从受限环境中释放到主体溶剂中,熵增加。其次,与协调水分子的蛋白残基直接结合、相互作用。第二点是重要的…。
5. 附件
- AutoDock Vina输入文件:sd_vina.zip
使用方法,以7d化合物为例:
1 2 3 4 5 6 7 8 9 | vina --receptor 3std_prot_h.pdbqt \ --ligand 7d.pdbqt \ --center_x 29.040 \ --center_y 37.456 \ --center_z 23.104 \ --size_x 40 \ --size_y 40 \ --size_z 40 \ --score_only |
6. 文献
- Chen, J. M.; Xu, S. L.; Wawrzak, Z.; Basarab, G. S.; Jordan, D. B. Structure-Based Design of Potent Inhibitors of Scytalone Dehydratase: Displacement of a Water Molecule from the Active Site. Biochemistry 1998, 37 (51), 17735–17744. https://doi.org/10.1021/bi981848r.
- Michel, J.; Tirado-Rives, J.; Jorgensen, W. L. Energetics of Displacing Water Molecules from Protein Binding Sites: Consequences for Ligand Optimization. J. Am. Chem. Soc. 2009, 131 (42), 15403–15411. https://doi.org/10.1021/ja906058w.
- Nguyen, C.; Gilson, M. K.; Young, T. Structure and Thermodynamics of Molecular Hydration via Grid Inhomogeneous Solvation Theory. 2011. arXiv:1108.4876v1. https://arxiv.org/abs/1108.4876
- E. L. Mehler, The Lorentz-Debye-Sack theory and dielectric screening of electrostatic effects in proteins and nucleic acids, in Molecular Electrostatic Potentials: Concepts and Applications, Theoretical and Computational Chemistry Vol. 3, 1996
- Bauer, M. R.; Mackey, M. D. Electrostatic Complementarity as a Fast and Effective Tool to Optimize Binding and Selectivity of Protein–Ligand Complexes. J. Med. Chem. 2019, 62 (6), 3036–3050. https://doi.org/10.1021/acs.jmedchem.8b01925.
- Vinter, J. G. Extended Electron Distributions Applied to the Molecular Mechanics of Some Intermolecular Interactions. II. Organic Complexes. J. Comput. Aided. Mol. Des. 1996, 10 (5), 417–426. https://doi.org/10.1007/BF00124473.
- Cresset的核心技术. 墨灵格的博客. http://blog.molcalx.com.cn/2019/10/08/cresset-science-xed.html



