摘要:在本文中,演示了如何读入一个SDF分子结构,用Vina将其docking到一个蛋白结合位点里,然后试图将对接结果用模板法进行键级与电荷归属,但是发现键级归属失败。因此,尝试使用OEChem的键级与电荷感知函数,从对接的坐标开始,识别分子的连接性、键级与电荷,识别出的结构可视化结果表明,获得了正确的结构,键级与电荷与输入的分子完全一致。
前言
随着机器学习与各种开源软件的流行,大家不可避免的遇到很多基础的化学信息学问题。以流行的分子对接Vina为例,它最大的问题在于对接前的小分子结构准备、将其它格式的小分子结构转换为PDBQT格式、以及对接后将PDBQT格式转换更方便可视化。尤其在将PDBQT转换为SDF等格式时,经常会出现电荷与键级不能正确归属的问题。比如,前文以LigGrep的数据集为例说明如何对vina结果进行过滤处理时发现作者提供的sdf文件就有很多的键级错误[1]。
ADT、OpenBabel与RDKit是大家常用的VINA分子格式转化工具,但是这些工具并非完美。比如,ADT可以完美地将Mol2,PDB转化为PDBQT,但是不能将PDBQT转化sdf或mol2。而Openbabel可以实现双向转换,但是在将sdf或mol2转为PDBQT的时候,有时存在PDBQT树形结构错误而导致在分子对接计算中某些两面角不能被搜索到而redocking失败,这个错误已经报告在OpenBabel的issue#2433里。虽然OpenBabel在将PDBQT转化为SDF的时候有较高的成功率,但是与LigGrep的算例类似,键级错误的比例还是非常高。Meeko是Scripps研究所VINA 1.2开发团队开发一种新的结构准备工具与格式转换工具,结合使用了RDKit与Openbabel库,解决了PDBQT转SDF等格式的键级问题[2]是该软件的特色之一。但是Meeko也存在与openbabel类似的PDBQT树结构错误而导致同样的re-docking失败,见:issue#7。RKDit并不直接处理PDBQT格式,但是RDKit可以完成很多结构准备工作。徐锡明博士开发的WatVina[3]配套了一个格式转化工具rdkit2pdbqt.py,借助RDKit将小分子SDF转化为PDBQT格式。rdkit2pdbqt.py还提供了一个PDBQT转RDKit分子的函数,这也为大家提供了PDBQT后处理的一种新的便利方法。
RDKit解决PDB或PDBQT读入时的键级问题的方法之一是使用RDKit的AssignBondOrdersFromTemplate。将对接后的分子,按模板重新归属键级。然而在实践中经常还是会有归属异常,本文以一个键级归属异常的分子ZINC08441965为例,尝试用OEChem的键级与电荷感知函数,根据PDBQT的原子坐标生成键级与电荷,以考察能否解决PDBQT分子格式转化的问题。
解决问题的思路
读取Vina对接计算的pose,提取坐标,根据原子的3D坐标用OEChem提供的函数来推断分子的键级与电荷。这几个关键的函数是:
- oechem.OEDetermineConnectivity()
- oechem.OEFindRingAtomsAndBonds()
- oechem.OEPerceiveBondOrders()
- oechem.OEAssignImplicitHydrogens()
- oechem.OEAssignFormalCharges()
演示
算例说明
前期进行测试时候发现基准数据集DEKOIS[4]中VEGF2的decoy有几个化合物根据模板归属键级失败,因此以其中一个失败的化合物ZINC08441965(sdf取自DEKOIS)为例来考察失败的原因与解决方法。
操作过程
用到的软件
OEChem: 是OpenEye的发布的商业软件,需要授权书才能测试。
RDKit: 开源免费的化学信息学工具。
rdkit2pdbqt: 徐锡明博士WatVina[3]的一部分,我认为这是非常棒的一个工具。演示中的pdbqt模块即是rdkit2pdbqt。
结果
与起始的3D结构相比,OEChem识别的出的结构有两处不同:1)起始结构的C=NH亚胺被归属为烯胺-NH2;2)起始结构与亚胺氮相连接的C-C单键被识别为C=C双键,除此之外,其它部分都一致。起始的ZINC08441965.sdf3D结构如下图所示(用鼠标可旋转查看),注意其中平面型的亚胺部分及其旁边的一个与腈基连接的C原子是都是平面型的,但是根据图形却为SP3杂化,这是不合理的!
图1.从DEKOIS的VEGFR2提取到的ZINC08441965三维结构:平面型sp3碳原子上有一个腈基与一个氢原子(略显白色),平面型碳原子几何结构与4个价键的sp3杂化是互相冲突的。
VINA对接之后,根据pose生成的坐标,OEChem得到比起始结构更加合理的结构, 主要理由在于:如下图2所示,之前不合理的平面型sp3杂化C被OEChem修正为平面型的sp2杂化方式,亚胺归属为连接在碳碳双键上的烯胺,因为共轭的关系,氨基平面化了。显而易见,OEChem解释的结构与初始的3D结构完全匹配。
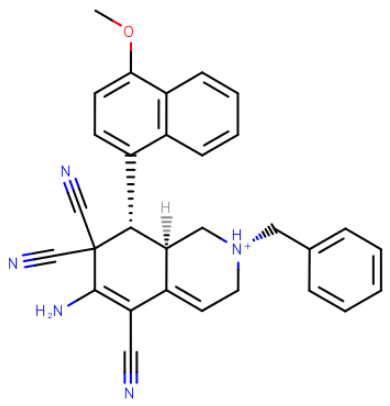
图2.OEChem进行键级与电荷识别之后的2D结构
相反,RDKit不能正确的归属键级与原子类型,主要是因为不合理的起始结构,除了DEKOIS 2.0提供的3D结构不对之外,ZINC数据库提供的3D结构也不对,具体见图3。这本例中,重新准备起始的结构再进行docking,键级归属就没有问题了。无论如何,OEChem可以单独根据坐标而得到合理的结构,无需模板就可以可靠地实现vina docking结果的格式转化。
图3. 从ZINC上下载到的ZINC08441965的3D结构,其3D几何与SMILES所表征的原子类型、键类型并不匹配:腈基sp杂化原子本应该是直线型,C=C双键sp2碳原子本应该是平面型,但是根据3D几何结构更像是sp3杂化。糟糕的起始结构导致后续键级与电荷识别失败:腈基氮可能被识别为氨基,SP2的烯烃碳可能会被识别为sp3的碳。
小结
在本文中,演示了如何读入一个SDF分子结构,利用rdkit2pdbqt将其转化为vina可识别的PDBQT,并将其对接到一个蛋白结合位点里,然后试图将对接结果用模板法进行键级与电荷归属,但是发现RDKit键级归属失败。而使用OEChem的键级与电荷感知函数,从对接的坐标开始,OEChem识别到了正确结构,可视化结果表明,OEChem生成的结构与初始结构基本一致,实际上是更加合理,完美解决了键级与电荷问题。
下一步将会使用DUDE数据集,在较大规模上进行测试,然后向大家分享结果。
文献
- 肖高铿. 分子对接后处理——与指定残基发生特定相互作用配体的过滤. 墨灵格的博客. 2021-11-02. http://blog.molcalx.com.cn/2021/11/02/docking-post-filtering.html
- Meeko. https://github.com/forlilab/Meeko.
- WatVina. https://github.com/biocheming/watvina
- Bauer, M. R.; Ibrahim, T. M.; Vogel, S. M.; Boeckler, F. M. Evaluation and Optimization of Virtual Screening Workflows with DEKOIS 2.0 – A Public Library of Challenging Docking Benchmark Sets. J. Chem. Inf. Model. 2013, 53 (6), 1447–1462. https://doi.org/10.1021/ci400115b.
拓展应用
OEChem强大的相互作用分析不仅可以用于视觉分析,还可以用来对docking结果进行后处理,比如过滤出满足特定相互作用模式的化合物。



