摘要:本文从激酶特性结合子口袋分析、相互作用分析与生物活性构象稳定性分析等三个角度考察了CT7001对CDK7与CDK2激酶选择性的原因。
肖高铿/2022-05-31
前言
CT7001(图1)是CARRICK THERAPEUTICS开发的选择性CDK7抑制剂,此前已经详细地介绍过其体外数据[1]。CT7001对CDK7/CDK2激酶的IC50分别为40与640nM,具有一定的选择性(16倍的差异),并且它们的共晶结构已经公开(PDB代码分别为7B5Q与5JQ5),因此本文的主要目的是从结构生物学提供的信息来解释CT7001对这两个靶标活性差异的原因。
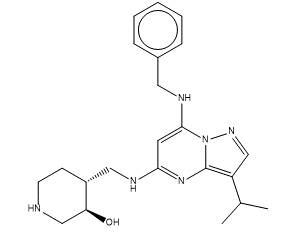
图1. CT7001的化学结构式
方法与结果
利用结合口袋的形状差异来设计选择性抑制剂是一个常用的策略[2],因此首先比较CDK7(PDB 7B5Q)与CDK2(PDB 5JQ5)的结合口袋以考察CT7001是否占据选择性的子口袋,结果如图2所示。
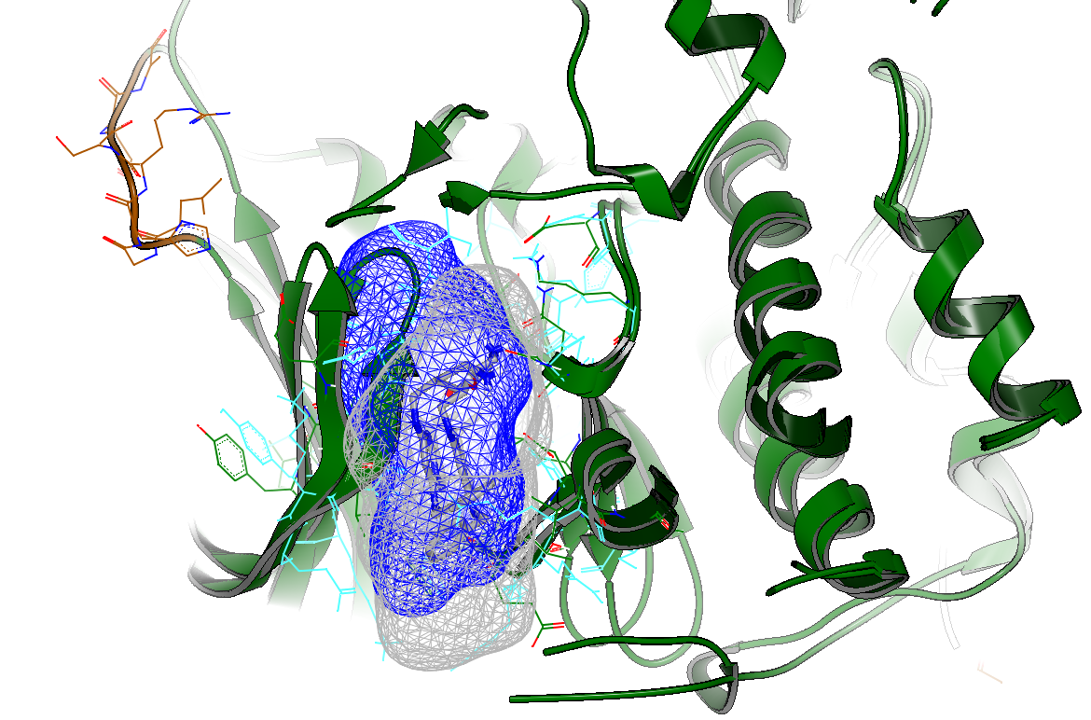
图2. CDK7(PDB 7B5Q)及CDK2(PDB 5JQ5)的结合口袋比较。蓝色:CDK7结合口袋;灰白色:CDK2结合口袋。
从图2可知,CDK7与CDK2的结合口袋确实存在较大差异,尤其图2左边有一个由loop连接的两个beta-片层结构:比起CDK2,在CDK7中该结构往左边偏移,这使得CDK7结合口袋在该方向上具有更宽敞的空间。这种差异以及其它的子口袋差异形成了CDK7与CDK2的选择性子口袋,如图3所示。

图3. CT7001与CDK7及CDK2的结合口袋三种不同视角比较。蓝色:CDK7结合口袋;白色:CDK2结合口袋。
然而CT7001似乎并没有占据CDK7或CDK2各自独特的差异性子口袋,而是结合于CDK7与CDK2的公共口袋处,如图3所示,这说明CT7001对CDK7与CDK2的活性差异另有原因。
 |
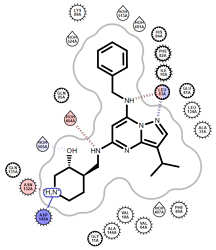 |
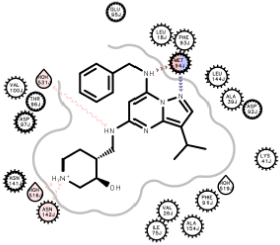
图4. CT7001与CDK7(左 PDB 7B5Q)及CDK2(右 PDB 5JQ5)的相互作用模式
用OEChem[3]分析PDB 7B5Q与5JQ5两个复合物结构,结果如图4所示。可以发现CT7001与CDK7、CDK2结合时母核的取向及其与绞链区的保守残基相互作用模式一致,而在侧链苯甲基以及羟基哌啶的取向与相互作用模式上有所不同。为了进一步定量分析,用分子对接软件Vina 1.2对复合物结构进行Vina的Docking打分,结果如表1所示。
表1. 用AutoDock Vina 1.2打分函数预测CT7001对CDK7与CDK2的结合亲和力
| Items | CDK7(PDB 7B5Q) | CDK2(PDB 5JQ5) |
|---|---|---|
| gauss 1 | 87.19586 | 89.24665 |
| gauss 2 | 1496.27128 | 1663.67783 |
| repulsion | 2.09848 | 3.04284 |
| hydrophobic | 45.44320 | 57.49107 |
| H-Bond | 2.32895 | 2.65225 |
| Score(kcal/mol) | -8.35325 | -8.87830 |
| IC50(nM) | 40 | 620 |
表1的Vina打分结果表明,CT7001与CDK7、CDK2相互作用时,具有相似的氢键、疏水、立体相互作用项,因而结合亲和力预测值相似,经典的分子Vina分子对接相互作用分析不能解释CT7001对这两个靶标的活性差异。
前面我们发现CT7001在CDK7与CDK2的结合位点里其苯甲基与哌啶环侧链的空间取向存在差异,这种差异也可能导致分子的张力能不同而产生生物活性差异。因此,有必要对CT7001的两种活性构象进行张力能分析。FreeForm[5]是专门用来评估构象张力能,考虑了溶剂效应、熵变与焓变。关于FreeForm的原理与更多算例请参见我之前的两篇博客文章[6,7],这里我用OpenFF2.0.0(Sage)力场来评估CT7001在CDK7与CDK2结合时的生物活性张力能,结果如图5、6所示。

图5. CT7001与CDK7(PDB 7B5Q)结合时的活性构象张力能
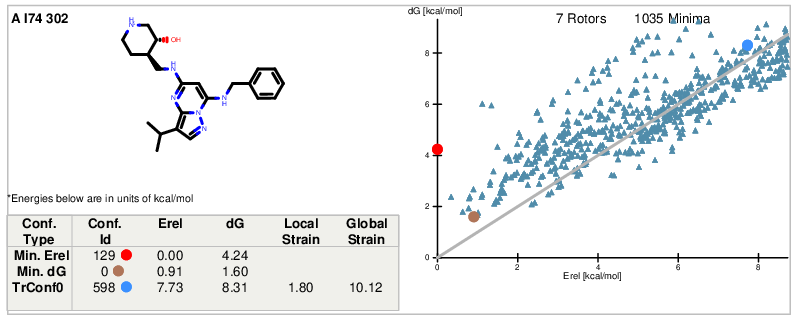
图6. CT7001与CDK2(PDB 5JQ5)结合时的活性构象张力能
CT7001与CDK7、CDK2结合时活性构象的张力能计算结果汇总为表2,CT7001的CDK7结合构象张力能(8.48 kcal/mol)要比CDK2结合构象张力能(10.12 kcal/mol)低约1.64kcal/mol,这与CT7001在两个靶标上的结合自由能ΔGbinding的差值1.62kcal/mol是几乎完全一致的。
表2. 用Openff-2.0.0 Sage力场计算CT7001的CDK7/CDK2结合活性构象张力能(单位:kcal/mol)
| Items | CDK7(PDB 7B5Q) | CDK2(PDB 5JQ5) |
|---|---|---|
| Relaxed Bioactive ΔG | 6.02 | 8.31 |
| Local ΔH | 2.45 | 1.80 |
| Global ΔE | 8.48 | 10.12 |
| ΔGbindinga | -10.08 | -8.46 |
a:根据IC50转化而来
CT7001的CDK7结合构象比CDK2结合构象低能的主要原因有两点:一方面,CDK7结合构象比CDK2结合构象多出了一个由哌啶环羟基与母核氮原子之间的分子内氢键(图7左黄色高亮),该分子内氢键稳定了CDK7结合构象;另一方面,在母核与侧链哌啶环之间的连接臂原子(图7左原子2)与哌啶环上连接原子(图7左原子3)之间的两面角1-2-3-4接近180度,这使得C2-C3两面角处于能量有利的邻位交叉式(见图7右),而在CDK2结合构象中C2-C3两面角(1-2-3-4)接近0度处于能量有不利的对位重叠式(见图8)。这些结构生物学信息为后续设计选择性更好的CDK7抑制剂提供了切入点。
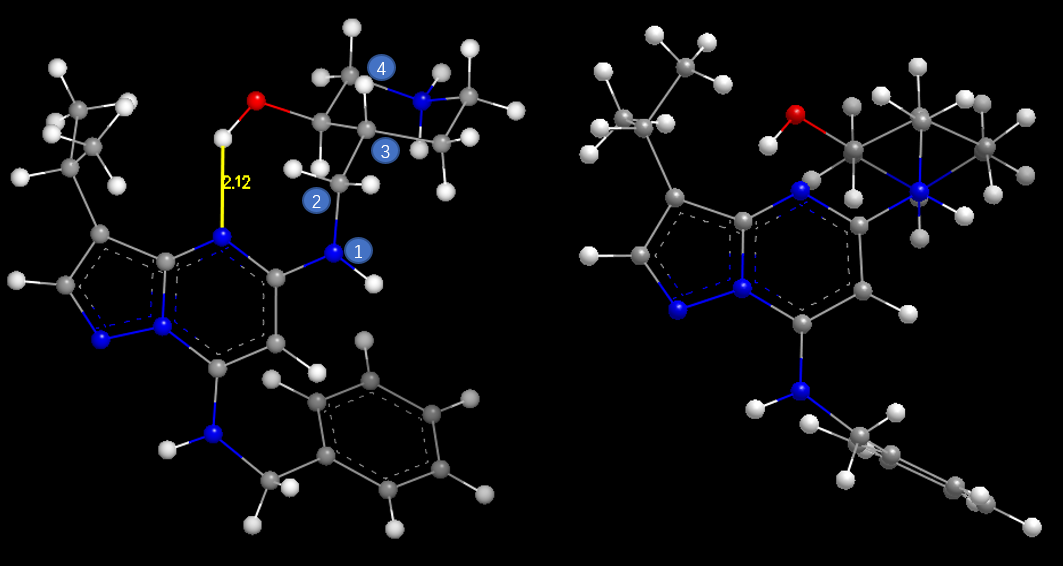
图7. CT7001与CDK7结合时的构象
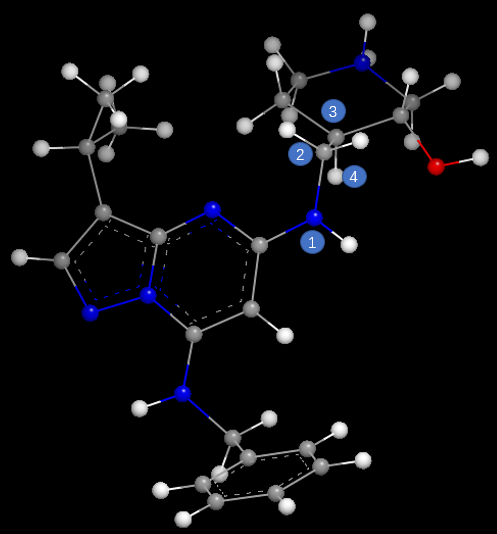
图8. CT7001与CDK2结合时的构象
总的来说,通过激酶特异性子口袋分析与配体-靶标相互作用分析没有发现CT7001对CDK7与CDK2的选择性原因,但是发现CT7001在CDK7、CDK2生物活性构象张力能上不仅存在显著差异,而且与活性差异相当,这提示可能是结合构象的张力能差异带来了选择性差异。当然,还有其它因素可能对活性差异有贡献,比如结合位点里的水[8]等,这里并没有对此类作用进行分析。
文献
- CDK7抑制剂用于癌症治疗:尝到成功的味道了吗? http://blog.molcalx.com.cn/2020/12/10/cdk7-inhibitors-in-cancer-therapy-the-sweet-smell-of-success.html
- Volkamer, A.; Eid, S.; Turk, S.; Rippmann, F.; Fulle, S. Identification and Visualization of Kinase-Specific Subpockets. J. Chem. Inf. Model. 2016, 56 (2), 335–346. https://doi.org/10.1021/acs.jcim.5b00627.
- OEChem. http://www.eyesopen.com/oechem
- Eberhardt, J.; Santos-Martins, D.; Tillack, A. F.; Forli, S. AutoDock Vina 1.2.0: New Docking Methods, Expanded Force Field, and Python Bindings. J. Chem. Inf. Model. 2021, 61 (8), 3891–3898. https://doi.org/10.1021/acs.jcim.1c00203.
- SZYBKI/FreeForm. https://www.eyesopen.com/szybki
- 肖高铿. FreeForm–构象稳定性评估及其在先导化合物优化中的应用. http://blog.molcalx.com.cn/2016/07/02/freeform-conformer-stability.html
- 肖高铿. 用张力能评估构象稳定性及其在先导化合物优化中的应用——更多力场选择. http://blog.molcalx.com.cn/2022/02/08/strain-energy-evaluation.html
- Robinson, D. D.; Sherman, W.; Farid, R. Understanding Kinase Selectivity Through Energetic Analysis of Binding Site Waters. ChemMedChem 2010, 5 (4), 618–627. https://doi.org/10.1002/cmdc.200900501.



