摘要:本文以一对结构相差一个溴原子而活性相差500倍的PTP1B抑制剂为例演示了Flare FEP在北京超级云计算中心GTX 3090 GPU计算资源上进行FEP计算的速度表现。靶标PTP1B全长为298个氨基酸残基,FEP计算共20个λ窗口,每个λ窗口进行了1ns时长的模拟。总共消费了3.13 GPU卡时,平均每个λ大约9.4卡分钟。整个计算在39分钟内完成,结合亲和力预测值与实验值刚好完全一致。这意味着,将Flare FEP与北京超级云计算中心的GTX 3090 GPU资源相结合,使得药化科研人员完全可以在一杯咖啡的时间内对结构相似的同系物完成一个相当精确的结合亲和力评估。
作者:肖高铿a,苏渤杰b
日期:2022-11-25
a. 广州市墨灵格信息科技有限公司
b. 北京超级云计算中心
前言
Flare FEP是一个高度集成且用户界面友好的自由能微扰(Free Energy Perturbation,FEP)计算工具,它由Cresset和英国爱丁堡大学的Julien Michel博士合作开发而来1。Flare FEP健壮、用户友好、经过充分验证2,使用“炼金术”转化来预测同系物的相对结合亲和力变化。
FEP计算有两个步骤非常耗费计算资源:一个是小分子的力场参数化,另一个是炼金术法过程中每个λ窗口的模拟。炼金术法FEP计算过程需要大量的GPU资源,在每对分子转化时的每个λ窗口都是独立的计算,因此可以用多个GPU分别同时进行。具体可以用多少张GPU卡加速取决于有多少对分子(linker)以及每对分子用了多少个λ窗口。得益于云计算供应商的发展,目前GPU资源可以方便从供应商那里以按量付费或包月、年付费的方式获得。根据之前我们的测试,Flare FEP对典型的算例(蛋白大约300-400氨基酸长度,每个λ窗口进行4ns的模拟,每对分子双向转化含18个λ窗口)在AWS云上常见的GPU计算资源测试的性能表现如表1所示。
表1. Flare FEP在AWS常见GPU计算资源上的性能表现
| System | Time (mins) for 1 λ window | Time (hrs) per-2-way 9 λ calc |
|---|---|---|
| g4dn.xlarge(Tesla T4) | 37.4 | 11.2 |
| p3.2xlarge (Tesla V100-SXM2) | 23.4 | 7.0 |
| g5.xlarge (A10G OpenCL) | 19.7 | 5.92 |
北京超级云计算中心GPU资源的特点在于可提供众多的RTX 3090 GPU计算资源,本文的主要目的是测试Flare FEP在关于北京超级云计算中心的RTX 3090 GPU上的性能表现,为大家在选择计算资源上提供可靠的参考。
测试材料与方法
测试方法
Wilson等人3报道的化合物32与33是一对活性相差500倍的PTP1B抑制剂,它们的结构与活性如图1所示。化合物32与靶标PTP1B的共晶结构已经解释出来,PDB代码为2QBP。化合物33是32的脱溴同系物,鉴于高度的结构相似性,可以认为两者与靶标具有同样的结合模式。
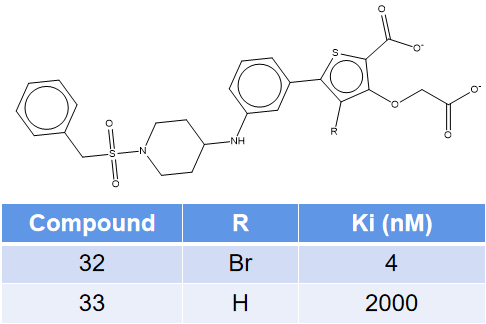
图1. 化合物32与33的结构式及其活性
在Flare V6.1里下载PDB 2QBP,然后用Protein Prep进行结构准备。准备过程按默认的方式进行,并将化合物32(即配体A 527)从结合位点里提取出来,化合物33是在提取出来32的基础上直接删除溴原子而来,这样就准备好了蛋白与化合物32以及33。
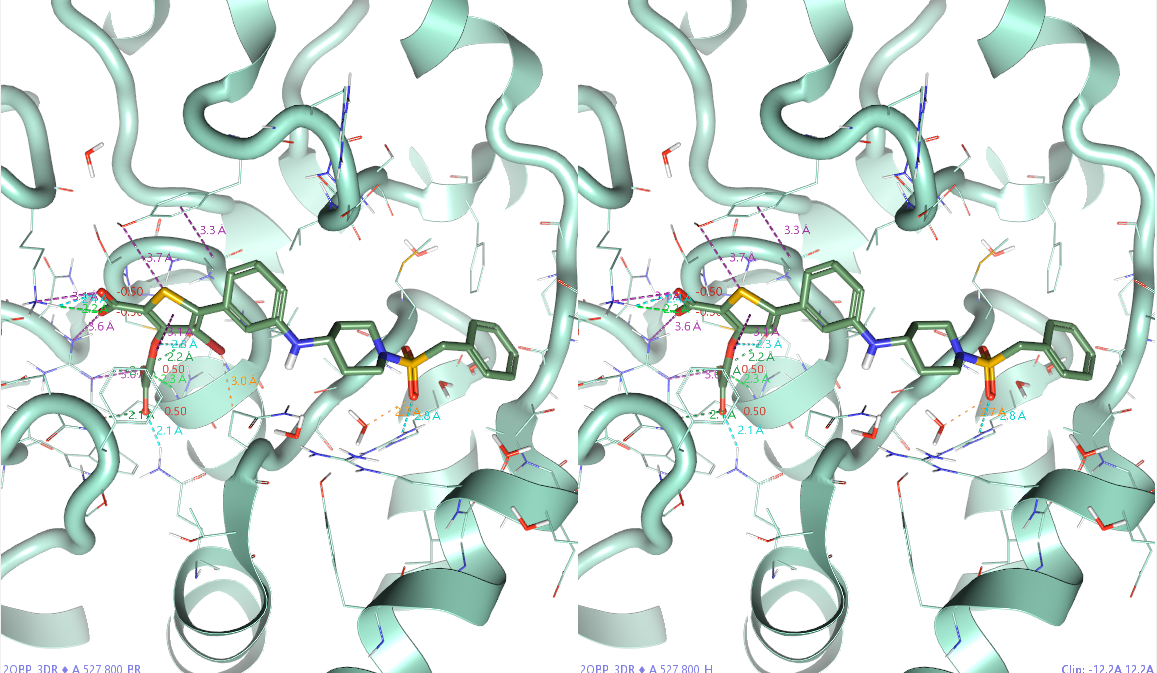
图2. 化合物32(左)与33(右)与PTP1B(PDB 2QBP,飘带状)的结合模式
比较化合物32、33的PTP1B的相互作用模式(图2),不能发现苯环上的溴取代带了什么新的相互作用。因此,常规的相互作用分析不能解释两者间500倍的活性差异。检查蛋白的A链,包含298个氨基酸残基。用Check protein对蛋白结构进行检查,发现没有什么错误之后,用3D-RISM预加水之后,开始进行FEP计算。
在Ligand表单选中32与33,然后点击Flare | 3D pose| FEP | New FEP Project创建FEP计算项目,参数如下:
- Ligands: Selected 2 Ligands
- Activity column: Ki
- Protein: 2QBP_P
- Chains: A Chain, A Water
点击Create按钮,弹出FEP Generate Graph对话框,设置如下参数:
- Select the type of graph: Generate new normal graph
- Mode: Benchmark
其它参数均采用默认值,然后点击Generate Graph弹出FEP计算界面。在确认Atom mapping无误之后,点击Run FEP,弹出FEP Calculation菜单(图3)。
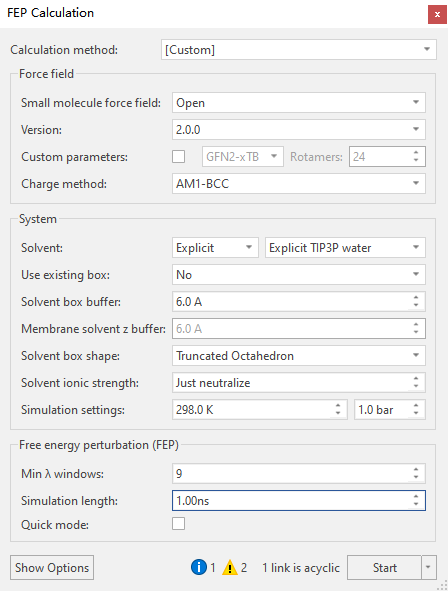
图3. FEP计算的参数设定
除了模拟时长(Simulation length)采用1ns之外,其它FEP计算参数采用默认值,其中最小的lambda窗口数为9,如图3所示。
计算环境
本次计算采用的集群为宁夏超算云N30分区。登录节点与计算节点的关键硬件参数如下表2所示。
表2. FEP计算的硬件环境
| 用途 | CPU与内存配置 | GPU配置 | 显存 |
|---|---|---|---|
| 头节点 | 48CPU物理核心/480GB内存 | 无 | 无 |
| 计算节点 | 48CPU物理核心/480GB内存 | 8张Nvidia 3090 | 24GB/卡 |
操作系统为Centos 7.9,显卡驱动为515.65.01,CUDA 11.7,队列管理系统为SLURM。
结果
计算完毕,得到图4所示的计算结果。箭头上下的数值为对应FEP计算转化的结合亲和力(ΔG)的差值(ΔΔG)。方框里面的A为ΔG实验值,P为ΔG预测值。其中预测值P是根据徐华锋4提出的数学方法计算而来。可以发现,计算值与实验值完全一致,这与我们在Hit Expander5用短时长(1ns)模拟的FEP进行排序与分类方法的正确性,也就是分子结构相差微小的分子间用1ns的模拟时长FEP计算进行初步排序与分类可提供足够准确的精度。
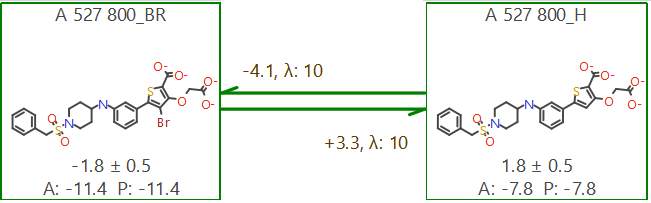
图4. 化合物32(左)与32(右)的FEP结合亲和力预测结果
本次计算蛋白长度为298个氨基酸长度、FEP计算每个方向10个λ窗口,双向转化共20个λ窗口,每个λ窗口模拟时长设为1ns,设置最大使用GPU计算卡数为8张。计算完毕,检查日志可以发现:从开始FEP计算到结束总共花了39分钟。注意这39分钟是完成作业所花费的时间,而不是每张卡39分钟。计算中,对FEP计算进度条的一个随机截图(图5)显示,FLARE FEP确实调用了8张来源于不同计算节点显卡,每张卡负责一个λ的计算。
图5. 在FEP计算中一个随机的进度条截图
机时系统显示,本次计算总共花费3.13GPU卡时,虽然在计算中也包含了拓扑网络的生成、力场的参数化以及500ps的分子动力学模拟平衡体系等计算,但是这些计算在本次实验中所用机时非常少,可以认为3.13小时全部都花在FEP计算本身上,则平均每个λ窗口的计算时间为9.4分钟,计算速度总结如表3所示。
表3. 在PTP1B抑制剂算例中,Flare FEP在RTX 3090 GPU设备上的计算速度
| System | Time (mins) for 1 λ window | Time (hrs) per-2-way 10 λ calc |
|---|---|---|
| RTX 3090 | 9.4 | 3.13 |
结论
本文以一对结构相差一个溴原子而活性相差500倍的PTP1B抑制剂为例演示了Flare FEP在北京超级云计算中心GTX 3090 GPU计算资源上的速度表现。其中靶标PTP1B全长为298个氨基酸残基,FEP计算的每个转化方向使用了10个λ窗口(双向共20个λ窗口),每个λ窗口进行了1ns时长的模拟。总共消费了3.13 GPU卡时,平均每个λ大约9.4卡分钟。整个计算在39分钟内完成,而且预测的结合亲和力与实验值刚好完全一致。这意味着,将Flare FEP与北京超级云计算中心的GTX 3090 GPU资源相结合,使得药化科研人员完全可以在1杯咖啡的时间内对结构相似的同系物完成一个相当精确的结合亲和力评估。
关于北京超级云计算中心
北京超级云计算中心具有强大的计算能力,丰富的软件资源,可信赖的支持团队,定制化的行业解决方案,可提供随需而用的超算资源,减少排队,适应多学科应用需求,可降低用户资源使用成本,并为大规模复杂技术和商业应用实现提供专业完整的解决方案。同时提供一对一专属微信群,7×24小时在线服务和5分钟快速响应机制。
文献
- Flare FEP. https://www.cresset-group.com/software/desktop/flare/flare-fep
- Kuhn, M.; Firth-Clark, S.; Tosco, P.; Mey, A. S. J. S.; Mackey, M.; Michel, J. Assessment of Binding Affinity via Alchemical Free-Energy Calculations. J. Chem. Inf. Model. 2020, 60 (6), 3120–3130. https://doi.org/10.1021/acs.jcim.0c00165.
- Wilson, D. P.; Wan, Z.-K.; Xu, W.-X.; Kirincich, S. J.; Follows, B. C.; Joseph-McCarthy, D.; Foreman, K.; Moretto, A.; Wu, J.; Zhu, M.; et al. Structure-Based Optimization of Protein Tyrosine Phosphatase 1B Inhibitors: From the Active Site to the Second Phosphotyrosine Binding Site. J. Med. Chem. 2007, 50 (19), 4681–4698. https://doi.org/10.1021/jm0702478.
- Xu, H. Optimal Measurement Network of Pairwise Differences. J. Chem. Inf. Model. 2019, 59 (11), 4720–4728. https://doi.org/10.1021/acs.jcim.9b00528.
- Hit Expander——一种快速评估并逐步完成苗头到先导流程的新方法,湿化学费用最少. 墨灵格的博客. http://blog.molcalx.com.cn/2022/10/27/hit-expander.html
联系我们,亲自测试FEP
下载算例项目文件,搭建测试环境,请联系我们。



