摘要:在本文中,我们提出了一种新的结合位点水热力学性质计算方法,也就是使用基于XED力场的3D-RISM来放置初始的水分子位置,然后用基于分子动力学模拟的GIST方法来计算结合位点里水的热力学学性质。具体的讲,本文以PDB 1FTM为算例,用Flare V6中内置的3D-RISM与GIST方法,测试了三种不同放置初始水的方法对GIST的影响,分别为1)用分子动力学模拟为干结合位点放置初始的水;2)用共晶结构的水链作为初始的水;与3)用基于XED力场的3D-RISM为干结合位点放置初始的水。结果表明,用基于XED力场的3D-RISM来放置初始水的GIST方法性能表现最佳。
肖高铿a,苑琪b/2022-07-17
a. 广州市墨灵格信息科技有限公司. 中国广东省广州市
b. Cresset BioMolecular Discovery Ltd. Cambridge, UK
1. 前言
水分子在蛋白质-配体结合中发挥着至关重要的作用,介导相互作用以及去溶剂化自由能都能显著提高配体的结合亲和力[1,2]。以Grid inhomogeneous solvation theory(GIST)为代表的基于分子动力学 (Molecular Dynamics,MD) 模拟的方法可用于评估水分子的自由能贡献[3-5]。对于溶剂暴露的结合位点来说,初始的水位置并不重要,因为在结合位点和溶剂之间高频的水分子位置交换保证了水合位点分析在纳秒时间尺度上可快速收敛。但是,对于封闭的结合位点而言,情况并非如此,因为蛋白折叠后水分子通常会被包埋在蛋白表面之下。此外,锁在配体占据的结合位点里的水分子也会出现类似的问题[6]。在这两种情况下,水合位点的分析在纳秒尺度上难以快速收敛,因此在进行分子动力学模拟前,预先将水分子放置到结合位点中对于精确确定水合位点的热力学性质至关重要。
目前,有几种放置水分子的方法可以用来为分子动力学模拟做准备。一种流行的方法是使用预先平衡的水盒。将蛋白质放入水盒后,去除那些与蛋白残基发生空间重叠的溶剂分子,这可能导致蛋白质周围和内部产生真空气泡,必须在生产运行(production run)前通过系统平衡来解决。平衡(Equilibration)可以平滑蛋白质周围水的密度,但不能填补封闭区域内产生的真空。这是由于溶剂往空腔扩散所需的时间过长,如果没有非常长的模拟,封闭空腔内采样可能会很差[7]。为了解决这个问题,人们开发了另一种放置水分子的方法——大正则蒙特卡罗( grand-canonical Monte Carlo,GCMC) 模拟[7]。通过在特定区域内迭代地插入和删除水分子,GCMC可以准确地对蛋白质周围和封闭腔内的水分子进行采样、预测水分子的位置。但由于需要大量迭代才能正确放置,因此GCMC的计算成本也相当高。为了更快速地放置水分子,人们还开发了统计力学方法,基于由3D-RISM[8,9]等算法生成的溶剂密度分布来放置显式水分子。3D-RISM利用Ornstein-Zernike方程对溶剂成分的分布进行计算,不需要长时间的分子动力学模拟,因此可以很快的产生可靠的溶剂分布和离子分布。Sindhikara等人[10]开发的Placevent就是一种统计力学方法,它使用频次函数(population function)将水分子一次一个地放置到占据概率最高的位置,直到达到足够的密度。此外,Fusani等人[11]提出了基于遗传算法的GAsol方法来将水分子放置到密度分布中。比之Placevent方法,GAsol预测水分子位置的精度更高。
在本文中,我们提出了一种新的结合位点水热力学性质计算方法,也就是使用基于XED力场的3D-RISM来放置初始的水分子位置,然后用基于分子动力学模拟的GIST方法来计算结合位点里水的热力学性质。具体地讲,本文以PDB 1FTM为算例,用Flare V6[12]中内置的3D-RISM与GIST方法,测试了三种不同放置初始水的方法对GIST的影响,分别为1)用分子动力学模拟为干结合位点放置初始的水;2)用共晶结构的水链作为初始的水;与3)用基于XED力场的3D-RISM为干结合位点放置初始的水。结果表明,用基于XED力场的3D-RISM来放置初始水的GIST方法性能表现最佳。
2. 方法
2.1 蛋白结构准备
将共晶结构(PDB 1FTM)从蛋白质数据库下载到Flare V6[12]中,并使用其中的Protein Prep工具小心地准备以添加氢原子、优化氢键、消除原子冲突并给蛋白结构分配最佳质子化状态。任何截短的蛋白质链被封端作为蛋白质准备的一部分。PDB 1FTM包含A、B、C三个链,将B与C链删除,仅保留A链,并将A链的配体AMQ从蛋白中提取出来。
2.2 3D-RISM分析
用Flare V6[12]中的3D-RISM模块针对准备好蛋白A链进行分析,使用了如下条件:
- XED force field and charge method
- 0.4Å grid spacing
- 14Å grid external border width
- Convergence tolerance: 10-8
- Maximum number of iterations: 10,000
- Total formal charge handling: neutralize with counterions.
计算完毕,蛋白的A链会多出一条名称为A 3DR的水链,注意3DR链中仅含氧原子。
2.3 用分子动力学模拟加水进行GIST分析(GIST_DRY)
用Flare V6[12]别对“干”结合位点Apo结构(不包含配体、金属离子与结晶水)进行GIST分析,使用了如下条件:
- Calculation method: Normal
- Ligand: None
- Grid spacing:0.5 Å
- Grid Definition:Ligand
- Chains: A Chain
- Simulation length: 20ns
- Solvent Model: explicit TIP4Pew Water
在这个计算中,因为在分子动力学模拟之前没有添加任何的水,所以计算之后结合位点里的水是依靠分子动力学模拟平衡进去。
2.4 用水链做为初始水的位置进行GIST分析(GIST_WET)
用Flare V6[12]对“湿”结合位点Apo结构(不包含配体与金属离子)进行GIST分析,共晶结构的A链水包含在GIST分析里面,具体的GIST分析条件如下:
- Calculation method: Normal
- Ligand: None
- Grid spacing:0.5 Å
- Grid Definition:Ligand
- Chains: A Chain, A Water
- Simulation length: 20ns
- Solvent Model: explicit TIP4Pew Water
在这个计算中,在分子动力学模拟之前将共晶结构的水链包含在GIST分析里,所以结合位点里初始的水仅来源于共晶结构里的水。
2.5 用3D-RISM预测初始水的位置进行GIST分析(GIST_3DR)
在经过2.2所述3D-RISM加了水链的蛋白结构中,先删除共晶水链;再用2.1的方法对蛋白结构进行准备,主要目的是给3DR水链加上了合适的氢;最后对结合位点Apo结构(不包含配体与金属离子)进行GIST分析,其中3DR水链包含在GIST分析里面。具体的GIST分析条件如下:
- Calculation method: Normal
- Ligand: None
- Grid spacing:0.5 Å
- Grid Definition:Ligand
- Chains: A Chain, A 3DR
- Simulation length: 20ns
- Solvent Model: explicit TIP4Pew Water
在这个计算中,结合位点里初始的水全部源于3D-RISM预测。
3. 结果
3.1 蛋白结构准备
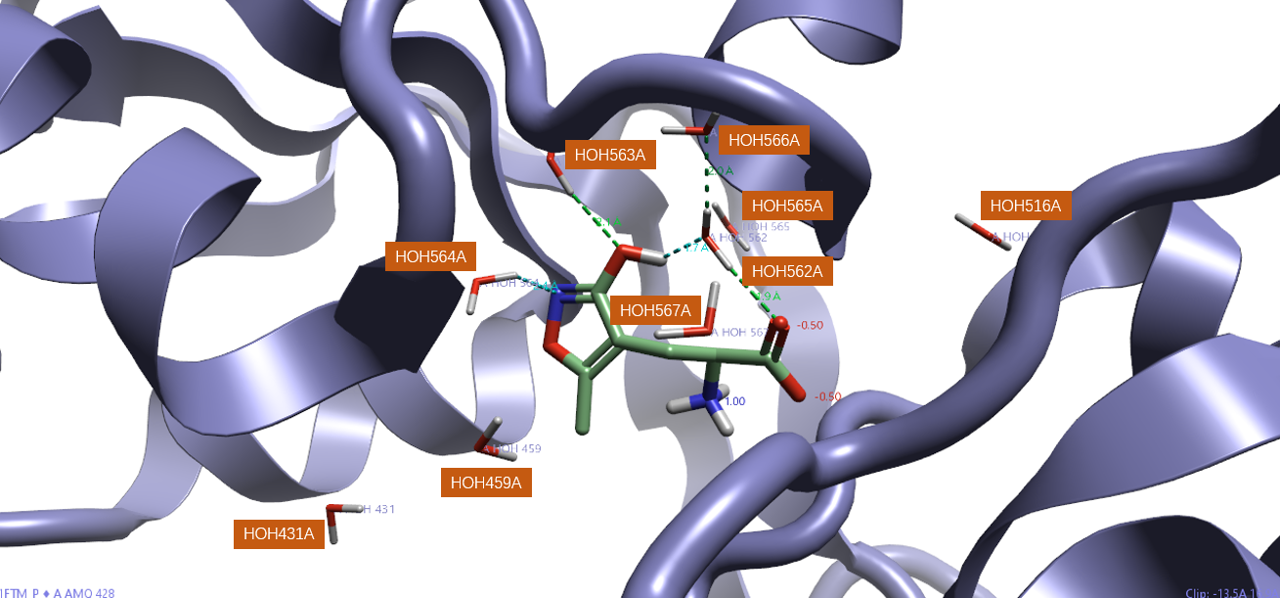
我们重点考察不同水放置方法对GIST分析的影响,因此首先观察一下结合位点内水的分布。图1显示了配体及周围的水分子,可以发现有9个水分子在配体6Å附近。
图1. PDB 1FTM的A链配体及其6Å内9个水分子
用Flare V6的fpocket对A链进行分析,发现配体周围的残基形成了一个独立、密闭的结合口袋(图2灰白色网格状表面);在上述的9个水分子中,HOH431A、HOH516A、HOH459A、HOH565A与HOH567A等5个水分子位于密闭口袋的外部,其余的4个水分子位于口袋的内部。这为考察不同计算方法的差异提供了非常好的素材。
图2. PDB 1FTM的A链配体所在结合口袋(灰白色网格表面)以及5个口袋外的水(HOH431A、HOH459A、HOH567A、HOH516A与HOH565A),其中HOH565A在背面没被展示出来。
我们重点考察的是不同水分子预测方法在PDB 1FTM体系上的性能表现差异,尤其三种不同初始水分子位置预处理方法对GIST分析方法的影响。
3.2 3D-RISM预测结果
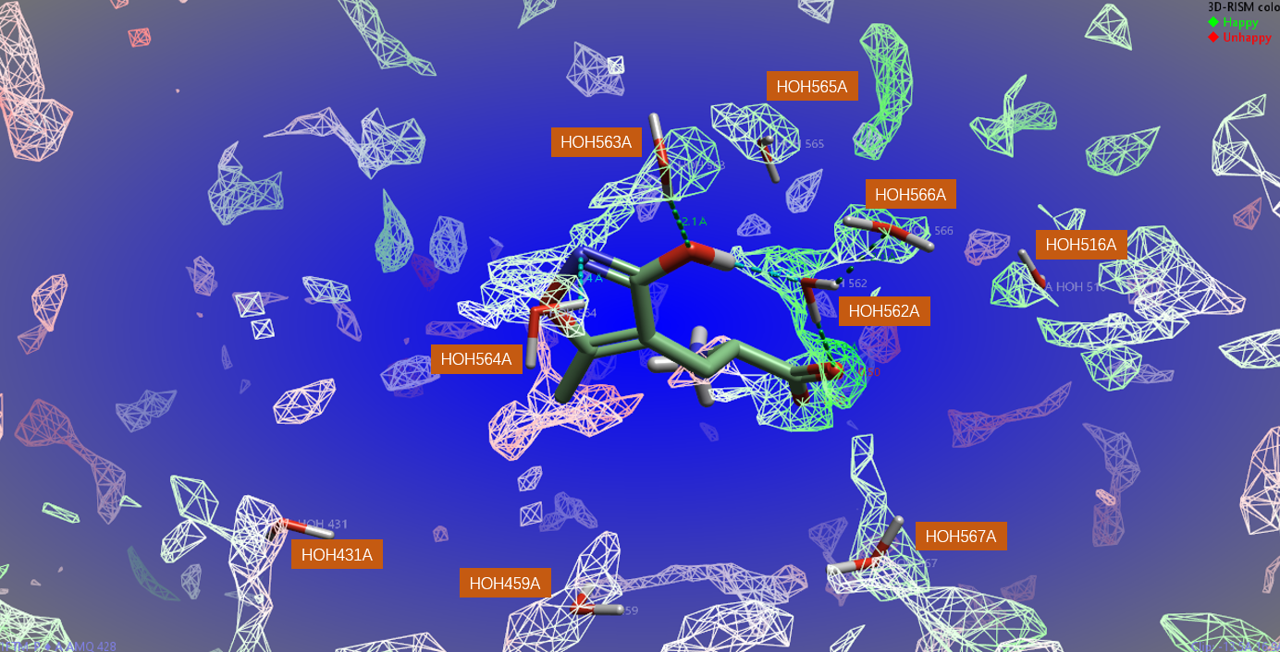
为了清晰起见,图3仅显示了配体、6Å内的9个水分子以及3D-RISM计算的氧(ρO) 密度等值图。氧(ρO) 密度等值图反映了3D-RISM计算发现氧原子(即水分子)高概率出现的区域,等值图根据ΔG进行着色。负值 ΔG(绿色)代表“Happy”(favorable)水分子:说明3D-RISM预测这些水分子呆在蛋白里比呆在水中更加稳定;正值ΔG(红色)等值图表示”Unhappy”(unfavarable)水分子,这些水分呆在蛋白里不如呆在水中稳定,因此容易被配体替换。图3显示的等值图的密度阈值ρO=5.0。
图3. 在结合位点里配体、水以及与基于XED力场3D-RISM预测的水密度(ρO=5)等值图
如图3所示,配体及其周围9个水分子均被ρO=5的水密度等值图覆盖,这说明3D-RISM预测的结果相当不错。除了给出水密度(ρO=5)等值图之外,还根据密度等值图生成了一个水链3DR,可视化结果如图4所示。
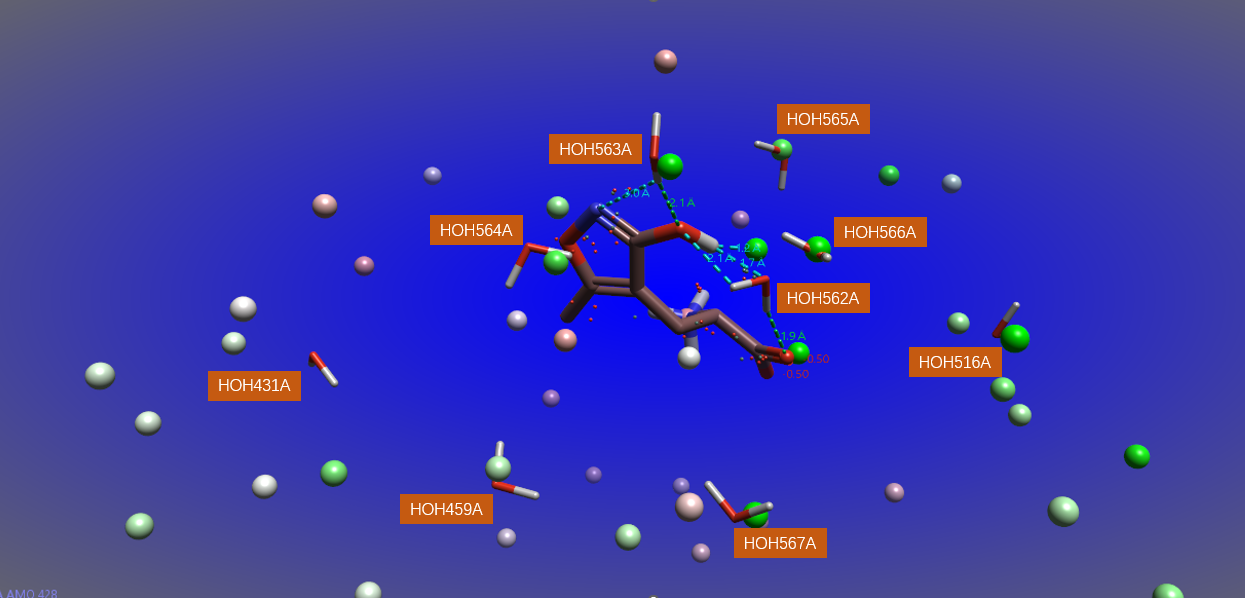
图4. 3D-RISM预测水链与9个共晶水的比较。圆球:3D-RISM预测的水分子位置;棒状水分子:A链共晶水分子。
如图4所示,可以发现除了水分子HOH431A之外,其余的8个水分子都有一个预测的水分子与之重合(预测的水与共晶水氧原子距离不大于1.4Å)。这进一步说明了3D-RISM预测的水分子位置与实验结果相当吻合,3D-RISM适用于密闭结合位点的水分子预测。
3.3 用分子动力学模拟加水进行GIST分析(GIST_DRY)
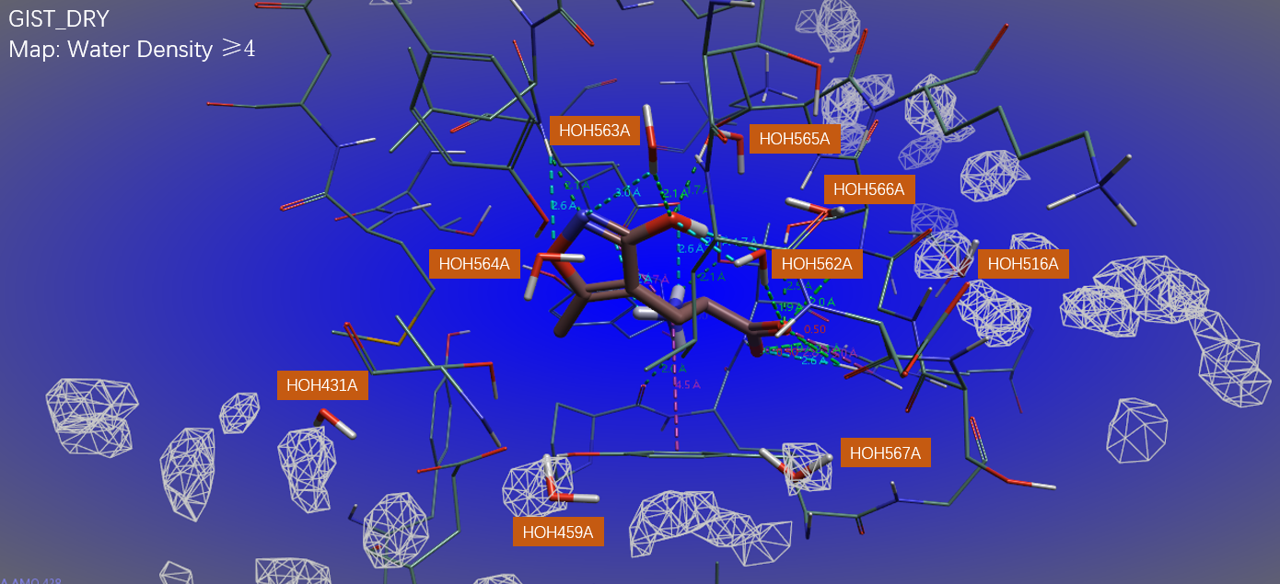
对“干”结合位点Apo结构(不包含配体、金属离子与水链)进行GIST分析的结果如图5所示,可以发现结合位点里没有水密度分布。9个水分子中,仅在结合口袋外部的4个水HOH431、HOH459、HOH567与HOH516被水密度为4的等值图覆盖,口袋内部的全部4个水与口袋外部的一个水HOH566等没有水密度等值图覆盖。这个结果与文献报道的一致,即使用分子动力学模拟预先平衡水盒加初始化的水不适用于密闭结合位点的GIST分析。
图5. 分子动力学模拟加水进行GIST分析(GIST_DRY)得到的水密度图
3.4 用水链做为初始水的位置进行GIST分析(GIST_WET)
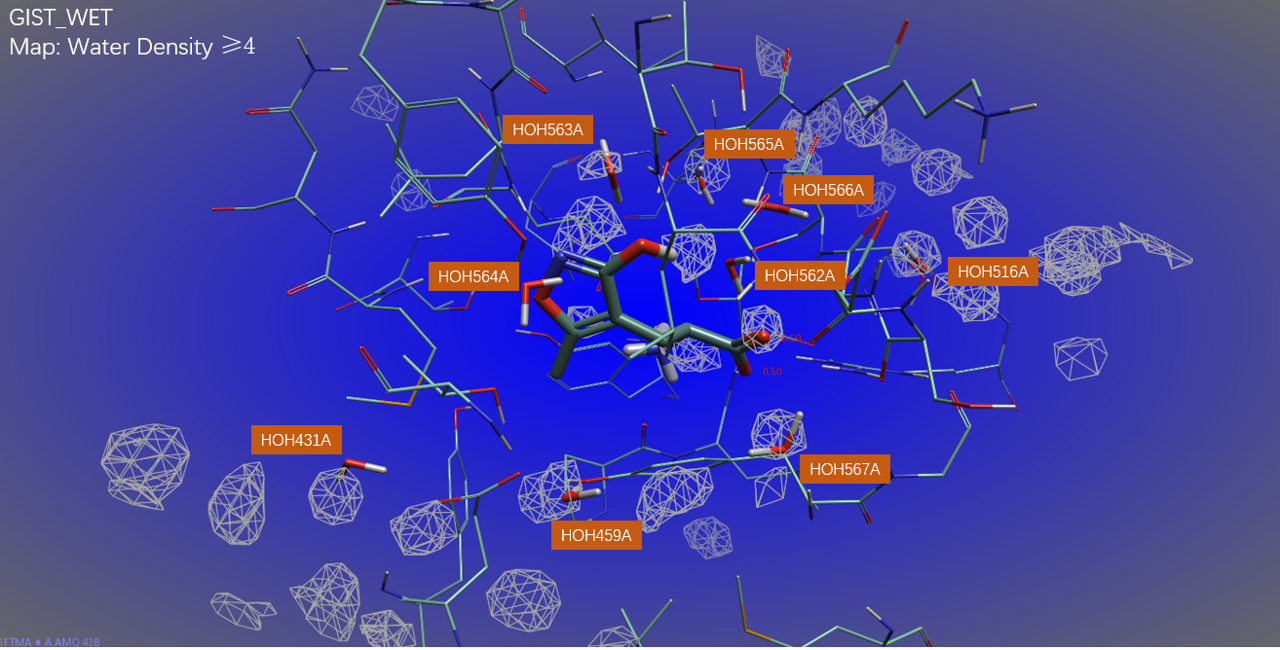
为了探索共晶水分子是否足以得到满意的GIST分析结果,对“湿”结合位点Apo结构(不包含配体、金属离子,但包含水链)进行了GIST分析,结果如图6所示。可以发现,在9个水分子中,三个在结合口袋内部的水(HOH564、HOH566与HOH562)与一个口袋外部的水(HOH566)没有被水密度等值图覆盖,其它的5个口袋外部水均被水密度为4的等值图覆盖。比之仅依赖分子动力学模拟水平衡加水的方法,利用共晶水作为密闭结合位点初始水位置的GIST分析方法提高了预测精度。
图6. 用水链做为初始水的位置进行GIST分析(GIST_WET)得到的水密度图
3.5 用3D-RISM预测初始水的位置进行GIST分析(GIST_3DR)
在这个计算中,以3D-RISM生成的水链作为初始化的水进行GIST分析。如图4所示,3D-RISM预测是水充满了结合口袋,不仅覆盖了湿口袋的共晶水,还在配体所在的位置放置了水。除此之外,结合口袋外部的整个蛋白都放置了3D-RISM预测的水。以3D-RISM预测水作为初始化水进行的GIST分析结果如图7所示,水密度为4的等值图覆盖结合位点附近全部的9个水分子。
图7. 用3D-RISM预测初始水的位置进行GIST分析(GIST_3DR)得到的水密度图
图8显示了GIST_3DR计算的ΔG等值图,红色等值图表示ΔG大于0.5kcal/mol, 绿色等值图表示自由能ΔG小于-0.5kcal/mol。观察到9个水分子都有等值图覆盖,且都为ΔG大于0.5,这说明这些水在apo结合口袋里是unhappy的。
图8. GIST_3DR分析得到的水ΔG等值图。红色等值图:ΔG大于0.5kcal/mol, 绿色等值图:ΔG小于-0.5kcal/mol。
Local happy water定义为ΔG小于-0.5kcal/mol且Water density大于4的水,local unhappy water定义为ΔG大于0.5kcal/mol且Water density大于4的水。对高密度、高能的local unhappy water进行替换或置换有可能提高化合物的亲和力,图9显示了水密度大于4的happy water(绿色)与unhappy water(红色)。
图9. GIST_3DR分析得到的红色local unhappy water与绿色local happy water等值图。红色:ΔG大于0.5kcal/mol,且water density大于4;绿色:ΔG小于-0.5kcal/mol且water density大于4。
GIST计算的热力学性质除了自由能之外(ΔG)之外,还有焓变(ΔH)与熵变(-TΔS)。图10显示了焓变等值图,其中红色等值图代表焓变大于2.0kcal/mol的区域,绿色等值图代表焓变小于-2.0kcal/mol的区域。综合利用水合位点上的热力学性质,可以帮我们进一步理解小分子的SAR,制定小分子结构优化的策略。
图10. GIST_3DR分析得到的ΔH等值图。红色:ΔH大于2.0 kcal/mol, 绿色等值图:ΔH小于-2.0kcal/mol。
图11显示了熵变等值图,其中红色等值图代表熵变(-TΔS)大于2.0kcal/mol的entropic unhappy区域,这意味着熵变对水自由能的贡献大于阈值2kcal/mol。
图11. GIST_3DR分析得到的熵变等值图。红色为-TΔS大于2.0 kcal/mol。
3.6 不同方法的比较
表1总结了上述四种不同水分子位置与稳定性预测方法在PDB 1FTM结合位点处的性能,其中位置是相对Flare Pocket计算的结合口袋而言(见3.1小节),内表示水分子位于结合口袋内部,外表示水分子位于结构口袋外部;其中3D-RISM(对干结合位点进行计算,参见3.2小节)、GIST_DRY(对干结合位点进行计算,参见3.3小节)、GIST_WET对湿结合位点进行计算,参见3.4小节与GIST_3DR(3D-RISM预放置水,参见3.5小节)列表示不同计算方法的水密度是否覆盖了实验水,其中3D-RISM的水密度阈值为5,GIST的水密度阈值为4,绿色表示该结晶水被水密度等值图覆盖,红色表示结晶水没有水密度等值图覆盖。
表1. 不同方法计算水密度图覆盖共晶水位置的比较
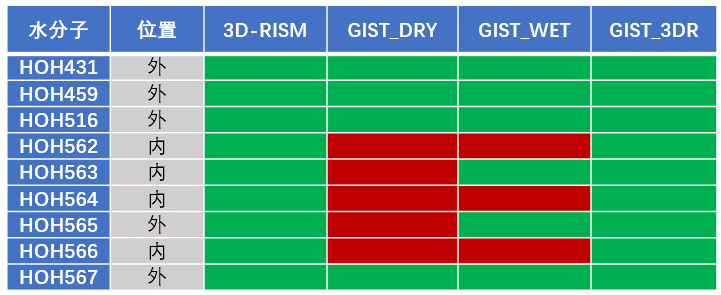
如表1所示,对于1FTM这个密闭的结合位点,采用分子动力学模拟平衡水作为对干结合位点加水的GIST分析方法(GIST_DRY)没有水密度出现在结合位点里,更别提水密度覆盖口袋的4个共晶水,性能表现最差;用共晶结构水链做为初始水的位置进行GIST分析(GIST_WET)的方法比之GIST_DRY方法有所改善,但结合位点里已依旧有3个共晶水没有密度覆盖;基于XED力场的3D-RISM与基于3D-RISM计算的水为初始水位置的GIST分析(GIST_3DR)的水密度不仅覆盖了结合位点外部的共晶水,而且结合位点里面的共晶水也有水密度覆盖,两者的性能表现最佳,因此适合于密闭结合位点水的位置与热力学性质计算。GIST_3DR比GIST_DRY与GIST_WET对密闭口袋里的有更好的水密度覆盖,这说明用XED力场电荷的3D-RISM为分子动力学模拟加水提高了GIST分析的精度,使得GIST也适用于对密闭结合口袋进行水的热力性质计算。
4. 结论
在本文中,我们提出了一种新的结合位点水热力学性质计算方法,也就是使用基于XED力场的3D-RISM来放置初始的水分子位置,然后用基于分子动力学模拟的GIST方法来计算结合位点里水的热力学性质。
本文以PDB 1FTM为例,用Flare V6[12]中内置的3D-RISM与GIST方法,测试了3D-RISM以及三种不同放置初始水的GIST方法的性能,这三种GIST方法分别为1)用分子动力学模拟为干结合位点放置初始的水;2)用共晶结构的水链作为初始的水;与3)用基于XED力场的3D-RISM为干结合位点放置初始的水。结果表明,用基于XED力场的3D-RISM与基于3D-RISM来放置初始水的GIST方法对密闭结合位点的水分子预测性能表现最佳,均可适用于密闭结合位点水的分析。
5. 文献
- Ladbury, J. E. Just Add Water! The Effect of Water on the Specificity of Protein-Ligand Binding Sites and Its Potential Application to Drug Design. Chem. Biol. 1996, 3 (12), 973–980. https://doi.org/https://doi.org/10.1016/S1074-5521(96)90164-7.
- Abel, R.; Young, T.; Farid, R.; Berne, B. J.; Friesner, R. A. Role of the Active-Site Solvent in the Thermodynamics of Factor Xa Ligand Binding. J. Am. Chem. Soc. 2008, 130 (9), 2817–2831. https://doi.org/10.1021/ja0771033.
- Lazaridis, T. Inhomogeneous Fluid Approach to Solvation Thermodynamics. 1. Theory. J. Phys. Chem. B 1998, 102 (18), 3531–3541. https://doi.org/10.1021/jp9723574.
- Nguyen, C.; Gilson, M. K.; Young, T. Structure and Thermodynamics of Molecular Hydration via Grid Inhomogeneous Solvation Theory. ArXiv 11084876 Phys Q-Bio. 2011. https://doi.org/10.48550/arXiv.1108.4876
- Nguyen, C. N.; Cruz, A.; Gilson, M. K.; Kurtzman, T. Thermodynamics of Water in an Enzyme Active Site: Grid-Based Hydration Analysis of Coagulation Factor Xa. J. Chem. Theory Comput. 2014, 10 (7), 2769–2780. https://doi.org/10.1021/ct401110x.
- Carugo, O. Statistical Survey of the Buried Waters in the Protein Data Bank. Amino Acids 2016, 48 (1), 193–202. https://doi.org/10.1007/s00726-015-2064-4.
- Ross, G. A.; Bodnarchuk, M. S.; Essex, J. W. Water Sites, Networks, And Free Energies with Grand Canonical Monte Carlo. J. Am. Chem. Soc. 2015, 137 (47), 14930–14943. https://doi.org/10.1021/jacs.5b07940.
- Kovalenko, A.; Hirata, F. Three-Dimensional Density Profiles of Water in Contact with a Solute of Arbitrary Shape: A RISM Approach. Chem. Phys. Lett. 1998, 290 (1–3), 237–244. https://doi.org/10.1016/S0009-2614(98)00471-0.
- Sindhikara, D. J.; Hirata, F. Analysis of Biomolecular Solvation Sites by 3D-RISM Theory. J. Phys. Chem. B 2013, 117 (22), 6718–6723. https://doi.org/10.1021/jp4046116.
- Sindhikara, D. J.; Yoshida, N.; Hirata, F. Placevent: An Algorithm for Prediction of Explicit Solvent Atom Distribution-Application to HIV-1 Protease and F-ATP Synthase. J. Comput. Chem. 2012, 33 (18), 1536–1543. https://doi.org/10.1002/jcc.22984.
- Fusani, L.; Wall, I.; Palmer, D.; Cortes, A. Optimal Water Networks in Protein Cavities with GAsol and 3D-RISM. Bioinformatics 2018, 34 (11), 1947–1948. https://doi.org/10.1093/bioinformatics/bty024.
- Flare 6.0.0. Cresset BioMolecular Discovery Ltd. https://www.cresset-group.com/flare