摘要:本文演示了如何用Flare QM的扭转角分析以发现高能的生物学构象并由此发现化合物优化的机会。具体地讲,以BMS Factor XIa抑制剂优化为例,一方面重现了丙烯酰胺高能活性构象的发现,解释了环化对活性33倍的优化效果;另一方面,发现了咪唑2位取代的高能活性构象,解释了大环化对活性78倍的优化效果。此外,还通过QM计算提出咪唑环可能的质子化状态假设,为理解化合物的SAR提供了新的视角。总的来说,基于QM的扭转角分析提供了一个高度可靠的工具以发现巨大的先导化合物优化机会。
肖高铿/2023-11-07
前言
NRG Therapeutics的Gills Ouvry博士分享了一张标题为Why torsions matter的图文1,我将他的观点总结为:扭转角分析(Torsion analysis)可以发现共晶结构中高能的生物学构象,这意味着巨大的先导化合物设计机会。他以BMS Factor XIa项目团队的研究为例2,如图1所示,分析肉桂酰胺系列化合物的生物学构象可以发现,在结合模式时的丙烯酰胺呈s-trans构象,这是一个高能构象,用环状的氨基甲酸内酯替换之后,活性提高了33倍。

图1. 扭转角分析发现设计机会1
在BMS的Factor XIa项目中2,如图2所示,除了涉及上述从化合物9到10的丙烯酰胺环化的设计之外,还涉及8到10的大环化设计。

图2. 大环化合物10的发现2
本文的目的是之一是演示如何用Flare3的静电分析与量子化学计算来重现Gilles Ouvery博士通过对丙烯酰胺的扭转角分析来发现优化机会,另一个目的是用QM方法来发现该分子相关的质子化状态、重现发现从8到10的大环化优化机会。
丙烯酰胺片段的环化
用Flare下载共晶结构PDB 4Y8Y,裁剪出配体的丙烯酰胺片段,加上全部氢原子,用Flare QM模块4在B3LYP-D3BJ/6-311G(d,p)理论水平对片段进行结构优化与Torsion Profile分析,结果如图3所示。
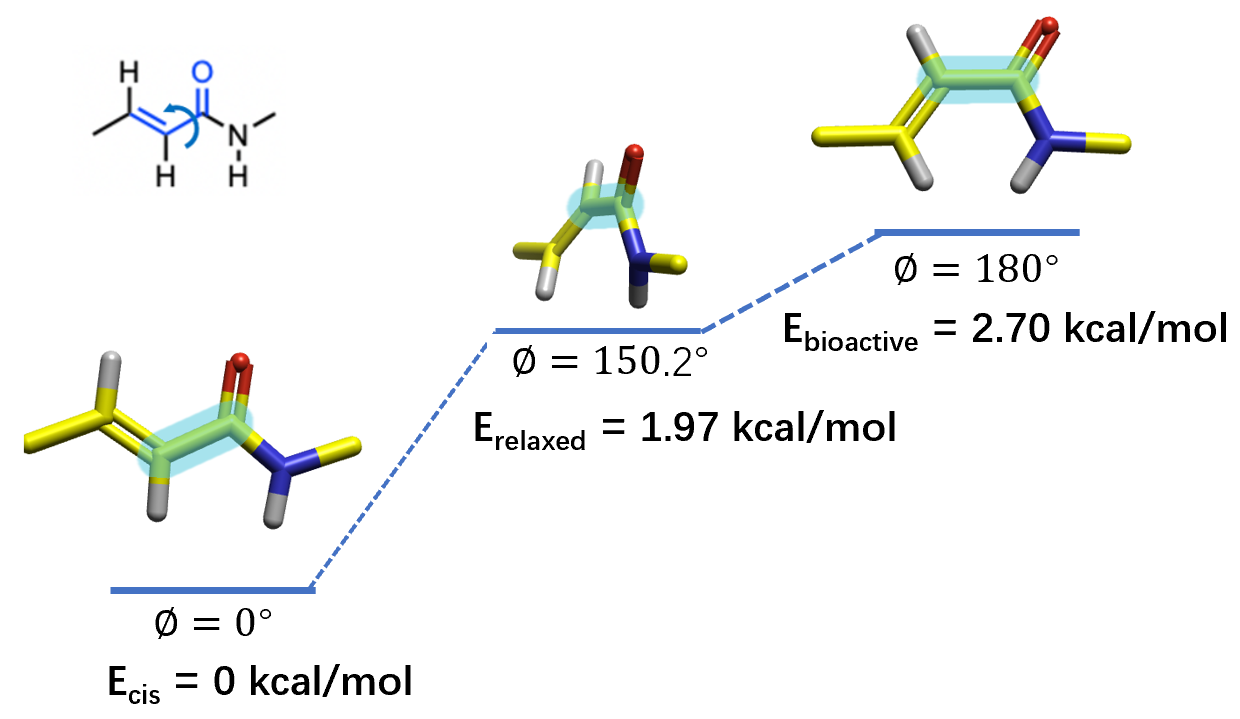
图3. 丙烯酰胺片段的构象能分析
基于QM的扭转角分析表明,s-cis式丙烯酰胺片段构象是全局最低能构象,s-trans式的生物活性构象比s-cis能量高了约2.7kcal/mol,因此设计将丙烯肉桂酸固定在s-cis活性构象的设计将有利于活性的提高。如图3所示,从9到10的优化获得33倍的活性提高证明了这一点。
咪唑环的质子化状态
如图4所示,BMS Factor XIa系列化合物2与苯环连接的咪唑具有两个潜在的不同质子化状态异构体。无论是在Gilles Ouvery1的长图中还是Clark等人2的文章中,均将质子化状态定为图4左的模式,即质子在苯基的Epsilon位(以下简称为异构体E)。但是我认为图4右,即质子在苯基Delta位(以下简称异构D)的质子化状态才是优势的异构体。

图4. PDB 4Y8Y共晶配体咪唑环的两个可能质子化状态。左:质子在苯基的Epsilon位(红色箭头);右:质子在苯基的Delta位(红色箭头)。
首先在Flare里生成PDB 4Y8Y共晶配体的两种异构体,然后计算两个异构体的分子表面,用XED力场计算MEP并对分子表面进行渲染着色,结果如图5所示。从图5可以发现,两种质子化状态在红色圆圈高亮处具有显著不同的静电模式,其中Epsilon质子化状态(图5左)存在显著地分子内静电排斥;而Delta质子化状态则呈现为分子内互补。鉴于该处涉及可旋转键链接的基团,因此这种分子内静电冲突可能导致生物活性构象稳定性变差,为此有必要进一步用QM对相关两面角进行分析以确认哪一种质子化状态更加稳定。
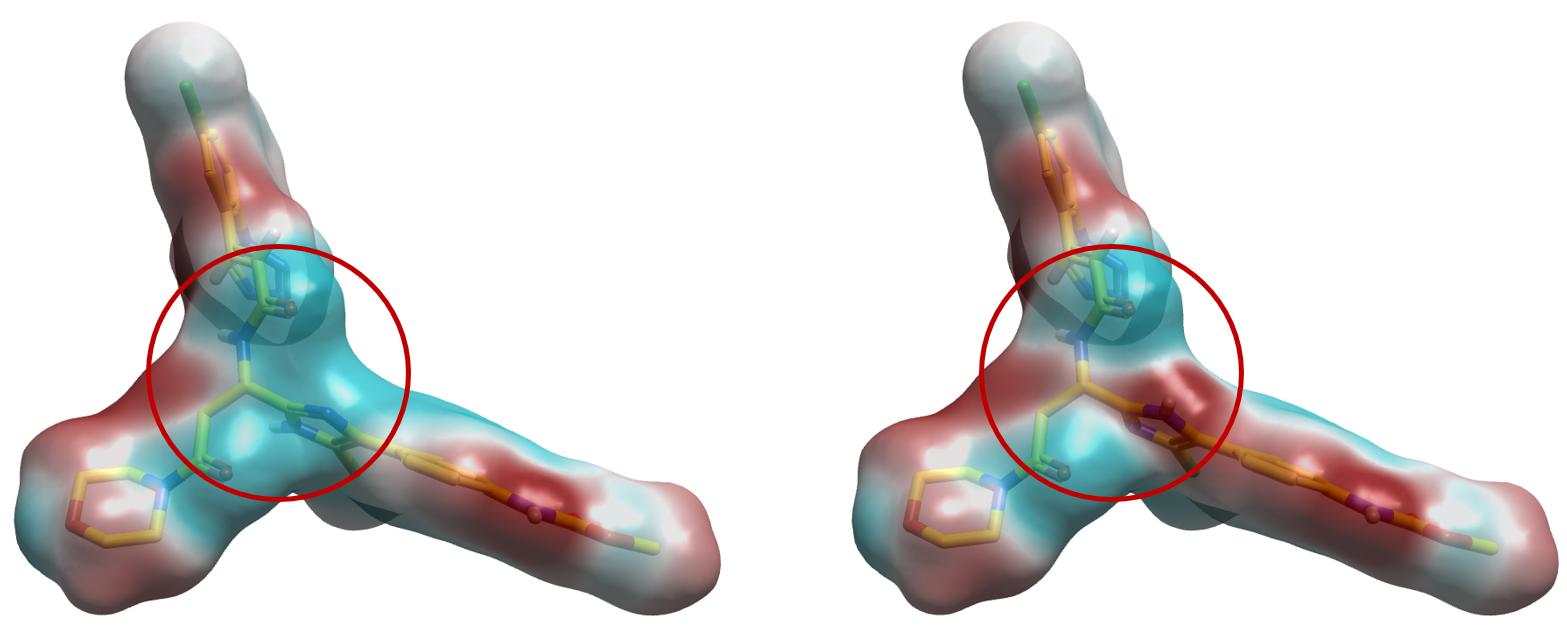
图5. PDB 4Y8Y共晶配体两个质子化状态的MEP分子表面。左:Epsilon位质子化;右:Delta位质子化。
如图5所示,还可以看出在苯基咪唑片段之间也存在分子内静电模式的差异。在苯基咪唑片段之间的N-C-C-C两面角(图6球状高亮原子所示)为26.3°,但是Epsilon质子化异构体的表面静电是互补的(图5左),不利于维持26°的两面角;而Delta质子化异构体的表面静电是互相冲突的(图5右),这与咪唑、苯环不在一个平面是一致的。

图6. PDB 4Y8Y共晶配体苯基咪唑片段的两个质子化状态异构体,两个环之间的两面角(球状原子)为26.3°。左:Delta位质子化;右:Epsilon位质子化。
在B3LYP/6-311+G(d,p)理论水平下计算苯基咪唑两个质子化异构体的生物活性构象与最低能构象的构象能,结果如图7所示。Epsilon异构体的全局最小构象两面角为0°,即苯环与咪唑环在同一个平面,但是生物活性构象与极小点构象能差较小,仅为0.301kcal/mol。Delta异构的全局最小构象两面角为26.1°,与生物活性构象的两面角26.3°基本一样。这说明,Delta异构体比Epsilon异构体的生物活性构象具有更好的构象稳定性。
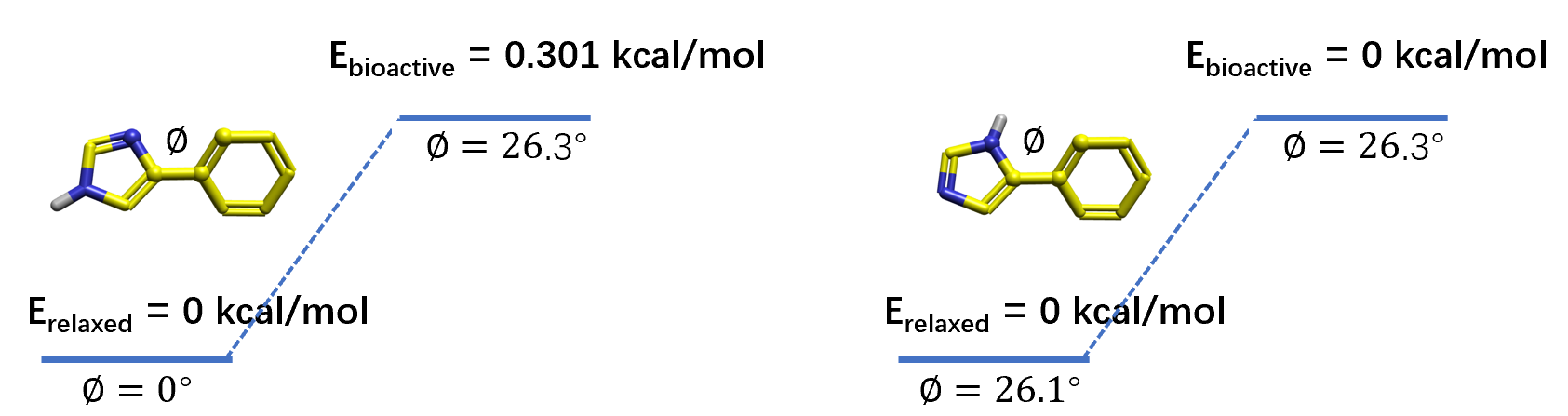
图7. 苯基咪唑片段两个质子化状态异构体生物活性构象与极小点构象的构象能。左:Epsilon位质子化;右:Delta位质子化。
此外,如图8所示,Delta质子化异构体能够形成分子内氢键,而Epsilon质子化异构体不能形成分子内氢键,这导致Delta质子化异构体具有更好的构象稳定性。
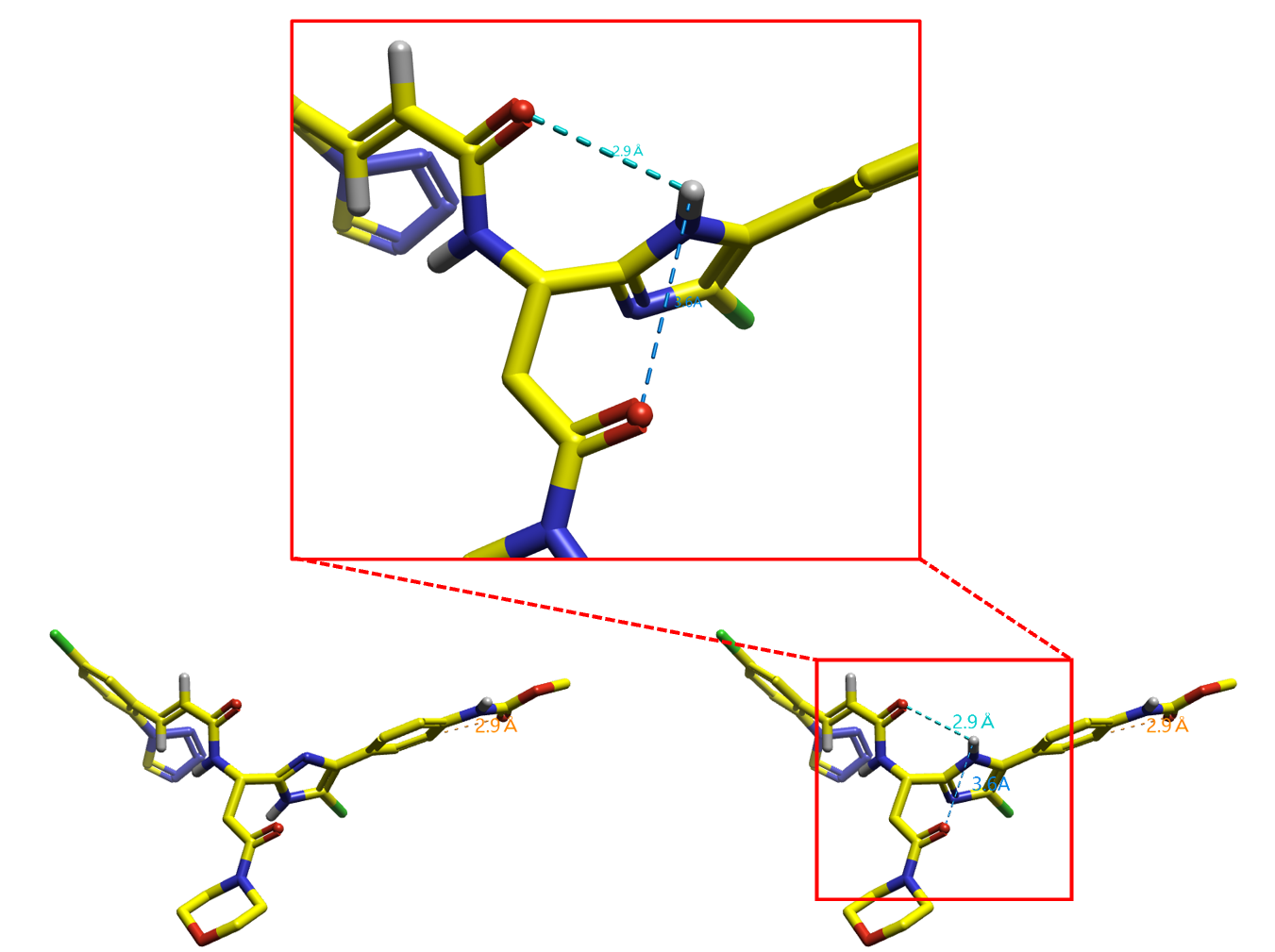
图8. Delta质子化异构体形成了分子内氢键(上)。左下:Epsilon质子化异构体;右下:Delta质子化异构体。
QM计算可以用来考察上述Delta异构体与Epsilon异构体的构象稳定性。在B3LYP/6-311+G(d,p)理论水平分别计算了如图9所示两个异构体片段的电子结构能量。结果表明,Delta异构体(图9左)与Epsilon异构体(图9右)的电子结构能量分别为-473.6850662与-473.6745405(Hatree),前者比后者低了6.6kcal/mol。这说明Delta比Epsilon质子化异构体具有更好的构象稳定性。

图9. 两个异构体片段的化学结构。左:Delta质子化异构体;右:Epsilon质子化异构体。
用大环化锁定生物活性构象
咪唑环2位C与侧链的C-C键是柔性的,在B3LYP-D3BJ/6-311+G(d,p)理论水平对图10所示的Delta质子化异构体片段进行了扭转角分析。结果如图10所示,全局最低能构象的两面角为65°。从4个已知的共晶结构(PDB 4YBY、4YBX、5QQO、5QQP)可以知道,该两面的活性构象分布在103-120°之间,比最低能构象的两面角大了40-60°,电子结构能量也高了不止2kcal/mol。这说明,生物活性构象是高能的,大环化设计可以锁定该构象以提高生物活性。如图2所示,从化合物8到10的大环化设计使得活性提高了78倍。
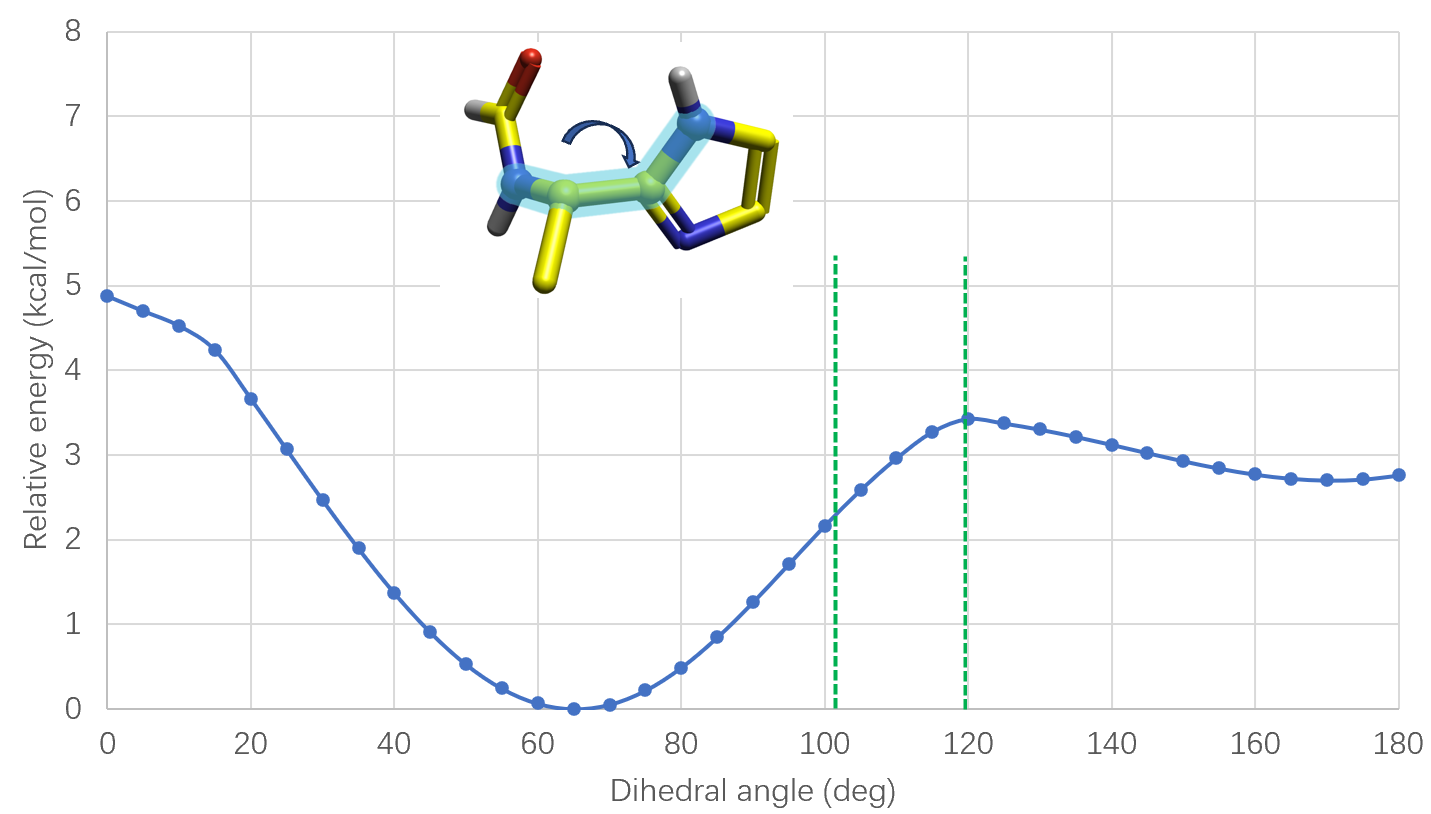
图10. 咪唑2位与其侧链C-C键的扭转角分析。绿色虚线:生物活性构象分布区间。
这个例子也说明了基于频次分布的扭转角分析方法不能代替基于QM的方法。用基于两面角分布频次的扭转角分析方法不能发现这个高能的两面角,而基于QM的扭转角分析方法可以可靠发现的高能生物学构象,从而发现巨大的化合物优化机会,
结论
本文演示了如何用Flare QM的扭转角分析来发现高能的生物学构象并由此发现化合物优化机会。具体地讲,以BMS Factor XIa项目为例,一方面重现了丙烯酰胺高能活性构象的发现,解释了环化对活性33倍的优化效果;另一方面,发现了咪唑2位取代的高能活性构象,解释了大环化对活性78倍的优化效果。此外,还通过QM计算提出咪唑环可能的质子化状态假设,为理解化合物的SAR提供了新的视角。总的来说,Flare QM扭转角分析提供了一个高度可靠的工具以发现巨大的先导化合物优化机会。
文献
- Gilles Ouvry. Why torsions matter. 2o23-11-3. GOuvry@linkedin.
- Clark, C. G., Rossi, K. A., Corte, J. R., Fang, T., Smallheer, J. M., de Lucca, I., Nirschl, D. S., Orwat, M. J., Pinto, D. J. P., Hu, Z., Wang, Y., Yang, W., Jeon, Y., Ewing, W. R., Myers, J. E., Sheriff, S., Lou, Z., Bozarth, J. M., Wu, Y., … Lam, P. Y. S. (2019). Structure based design of macrocyclic factor XIa inhibitors: Discovery of cyclic P1 linker moieties with improved oral bioavailability. Bioorganic & Medicinal Chemistry Letters, 29(19), 126604. https://doi.org/10.1016/J.BMCL.2019.08.008
- Flare™, version 7, Cresset®, Litlington, Cambridgeshire, UK; http://www.cresset-group.com/flare/; Cheeseright T., Mackey M., Rose S., Vinter, A.; Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation J. Chem. Inf. Model. 2006, 46 (2), 665-676; Bauer M. R., Mackey M. D.; Electrostatic Complementarity as a Fast and Effective Tool to Optimize Binding and Selectivity of Protein–Ligand Complexes J. Med. Chem. 2019, 62, 6, 3036-3050; Maximilian Kuhn, Stuart Firth-Clark, Paolo Tosco, Antonia S. J. S. Mey, Mark Mackey and Julien Michel Assessment of Binding Affinity via Alchemical Free-Energy Calculations J. Chem. Inf. Model. 2020, 60, 6, 3120–3130
- Flare QM. http://www.molcalx.com.cn/cresset/flare/flare-qm
联系我们,获取软件试用
想要亲自尝试Flare基于QM的扭转角分析,请申请免费的Flare试用版。



