摘要:Alexander等人借助自由能计算、能量拆解预测了Hot spot、并用实验确认了其中3个氨基酸残基;以Hot spot为模板,用ROCS对ZINC11进行虚拟筛选,发现了苗头化合物7.18;再通过相似性搜索,发现活性提高的化合物7.44,这是首个RUNX1/ETO四聚体PPI小分子抑制剂。本策略也适于其它PPI抑制剂的发现。
文献来源:Metz A, Schanda J, Grez M, Wichmann C, Gohlke H. From determinants of RUNX1/ETO tetramerization to small-molecule proteinprotein interaction inhibitors targeting acute myeloid leukemia. J Chem Inf Model. 2013;53(9):2197-2202.
一. 背景
作者Metz Alexander等在前期发现了一个128mer融合蛋白,它包含了一个完整的NHR2结构域(NC128)并可以干扰RUNX1/ETO四聚体形成、抵消白血病的细胞特征。然而这样的融合蛋白缺乏合适ADME特性,因而需要开发与融合蛋白具有同样的作用模式但ADME更好的小分子PPI(Protein-protein interaction, PPI)抑制剂。
NHR2四聚体是一个二聚体的对称性二聚体,每个二聚体含有两个α-螺旋单体,头尾交互摆放形成一个4-螺旋束(图1)。靶向这样的PPI靶标困难难处在于:尺寸大、没有典型的结合口袋、PPI作用非常稳固。然而并非无解,靶向含有Hot spot的结合口袋区域已经在多个案例中证明是一种成功的PPI抑制剂设计策略。
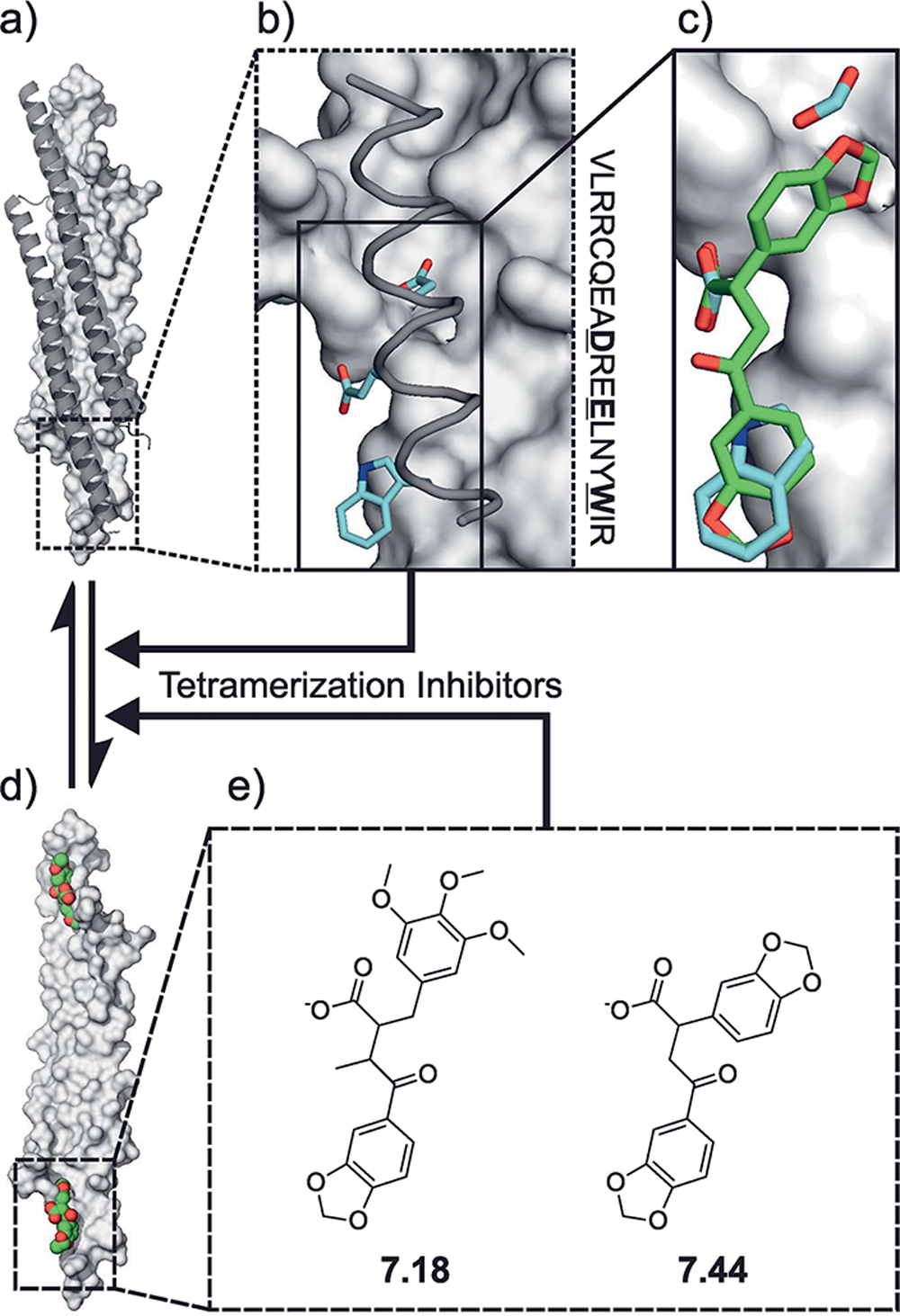
图1.NHR2四聚体抑制剂的识别
二. 虚拟筛选流程
1. Hot Spot的预测与确认
计算结合自由能,对能量进行拆解,分析每个残基对NHR2二聚体-四聚体过渡时(四聚体化)的能量贡献,推测五个残基(W498,W502,D533, E536, W540)对四聚体的稳定化起到重要的贡献(”Hot spot”)。对这五个残基进行丙氨酸定点突变,发现可以阻止四聚体的形成但不影响二聚体的形成。
2. 结合位点的确认
扫描NHR2二聚体-二聚体的结合界面,发现最大、最深的口袋为269A3, 与常规的配体结合位点或其它蛋白-蛋白相互作用界面比较,该口袋还是偏浅。考虑到该口袋与Hot spot靠的很近,而且包含有Hot spot D533,因此作者认为这个口袋对蛋白-蛋白的高亲合力相互作用非常重要: 这是NHR2界面上结构模组(Motif)的必要部分,适于干预。
3. 虚拟筛选方案的确认
基于结构的药物设计策略已经成功地用于发现PPI抑制剂,大部分设计是从多肽或配体结合的复合物结构出发。作者决定从NHR2四聚体的结构模组(Motif)出发采用基于结构的策略去发现PPI抑制剂。于是进一步对Hot spot进行分析,发现残基D533、E536、W540是合适的模板Motif(图1 b)。设计这三个Hot spot的模拟物或许可以抑制NHR2四聚体。
在这个模板Motif的基础上,作者认为NHR2结构域上的一段18mer肽(P1)可以作为NHR2四聚体抑制剂,它含有-NH2端成&alpha-螺旋的mer12(Ac-EADREELNYWIR-NH2),将包含Hot spot的结构模板包含在内。实验表明,mer18(P1)对NHR2四聚体形成的抑制活性IC50=250μM。这证明了NHR2四聚体化可以被比其结构域小的多的分子抑制。
对P1上的三个hot spot残基进行丙氨酸突变或任意两个进行突变,发现Hot spot的突变都会导致P1的抑制活性丧失。这个实验结果进一步证明了这三个Hot spot残基的重要性,用复合物晶体结构中这个三个残基的位置与取向做为虚拟筛选条件寻找模拟Hot spot相互作用的潜在PPI抑制剂。
4. 虚拟筛选
虚拟筛选采用OpenEye的ROCS来进行,查询结构(Query)由Hot spot残基D533、E536、W540组成。用ROCS搜索了ZINC11数据库的~6 x 106化合物。ROCS将数据库化合物叠合到查询结构上,比较形状与颜色(氢键供体、受体、负电中心、正电中心、疏水球与芳香环等性质)。作者总共建立了3个搜索条件,每个搜索取打分最高的1000个化合物,最后总共挑选了80个化合物进行实验测试。
5. 实验验证
7个化合物显示出显著的抑制活性,化合物7.18(见图1 e)在1mM的抑制率达到了80.3%。图2展示了Hot spot与化合物7.18的ROCS叠合效果。
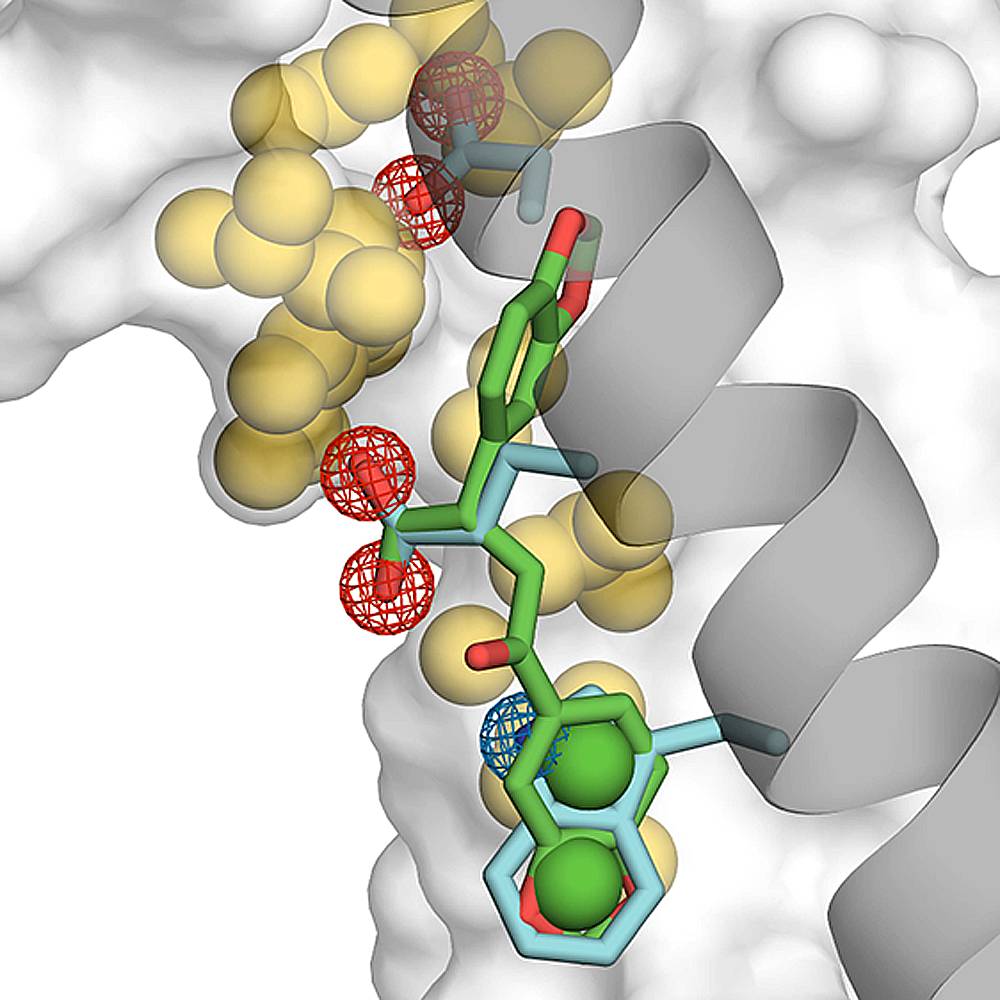
图2. Hot spot与化合物7.18的ROCS叠合效果
6. 苗头化合物的拓展
采用指纹图谱相似从化合物数据库里搜索化合物7.18的衍生物,发现化合物7.44(见图1 e)显示出显著改善的体外活性,IC50=630μM。
比较7.18、7.44与Hot spot的ROCS叠合结果,可以解释为什么活性得以改善(见图3)。比起化合物7.18,一方面7.18的三甲氧基被替换后的化合物7.44与蛋白排斥体积的重合更少;另一方面7.44的结构更短、更紧凑、更少的柔性,因此在熵上更具优势。
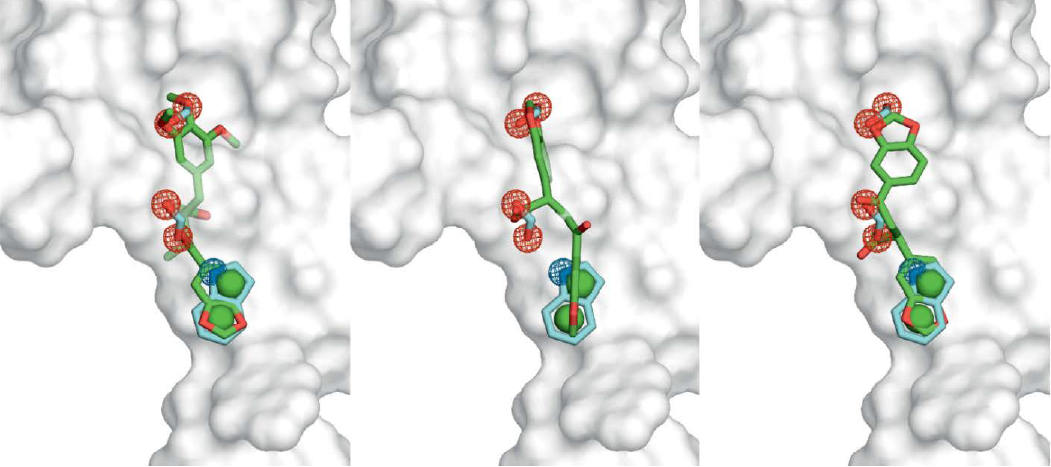
图3.左:7.18与查询结构(III);中:7.44与查询结构(I);右:7.44与查询结构(III)
三. 亮点总结
- 采用自由能计算预测Hot spot,并获得实验确认,确定了Hot spot
- 将复合物结构里Hot spot作为ROCS虚拟筛选的查询结构进行虚拟筛选
- 对苗头化合物进行拓展,用相似性搜索发现活性改善的化合物
四. 相关主题(点击标题可跳转到原文)
- Ligandscout案例 | HIV-1整合酶与LEDGF/p75蛋白-蛋白相互作用小分子抑制剂的发现
- Ligandscout案例 | 第四军医大学刘吉元等人首次发现Mcl-1-PUMA调节小分子
- 结合自由能计算与能量拆解:Flare/WaterSwap
晶状体上皮衍生生长因子(LEDGF)或转录共活化因子p75(LEDGF/p75)在HIV整合中起到至关重要的作用。HIV-1整合酶(Integrase,IN)与它的细胞因子LEDGF/75之间的蛋白-蛋白相互作用或许可以作为开发全新抗HIV药物的靶点。在本研究中,用Ligandscout基于结构的策略开发了一个HIV-1的IN-LEDGF/p75相互作用的基于结构的药效团模型。并用该模型虚拟筛选CHIME数据库、发现了一个苗头化合物CHIBA-3002。对苗头化合物进行进一步优化:采用理性设计、合成了4个化合物、并进行了生物活性评价。最后发现一个小分子化合物CHIBA-3003可以在uM水平干扰HIV-1的IN-LEDGF/p75相互作用。这是首个专一作用于HIV-1的IN-LEDGF/p75蛋白-蛋白相互作用并有活性的化合物。本研究的靶点不同于现有抗病毒药物,为抗HIV药物开发开辟了全新方向。
尽管在寻找抗癌小分子的道路上已经付出巨大的努力,但是发现高效、安全的小分子依旧是个挑战,因为目前的许多药物还是缺乏足够的选择性、还不能专一地攻击肿瘤细胞。两个凋亡蛋白骨髓细胞白血病-1(Myeloid-cell leukemia-1,Mcl-1)与p53上调调控子(p53 upregulated modulator of apoptosis, PUMA)的蛋白-蛋白相互作用(PPI)界面(Mcl-1-PUMA)已经被证实可以作为癌症治疗的靶点,干扰Mcl-1-PUMA的结合可以减少癌细胞的存活并且保护正常细胞免于凋亡,因此Mcl-1-PUMA调节分子有可能是理想的高效、安全抗癌药物。然而,目前尚未发现以Mcl-1-PUMA为靶标的小分子。第四军医大学的刘吉元等人通过药效团建模、虚拟筛选以及实验验证等首次发现了作用于Mcl-1-PUMA的双功能小分子,实验表明,该小分子不仅可以减少PUMA-依赖的细胞凋亡,而且还同时降低癌细胞中Mcl-1介导的抗凋亡活性。本研究为开发高效、安全的抗癌药物提供了新的研究方向。
WaterSwap是分子对接软件Flare的一个计算模块,专门用来计算配体-蛋白的结合自由能,它可以:1)研究配体结合的能量学特点;2)将结合能分解为每个残基的贡献以发现优选的配体相互作用模式;3) 计算一系列化合物的结合自由能(ΔG of binding)以帮助您优选配体,或者比较几种结合模式以发现最可能的结合模式。



