用XED力场分析激酶抑制剂的C─H···O=C氢键
摘要:量子化学计算表明,C─H···O氢键相互作用强度相当强,大约是N─H···O氢键相互作用强度的一半。最近的研究表明,几乎所有的FDA批准上市的激酶抑制剂均与激酶铰链存在C─H···O型氢键相互作用。经典的氢键相互作用不是激酶抑制剂与铰链相互作用所必须的,也可仅通过C─H···O型氢键相互作用而保留抑制剂的活性。Flare的XED静电分析工具可以帮我们非常方便地分析这种非经典的C─H···O氢键相互作用。
肖高铿/2022/01/25
1. 关于XED力场
静电势是配体结合的关键,由于有意义的静电势的计算与可视化相当难而被人忽视。之前,我们已经展示过静电可高效地用于骨架跃迁、虚拟筛选、配体叠合与SAR解释。配体的静电可通过一个探针原子绕着配体构象计算相互作用能而获得。探针原子与配体之间的相互作用能定义了空间中一个点的静电特征。如图1所示,可以将静电势可视化为等值面。Cresset的XED力场通过原子偏心电荷模型可以详细地描述分子的静电特征[1],XED力场最为关键的是它能够将偏电荷的π与σ成分分离开来。
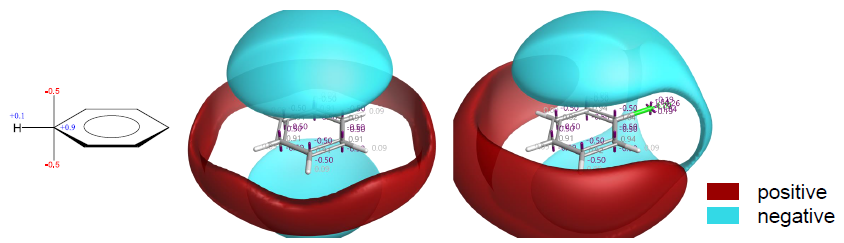
图1. 苯的XED模型、外加的点电荷与静电
XED力场不仅可以用来算配体的静电,还可以用来计算受体的的静电,以及配体与受体之间的静电互补性、蛋白-配体相互作用等分子设计领域[2,3]。在本文中,我们将用它来解释激酶抑制剂的C─H···O=C氢键相互作用。
2. C─H···O=C氢键相互作用
蛋白激酶抑制剂疾病谱广泛,包括诸如神经退行性疾病、炎症、心血管与癌症等重大疾病。根据Derewenda等人统计[4],到2020年为止,FDA大约批准了48个激酶抑制剂用于治疗各种疾病,其中适应症最多的为肿瘤,少部分为炎症与自身免疫疾病。激酶抑制剂设计的难点在于选择性:人类激酶谱包含了517个蛋白激酶,它们有着同源的激酶结构域,特别是大部分激酶抑制剂靶向的ATP结合口袋,具有显著的氨基酸与结构保守性。大部分的上市激酶抑制剂为Type I抑制剂,也就是与ATP竞争性结合的抑制剂。激酶的ATP结合口袋对腺嘌呤具有选择性是由于它具有一个特别的氢键 (H-bond) 网络,它涉及一个被称为“铰链(hinge)”的溶剂暴露骨架,是一个连接激酶催化结构域N-和C-末端亚结构域的短寡肽。铰链包含三个氨基酸,城延长展的β折叠二级构象,根据它们在“看门人(gatekeeper)”残基下游序列中的位置将它们表示为gk+1、gk+3和gk+3。ATP的腺嘌呤与铰链结合的方式是腺嘌呤的N10氨基做为供体与gk+1的主链羰基形成氢键,而N1氮作为供体与gk+3主链酰胺形成氢键 。第三个相互作用是非经典氢键,其中腺嘌呤的C2-H提供一个氢与gk+3的主链羰基氧形成氢键,如图2所示。
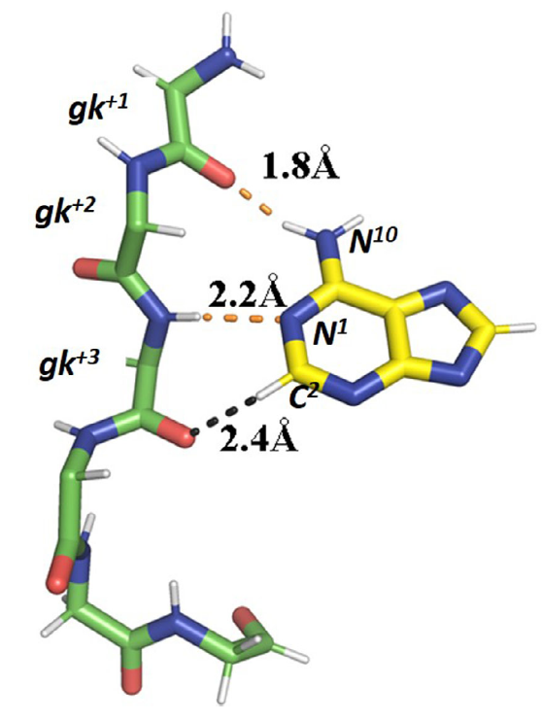
图2. 以AMPPNP与RSK2 N-端结构域的复合物晶体结构PDB 3G51(分辨率为1.8 Å)为例,说明ATP的腺嘌呤部分与激酶的铰链寡肽之间的氢键相互作用[4]
大多数抑制剂骨架模拟了腺嘌呤与铰链的相互作用模式,通过氢键供体与受体,与gk+3、gk+1的羰基以及gk+1的酰胺发生氢键相互作用。虽然Xing等人[7]对这些典型的氢键相互作用进行了相当详细的分析,但仅限于涉及电负性供体和受体基团的典型相互作用。也有许多的文献称在激酶-抑制剂复合物中发现了C─H···O=C型氢键(也被称为弱氢键)[7,8],但对之进行系统性的研究却甚少。Derewenda等人[4]对FDA批准上市的29个激酶抑制剂与其靶标的共晶结构进行了系统分析,结果发现在抑制剂与铰链羰基之间广泛存在着C─H···O=C氢键相互作用。此外,作者还注意到在FDA批准的激酶抑制剂中发现的骨架类型非常有限,在利用激酶铰链区内的羰基上是一致的:不仅作为典型氢键的受体,而且还是弱氢键的受体,其中C-H基团通常来自芳环或与电负性氮原子相邻。
作者一开始在PDB中确定了31个相关的晶体结构。 其中,21 个结构为 1 型抑制剂(竞争性抑制剂),7个为II型抑制剂(与非活性激酶构象结合),3 个属于 VI 型抑制剂(共价、不可逆抑制剂)。 除了VI 型抑制剂cobimetinib因为没有与铰链接触而被排除在分析之外,对其余的30个抑制剂-激酶复合物结构进行了进一步的分析(参见图 2)。
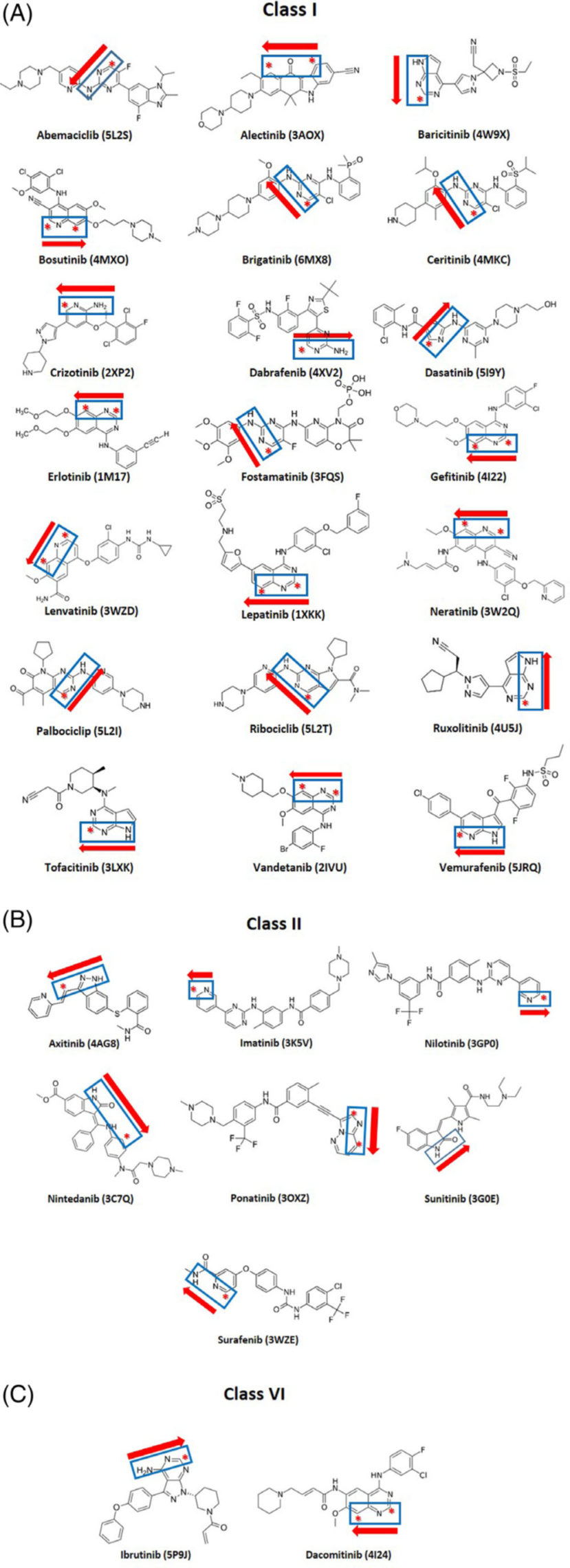
图3. 30个FDA批准上市的激酶抑制剂的化学结构。 蓝色框高亮部分为与激酶铰链片段形成氢键相互作用的供体/受体原子;红色星号表示碳原子,它作为C─H···O=C氢键中的供体;箭头表示氢键基团相对于铰链的方向,即箭头的方向是从gk+1到gk+3。 其中(A)为I型抑制剂; (B)为II型;(C)为VI型抑制剂。图片来自文献4。
显然,C─H···O=C氢键在上市的激酶抑制剂里广泛存在,本文的主要目的是用Flare的XED力场来分析、可视化这种C─H···O=C氢键相互作用。
3. 用XED力场发现、可视化分析C─H···O=C氢键相互作用
3.1 可视化分析CDK6抑制剂的C─H···O=C氢键
Flare提供了配体表面计算工具(见图4)可以用分析正、负静电势、疏水势以及势差。氢键的本质是静电相互作用,因此可借助Flare的XED力场来进行可视化分析。
图4. Flare提供了配体的表面分析工具,包括正、负XED力场的静电分析工具。
以上市的CDK6激酶抑制剂Ribociclib为例,其共晶结构为PDB 5L2T。在Flare里下载共晶结构,用protein prep进行标准的结构准备,接着用Contact分析相互作用,再用LIGAND选项卡下的+ev分析配体的正静电势,如图5所示。
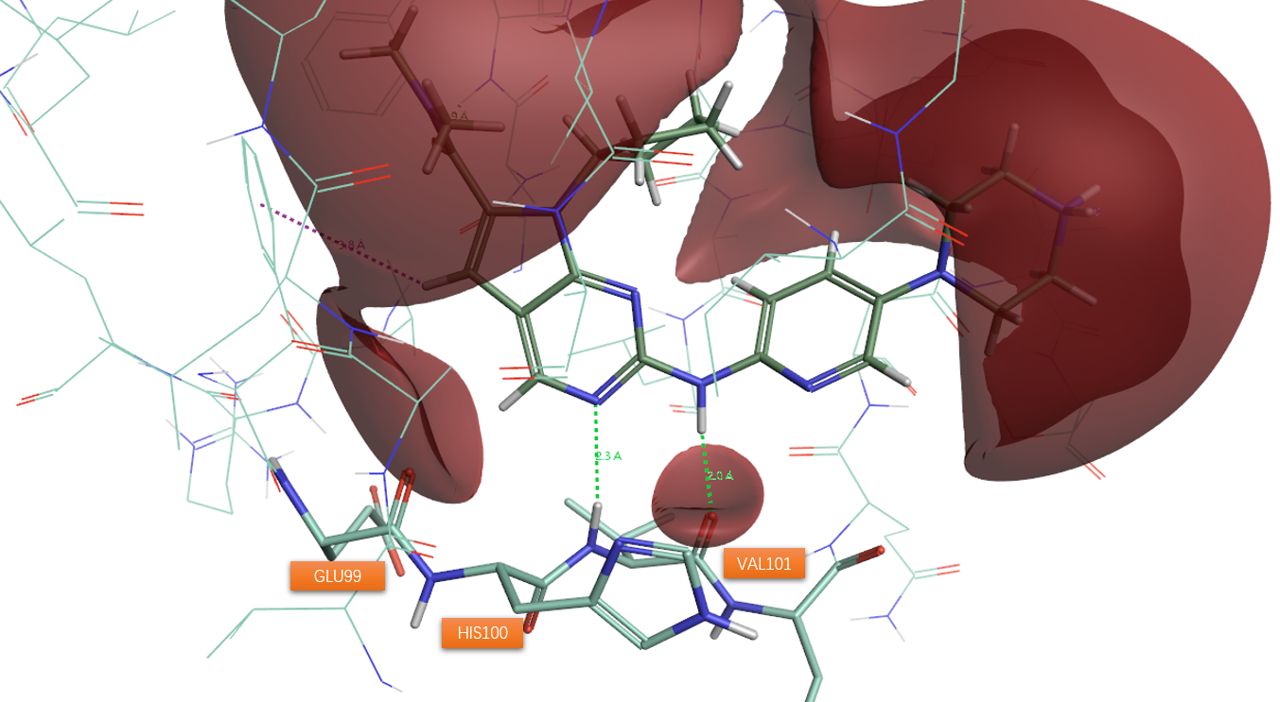
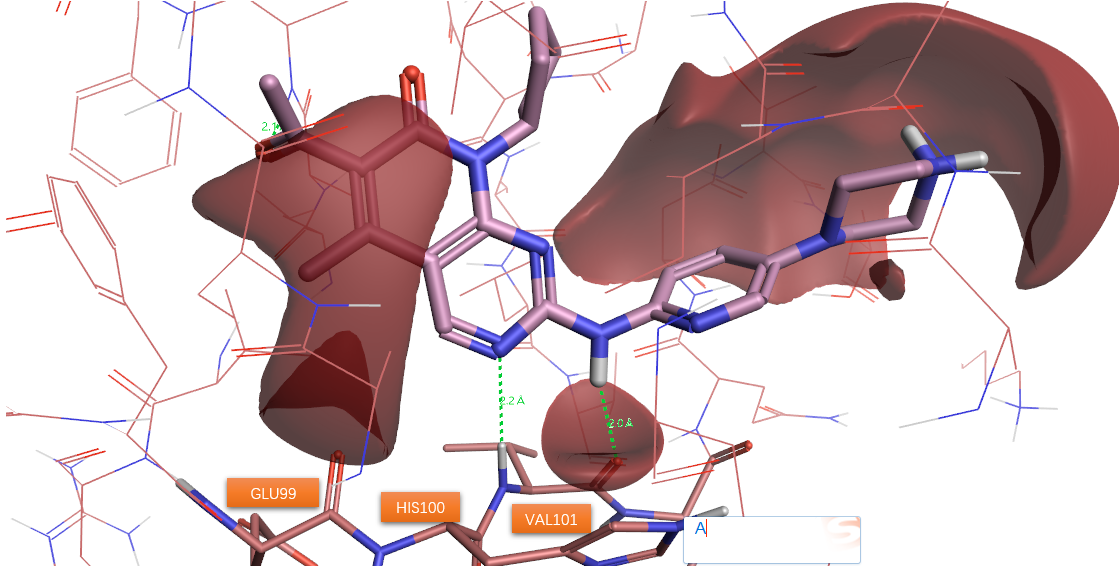
图5. Ribociclib与CDK6的相互作用示意图。其中红色等值图XED静电相互作用2.0等值面。
如图5所示,抑制剂Ribociclib与CDK6铰链gk+3残基VA101的酰胺NH供体以及羰基氧受体发生了经典的氢键相互作用。在沿着配体连接两环的N-H氢键供体的方向上,有着一个红色区块与VAL101羰基氧重合,这与配体N-H···O=C-Val氢键相互作用是一致的。配体嘧啶环上的氮作为氢键受体与HIS100的酰胺NH发生经典的氢键相互作用。在配体嘧啶环受体N邻位C-H方向上,有一个红色正静电势区块与蛋白的gk+1残基GLU99羰基氧重合,这意味着这这个原子对上发生了C-H···O=C非经典氢键相互作用。
图6. Palbociclib与CDK6的相互作用示意图。其中红色等值图XED静电相互作用2.0等值面。
在另一个CDK6抑制剂Palbociclib上,我们也观察到了相似的作用模式。如图6所示,分析共晶结构5L2I,铰链区gk+1残基VAL99的羰基氧与配体的正静电势重合,而这正静电势正是在配体的嘧啶环N邻位C-H轴向上,因此可认为发生了C-H···O=C氢键相互作用。配体的嘧啶环N作为受体与gk+2残基HIS100的酰胺NH发生经典的氢键相互作用。而配体连接两环的NH作为供体,与铰链gk+3残基VAL101的羰基氧发生经典的N-H···O=C氢键相互作用。
3.2 可视化分析CDK2抑制剂的C─H···O氢键
图7. CDK2抑制剂相互作用模式
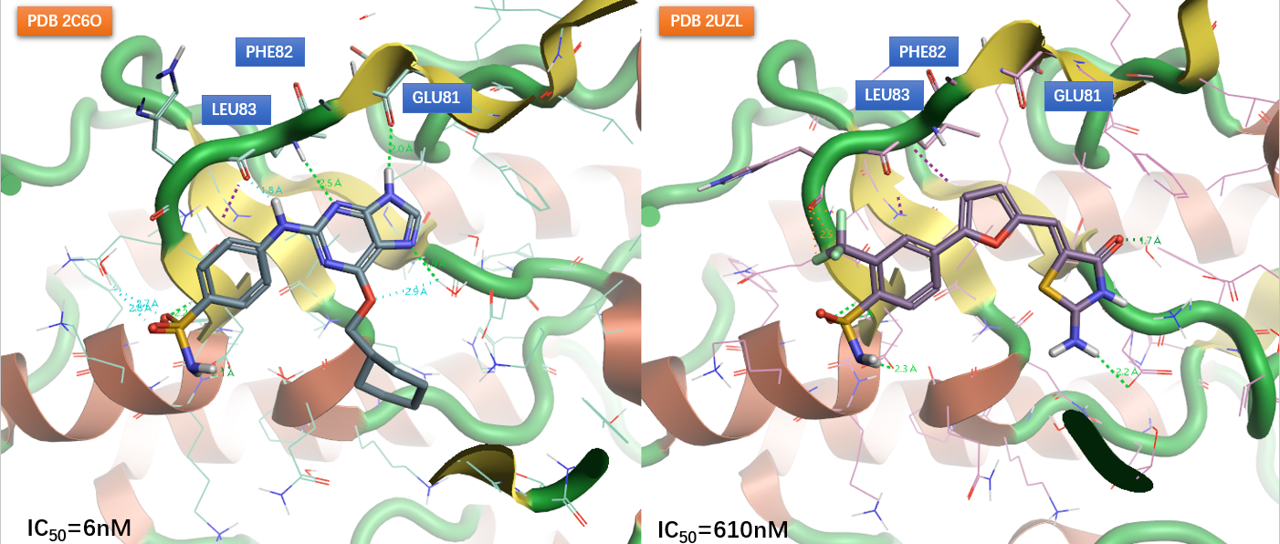
图7左展示了一个CDK2抑制剂与靶标之间复合结构的相互作用模式(PDB 2C6o)。该抑制剂的一个连接两环的NH作为氢键供体与铰链gk+3残基LEU83的羰基氧发生氢键相互作用;一个芳香氮作为受体与gk+2残基LEU82的酰胺NH发生氢键相互作用;还有一个配体咪唑环上的NH作为供体与gk+1残基GLU81的羰基氧发生氢键相互作用。相比之下,以经典氢键相互作用模式观察PDB 2UZL的配体,看起来该抑制剂与激酶铰链之间不存在任何的氢键相互作用,如图7右所示。
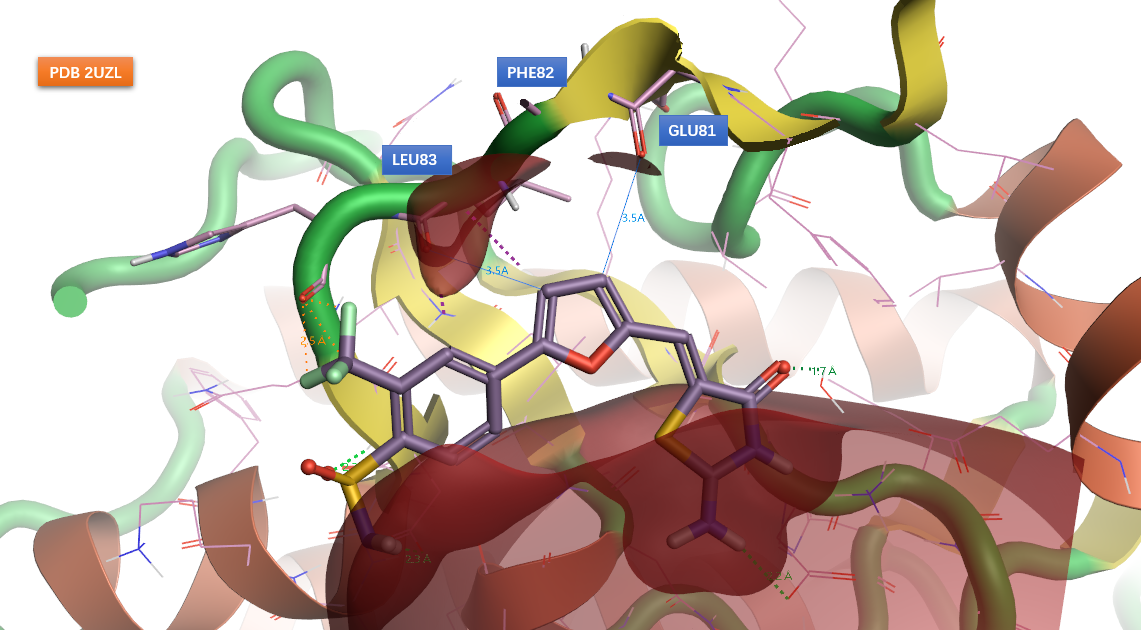
图8. PDB 2UZL的CDK2抑制剂静电
但是借助XED静电场,如图8所示,可以看到在沿着呋喃环的两个C-H轴向上,配体的两个正静电势区分别与铰链的gk+1残基GLU81、gk+3残基LEU83的主链羰基氧重合;并且从碳到氧的距离均为3.5Å,处于发生接触的距离之内。因此我认为配体呋喃环C-H与gk+1、gk+3的羰基氧发生了C─H···O=C氢键相互作用。根据Vargas等人[11]的量子化学计算研究,在真空中C─H···O=C氢键相互作用强度大约是N-H···O氢键相互作用强度的一半;此外,2UZL的配体比2C6O的配体少了一个与gk+1酰胺NH之间的氢键相互作用;这两个因素可能是前者比后者活性差的主要原因。2UZL的算例也说明了,I型抑制剂与铰链之间经典的氢键相互作用模式不是必须的。
4. 小结
量子化学计算表明,C─H···O=C氢键相互作用强度相当强,大约是N─H···O氢键相互作用强度的一半。几乎所有的FDA批准上市激酶抑制剂均与激酶铰链存在C─H···O=C型氢键相互作用。经典的氢键相互作用不是激酶抑制剂与铰链相互作用所必须的,也可仅通过C─H···O=C型氢键相互作用而保留抑制剂的活性。Flare的XED静电分析工具可以帮我们非常方便地分析这种非经典的C─H···O=C氢键相互作用。
5. 拓展应用
化合物的ADME与分子的lipinski氢键受体与供体数相关,经典的氢键受体/供体数量越大,极性表面积也越大,吸收、血脑屏障透过性越差。通过C─H···O=C氢键相互作用代替经典的氢键相互作用可以实现保留活性而改善化合物的ADME,尤其适合于先导化合物优化。
Flare提供了python界面,可以批量的计算一个氢键受体或供体处配体的静电势以便快速、高通量地进行分子对接后处理、将满足C─H···O=C氢键相互作用配体的过滤与识别出来[12]。
还可以借助BLAZE或SPARK软件,将XED静电用于高通量虚拟筛选,发现新型骨架先导化合物。
6. 文献
- Vinter, J. G. Extended Electron Distributions Applied to the Molecular Mechanics of Some Intermolecular Interactions. J. Comput. Aided. Mol. Des. 1994, 8 (6), 653–668. https://doi.org/10.1007/BF00124013.
- Cheeseright, T.; Mackey, M.; Rose, S.; Vinter, A. Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation. J. Chem. Inf. Model. 2006, 46 (2), 665–676. https://doi.org/10.1021/ci050357s.
- Bauer, M. R.; Mackey, M. D. Electrostatic Complementarity as a Fast and Effective Tool to Optimize Binding and Selectivity of Protein–Ligand Complexes. J. Med. Chem. 2019, 62 (6), 3036–3050. https://doi.org/10.1021/acs.jmedchem.8b01925.
- Derewenda, Z. S.; Hawro, I.; Derewenda, U. C─H⋯O Hydrogen Bonds in Kinase-Inhibitor Interfaces. IUBMB Life 2020, 72 (6), 1233–1242. https://doi.org/10.1002/iub.2282.
- Manning G, Whyte DB, Martinez R, Hunter T, Sudarsanam S.The protein kinase complement of the human genome. Science. 2002;298:1912–1934.
- Fabbro D, Cowan-Jacob SW, Mobitz H, Martiny-Baron G.Targeting cancer with small-molecular-weight kinase inhibitors. Methods Mol Biol. 2012;795:1–34.
- Xing L, Klug-Mcleod J, Rai B, Lunney EA. Kinase hinge binding scaffolds and their hydrogen bond patterns. Bioorg Med Chem. 2015;23:6520–6527
- Pierce AC, Sandretto KL, Bemis GW. Kinase inhibitors and the case for CH…O hydrogen bonds in protein-ligand binding. Proteins. 2002;49:567–576.
- Pierce, A. C.; Ter Haar, E.; Binch, H. M.; Kay, D. P.; Patel, S. R.; Li, P. CH⋯O and CH⋯N Hydrogen Bonds in Ligand Design: A Novel Quinazolin-4-Ylthiazol-2-Ylamine Protein Kinase Inhibitor. J. Med. Chem. 2005, 48 (4), 1278–1281. https://doi.org/10.1021/jm0492249.
- Panigrahi SK. Strong and weak hydrogen bonds in protein ligand complexes of kinases: a comparative study. Amino Acids. 2008;34:617–633.
- Vargas, R.; Garza, J.; Dixon, D. A.; Hay, B. P. How Strong Is the C α −H···OC Hydrogen Bond? J. Am. Chem. Soc. 2000, 122 (19), 4750–4755. https://doi.org/10.1021/ja993600a.
- 肖高铿. 分子对接后处理——与指定残基发生特定相互作用配体的过滤. 墨灵格的博客. 2021-11-02. http://blog.molcalx.com.cn/2021/11/02/docking-post-filtering.html