摘要:本文演示了如何将Ligandscout的基于片段虚拟筛选与AutoT&T 2联合使用,实现化合物的从头设计。首先用Ligandscout进行基于结构的药效团模型识别;再将选定的药效团特征设置为optional进行基于片段的虚拟筛选;最后用AutoT&T 2将命中的片段链接到先导化合物的保留片段上生成全新的化合物。
肖高铿 2018-08-31
1. 前言
基于片段的虚拟筛选是进行结构替换的一种重要策略。Ligandscout可以通过对药效团元素设置optional来实现对片段数据库进行虚拟筛选,发现可以替换原有先导化合物感兴趣部分的片段。本教程以图1化合物的CDK2抑制剂(PDB 1KE7的共晶配体)为例,演示如何用药效团技术对片段数据库虚拟筛选发现能够替换高亮部分的潜在片段。
Figure 1. CDK2抑制剂结构(PDB: 1KE7), 需要替换的部分已用高亮显示
AutoT&T 2是一款高效的药物分子从头设计软件,它可以从基于结构的虚拟筛选(分子对接,药效团技术)命中的化合物中识别对结合有利的片段、并将其裁剪、移植到先导化合物上进行全新结构设计。AutoT&T也可将Ligandsdcout基于片段虚拟筛选命中的片段分子连接到先导化合物需要保留的片段上。将Ligandscout与AutoT&T两者结合可以实现更加高效的药物分子从头设计。因此,本教程的另一目的是演示如何联用AutoT&T 2与Ligandscout进行这种高效的药物分子设计。
2. 基于片段的虚拟筛选
2.1 基于结构的药效团模型识别
在Ligandscout的Structure-based界面的PDB代码框里输入1KE7,点击Download,再点击Create Pharmacophore按钮生成基于结构的药效团模型。具体操作过程参考:ligandscout教程–基于结构的药效团识别。
2.2 设定可选的药效团特征(元素)
在本例中先导化合物要被替换的部分对应于Figure 2药效团特征4、5、6覆盖的部分。因此,虚拟筛选时仅需要对4、5、6号药效团特征进行匹配(必须匹配),而将1、2、3号药效团特征设为optional(即可匹配、也可以不匹配)。按住鼠标Ctrl键,鼠标点击图2中标号为1、2、3的药效团特征以便选中这个三个特征,再点击菜单Pharmacophore | Mark Features as Optional。现在我们有了个可以进行部分匹配的药效团模型。
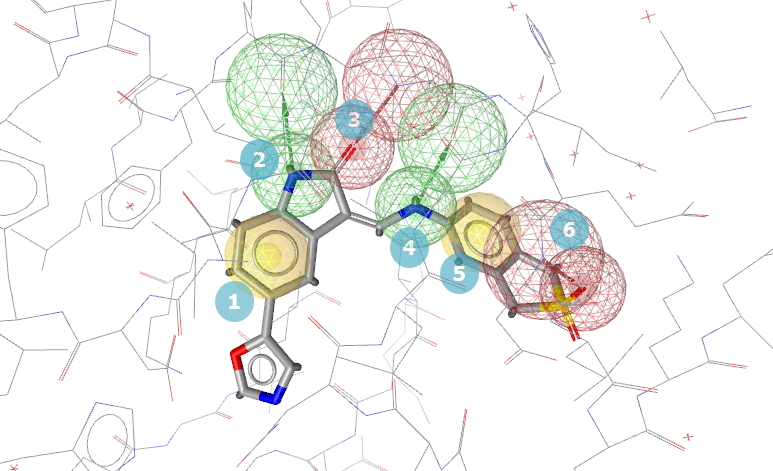
Figure 2. 从PDB 1KE7识别的CDK2抑制剂药效团模型
2.3 虚拟筛选
将生成的药效团模型复制(copy)到虚拟筛选模块下,在虚拟筛选模块里导入片段数据库进行虚拟筛选。更详细的流程参见:Ligandscout教程–基于药效团的虚拟筛选。
将虚拟筛选命中的化合物导出(File | Save As| .sdf)为SDF文件vs_hit.sdf。
2.4 结果分析
在Structure-based模块里,插入2.3的虚拟筛选结果(File|insert|vs_hit.sdf),可以发现命中的化合物(灰色)基本覆盖在标号为4、5、6的药效团特征上(Figure 3)。在分子表单区可以对每个片段打分计算:结合亲和力、药效团匹配度等等。还可以在表单里点击一个具体的片段以便详细观察该片段与药效团的匹配性,与1KE7配体的重合程度等以便考虑是否可以将一个片段代替原有的片段(Figure 4)。
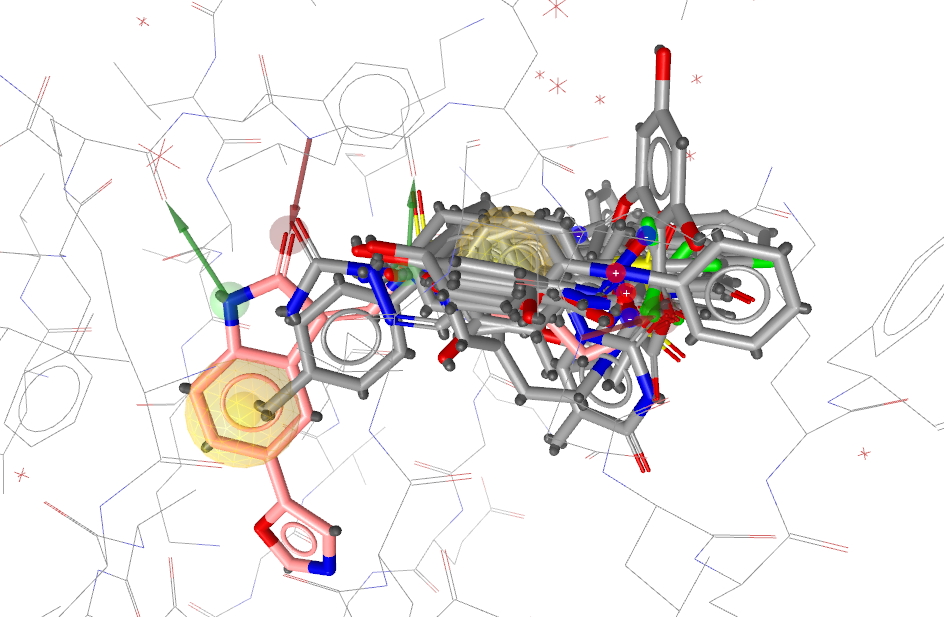
Figure 3. 虚拟筛选结果:将命中的29个化合物(灰色)与原有化合物(粉红色)比较
如果发现药效团、化学键等各方面都匹配的化合物,可以设置标记以便导出感兴趣的片段,并可以通过键的操作生成新的化合物。这个生成新化合物的过程还可以用AutoT&T 2全自动完成。
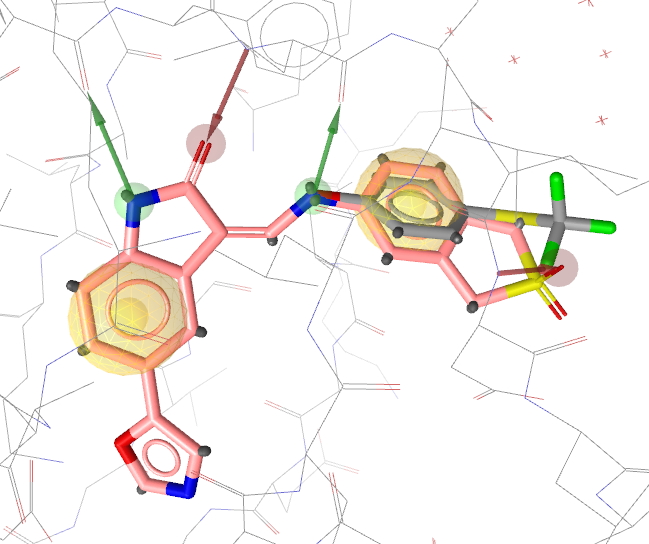
Figure 4. 片段与原有化合物叠合良好的实例:不仅药效团匹配,而且键也很匹配
3. 与AutoT&T 2联用设计全新的化合物
AutoT&T 2是中国科学院上海有机所王任小课题组开发的高效药物分子从头设计软件,可以分析虚拟筛选命中的结构、裁剪结合亲和力高的片段并移植到原有化合物上生成活性更高的化合物,具体介绍见:AutoT&T 2.0-高效的药物分子从头设计软件。
将CDK2的1KE7蛋白结构部分保存为protein.pdb,将抑制剂保存为lead.sdf。用视图软件打开lead.sdf文件,确认键断裂位点的索引号。在本例中,我们计划要将17、18号的键断开(Figure 5),17号原子的(Figure 5. 黄色)那一边片段要保留,而18号原子(Figure 5. 红色)那一边的基团要被替换。
Figure 5. CDK2抑制剂结构(PDB: 1KE7), 17、18号原子间的键要断开,黄色边的基团保留而红色边的基团要被替换。
根据上面的规划,键入如下命令,即可用AutoT&T 2自动开始从虚拟筛选结果里裁剪基团、并移植到先导化合物保留片段的原子上,生成全新的化合物:
1 | $CCBGKIT_BIN/GrowLeadOpt -l lead.sdf -p protein.pdb -vs vs_hit.sdf -c '(17,18)' -o results.sdf |
本例中AutoT&T 2生成了9个全新的化合物(Figure 6),可以将结果导入到Ligandscout里或Flare等视图软件进一步评估。
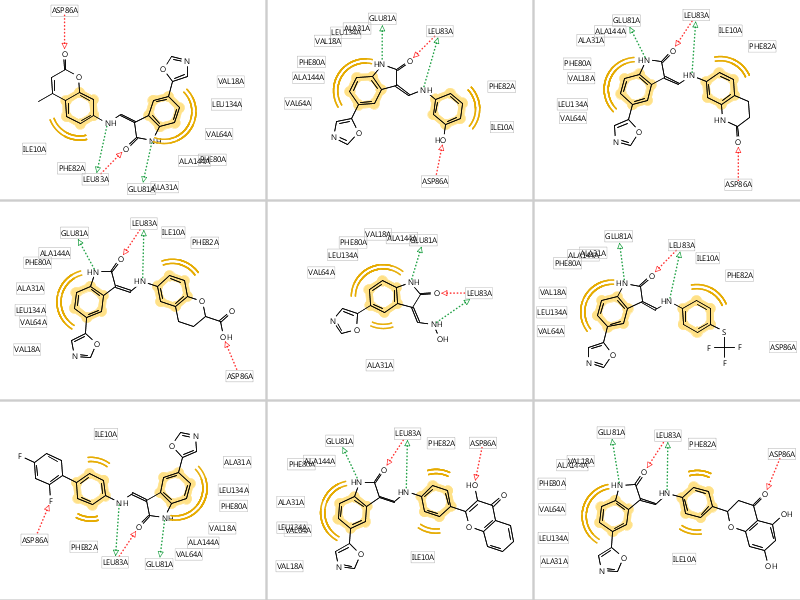
Figure 6. AutoT&T 2生成的9个全新结构
4. 小结
在本教程中,我们演示了:
- 生成基于结构的药效团模型,并将选定的药效团特征设置为optional建立部分匹配的药效团模型,最后进行了基于片段的虚拟筛选;
- 与AutoT&T联合使用,从虚拟筛选命中的片段中识别结合亲和力更好的片段,并根据化学键匹配移植到先导化合物的保留片段上获得新化合物。
总的来说,Ligandscout的虚拟筛选技术与AutoT&T的从头设计技术组合,可以非常高效的进行从头设计。
5. 接下来可以做什么
- 进一步评估化合物的构象稳定性
- 用Flare的WaterSwap评估化合物的结合亲和力
6. 相关案例
6.1 欧莱雅公司:片段虚拟筛选发现美容分子
欧莱雅公司的研究人员对已经批准上市的美容用分子进行碎片化、官能团化与重新组合等一系列处理,构建了适于虚拟筛选的虚拟库。对该虚拟库进行了基于药效团的虚拟筛选以及三个月的优化阶段后,发现了一个各种性质都理想的先导物,其有望在美容应用领域里作为皮肤护理的候选分子。本项目的成功为其它美容靶点的先导物发现铺平了道路。
7. 联系我们
采购ligandscout或AutoT&T 2,请联系我们:




