2019-nCoV 3CL水解酶与抑制剂的复合物结构
摘要:由上海科技大学饶子和/杨海涛课题组测定的2019-nCoV冠状病毒3CL水解酶(Mpro)的高分率晶体结构于2020年2月6日在PDB数据库公开,PDB代码为6LU7。本文分享了对该复合物结构分初步的分析结果,包括抑制剂与Mpro的相互作用模式,提供了药效团模型。此外,还推荐了系列共价抑制剂的设计方法与案例。
肖高铿/2020-02-06
关于2019-nCoV(即COVID-2019)
冠状病毒属于套式病毒目、冠状病毒科、冠状病毒属,是一类具有囊膜、基因组为线性单股正链的RNA病毒,是自然界广泛存在的一大类病毒。冠状病毒属包含约25种冠状病毒(CoVs),是引起高度流行疾病的重要病原体,对人和动物通常是严重或致命的。目前尚未有药物被批准专门用于防治CoV感染。细胞进入的不同宿主受体,结构蛋白(抗原)保守性差,以及冠状病毒的高突变和重组率,使得广谱抗冠状病毒药物和疫苗的开发成了重大问题。
某些冠状病毒会感染人类并引起疾病,比如中东呼吸综合征(MERS)和严重急性呼吸综合征(SARS),其症状可从普通感冒到重症肺部感染。此次在武汉首先发现的冠状病毒是一种先前尚未在人类中发现的病毒株系,被WHO命名为2019新型冠状病毒(2019-nCoV,COVID-2019),其全基因组序列可以从NCBI下载。
NCBI的病毒分类工具PASC将2019-nCov分类为严重急性呼吸综合征相关冠状病毒种(Severe acute respiratory syndrome-related coronavirus),该种还包含2003年爆发的SARS病毒,两者基因组序列相似度为80%。在已发布的数据中,2019-nCoV与2017年2月从国内的蝙蝠中采集到的Bat SARS-like coronavirus isolate bat-SL-CoVZC45基因组序列相似性最高,为88%。
国内外许多组织结构、个人为了方便大家更快的掌握2019-nCoV的生物信息学与潜在靶标信息,提供了许多有用的工具,这里列出一些:
- 2019-nCoV基因组信息
- Zhang Lab全基因组蛋白3D结构模型
- GHDDI的2019-nCoV科研数据与信息共享平台
国家生物信息中心/国家基因组科学数据中心:2019新型冠状病毒信息库。
美国Michigan大学的Zhang Lab将2019-nCoV全基因组蛋白预测3D结构模型:https://zhanglab.ccmb.med.umich.edu/C-I-TASSER/2019-nCov
清华大学全球健康药物研发中心(GHDDI)建立2019-nCoV科研数据与信息共享平台:https://ghddi-ailab.github.io/Targeting2019-nCoV/nCov_Targets
关于2019-nCoV 3CL蛋白水解酶(Mpro)
2020年1月26日中国科学报陈欢欢报道[1]:继1月25日上海科技大学免疫化学研究所和中国科学院上海药物研究所抗2019-nCoV冠状病毒感染联合应急攻关团队公布30个可能的抗2019-nCoV冠状病毒老药和中药后,1月26日,联合攻关团队及时公布由上海科技大学饶子和/杨海涛课题组测定的2019-nCoV冠状病毒3CL水解酶(Mpro)的高分率晶体结构,以便有更多的科技工作者、特别是药物研发的科技人员使用,晶体结构的坐标可到PDB蛋白质结构数据库(Protein Data Bank, PDB)下载(PDB ID: 6LU7)。饶子和院士和蒋华良院士强调,大家一起努力,研发出更多更好的抗新型肺炎药物。
Figure 1. 2019-nCoV冠状病毒3CL水解酶(Mpro)与抑制剂的复合物晶体结构(PDB 6LU7)。其中飘带状展示的部分为酶,棒状绿色C原子展示的是抑制剂结构,棒状灰色碳原子展示的是结合位点与抑制剂发生氢键相互作用的蛋白氨基酸残基。注意:抑制剂与蛋白的CYS145发生了一个共价相互作用(黄色的键)。Tip:通过鼠标中间键滚轮可以缩放模型。
同属于严重急性呼吸综合征相关冠状病毒种的SARS-CoV病毒也是一种正链病毒(Positive stranded RNA virus), 其复制酶(Replicase)对病毒内部的核苷酸(RNA)进行翻译,构成多蛋白(polyprotein),它主要进行病毒的复制与转录,但要发挥作用必须通过蛋白质的溶解作用(Proteolytic processing)变成一些具有一定生物化学功能的多肽(polypeptide),此步工作是由水解蛋白酶(Proteinase)来完成[14,15]。因此,3CL蛋白水解酶(3CL Mpro)被认为是理想的抗SARS-CoV药物筛选靶标。对应的酶也被认为是2019-nCoV潜在的靶标。
研究人员比较了四种CoV 3CL Mpro的晶体结构和一个同源建模的模型,发现它们具有高度保守的底物识别口袋。更关键的是,该口袋催化底物水解的一对关键残基Cys145、His41取向相对保守。其中His41作为质子供体,而Cys145对底物羰基碳发起亲核进攻。广泛接受的是:只要在催化活性的Cys残基和设计的抑制剂之间形成共价键,形成类似于底物裂解过程中的中间体,就可以提高抑制剂的效力。酶活性测定进一步支持了这一结论:基于这个保守的结构区域,设计了基于机理的不可逆抑制剂。SARS-CoV和猪传染性胃肠炎病毒的Mpro-抑制剂复合物晶体结构进一步阐明了该抑制机制。研究人员设计了多个抑制剂,它们对多种CoV Mpro表现出快速的体外灭活、强效的抗病毒活性以及极低的细胞毒性低。对这些化合物进一步修饰,可能发现针对现有和未来可能出现的CoV相关疾病的临床潜力的药物。
不可逆共价抑制剂N3与2019-nCoV Mpro的相互作用模式
N3化学结构如下图2所示,是研究团队前期为抗SARS-CoV而设计的Mpro不可逆共价抑制剂[2,16]。
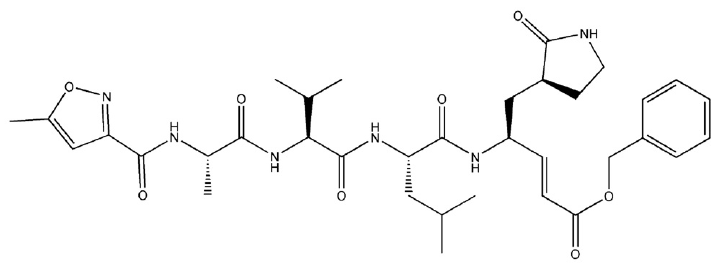
Figure 2. 抑制剂N3的化学结构[2,16]
抑制剂N3含有一个α,β-不饱和酯的Michael加成受体片段,与蛋白结合时,该片段与CYS145发生Michael加成,晶体结构(PDB 6LU7,图1)可以明确地看到这一点。图3展示了抑制剂以及与其共价相互作用的CYS145,以及抑制剂与蛋白之间的相互作用模式:其中橘黄色的球表示与CYS145共价的碳原子;绿色箭头表示氢键供体;红色箭头表示氢键受体;黄色的球表示疏水相互作用。
Figure 3. PDB 6LU7揭示的抑制剂与蛋白相互作用的药效团:其中橘黄色的球表示与CYS145共价的碳原子;绿色箭头表示氢键供体;红色箭头表示氢键受体;黄色的球表示疏水相互作用。
需要注意的是,内酰胺环的几何结构并不合理:sp2杂化的酰胺碳并不是平面型,且C=O键过长,以至于到达单键的长度。这可能会导致部分软件将该基团识别为醇而不是羰基。该酰胺sp2的O应该也是氢键受体,并可能与HIS163具有氢键相互作用。与CYS145巯基共价的碳原子的几何结构也不合理:如图3所示,并不是理想的sp3应该具有的四面体,该碳原子近乎平面结构,需要进一步确认原因。总的来说,使用或分析相互作用时,需要对这部分结构仔细地准备与处理。
PDB 2HOB晶体结构为SARS-CoV 3CL水解酶(Mpro)与N3的复合物[2]。比对2019-nCoV(6LU7)与SARS-CoV(2HOB)的Mpro蛋白序列,其PID为96.08% ,说明两者在进化上是同源的、氨基酸序列非常相似。2HOB与6LU7按Cα进行叠合的RMS=0.71Å,说明两者在3D上也非常一样。图4展示了叠合后的结合口袋部分,可以发现结合位点处的残基基本一样,但2HOB的共价碳原子与内酰胺环的几何结构更合理,因此可以参照2HOB对6LU7的配体进行修复与准备。从叠合结果也可以看到,结合口袋仅第46位残基(红框内棒状展示的残基)存在区别:6LU7为SER,而2HOB为ALA。这也暗示着SARS-CoV Mpro有效的抑制剂应该也是2019-nCoV Mpro的有效抑制剂。根据前期的研究,N3是SARS冠状病毒3CL水解酶共价不可逆抑制剂,对不同SARS-CoV Mpro的Ki大约在1.9-9μM。
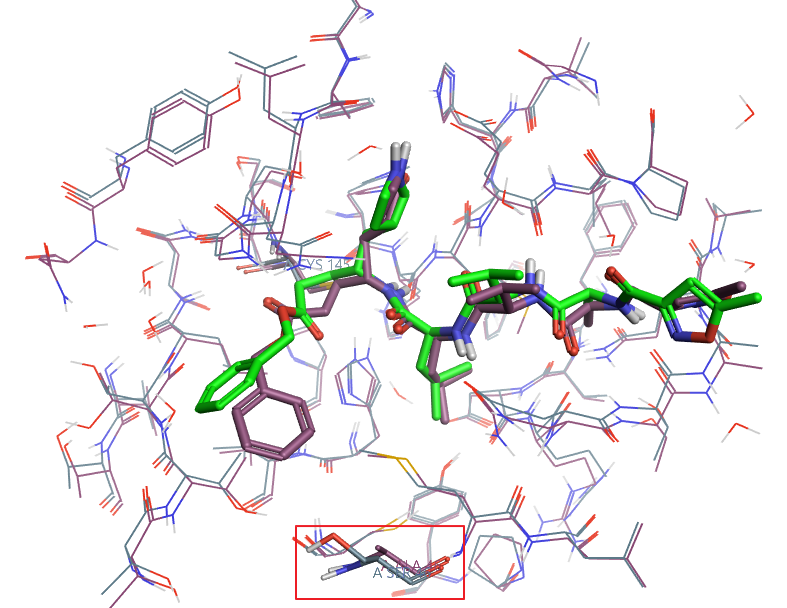

Figure 4. 上:PDB 6LU7与2HOB的叠合,仅在第46位的残基上(红框内棒状展示的残基)存在区别:6LU7为SER,而2HOB为ALA;下:2019-nCoV的M与SARS-CoV的Mpro序列比对,结合位点处仅第46位的残基存在SER与ALA的区别。
此外,Mpro结合位点相当宽且浅(图5),该口袋可能并不适合于开发常规的类药有机小分子。鉴于CYS145在多种CoV Mpro中是催化底物的关键残基、并且具有保守性,因此靶向CYS145的共价抑制剂可能是发现广谱CoV抑制剂的研究方向。N3是肽样分子,两端原子间的距离大约为21.3Å,是非常大的分子。最近文献报道的针对MERS-CoV Mpro的共价抑制剂GC376也是靶向该CYS残基,但是分子却小了很多。GC376与MERS-CoV Mpro的复合物晶体结构为PDB 5WKJ,其相互作用模式与N3-Mpro的相互作用模式有很多相同之处,值得借鉴。
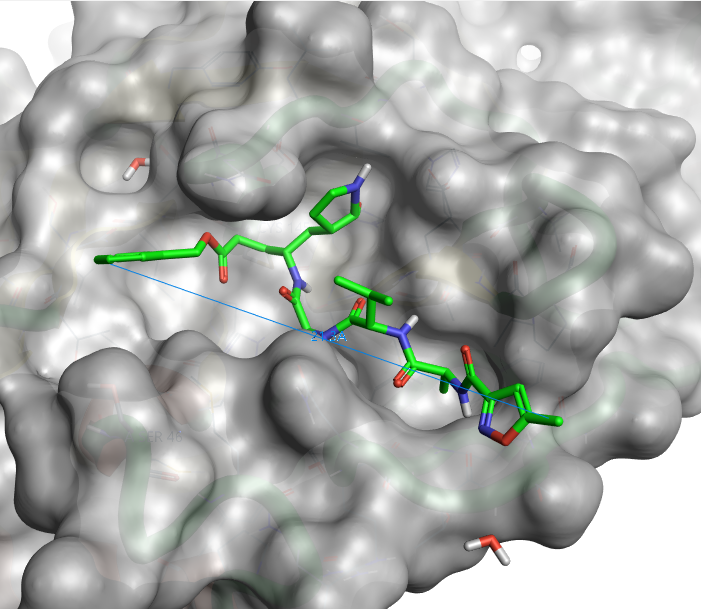
Figure 5. PDB 6LU7结合位点处的Mpro分子表面与抑制剂N3,N3两端原子间的距离有21.3Å
已知的SARS-CoV Mpro抑制剂
前面提到2019-nCoV与SARS-CoV的Mpro非常相似:蛋白氨基酸序列PID=96.08%);底物结合位点也基本一致,只有1个氨基酸残基的差异;比较N3与2019-nCoV与SARS-CoV的Mpro复合物3D结构,Cα的RMS=0.71Å,抑制剂结合模式也基本一样。因此,对SARS-CoV Mpro有效的抑制剂应该对2019-nCoV Mpro也是有效的。这里提供了BindingDB,PubChem,ChEMBL与PDB等四个数据库里靶向SARS-CoV Mpro相关化合物的检索链接,点击即可:
共价抑制剂发现相关的技术
1. 用Gaussian软件模拟设计的化合物能否与CYS发生共价结合、是否可逆共价结合
之前选择性JAK3抑制剂PF-06651600的设计[3]一文详细地介绍了辉瑞公司设计共价抑制剂的经验,强力地向大家推荐这篇文章并阅读原文[4]。共价抑制剂设计的一个重点是:如何在合成化合物前知道设计的化合物能否与CYS巯基发生共价结合,是否可逆结合呢?这个问题可以在可逆Michael受体硫醇加成反应的DFT理论计算5一文找到答案,该文详细地介绍了Houk课题组的采用Gaussian进行量化计算预测的方法6。
2. 用Gaussian软件预测设计的迈克尔受体共价抑制剂是否具有遗传毒性
共价抑制剂除了能与CYS巯基加成之外,也可能与生物大分子比如核酸的-NH2加成进而产生遗传毒性。现有的遗传毒性预测方法对Michael受体的化合物预测能力不足,但Townsend等人用DFT预测1,4-Michael受体的AMES致突变性几乎100%准确[7,8]。具体地计算方法,可以参考我们准备的AMES预测教程[9]。
3. 用Ligandscout进行共价抑制剂的虚拟筛选
Schulz R等人[10]采用基于结构的方法识别了3C蛋白酶(3C Protease)共价键结合的3D药效团模型;并用包含33个活性化合物、1771个decoy化合物的数据集对该药效团模型进行验证,发现该模型具有非常好的敏感性与专一性;对一个3000个化合物的数据库进行虚拟筛选发现3C蛋白酶共价键结合的不可逆抑制剂,最后通过骨架跃迁、结构优化发现了化学稳定的共价抑制剂。我们在Ligandscout案例 | 共价键结合药物的虚拟筛选[11]一文中对此进行了详细的介绍,可以直接借鉴过来用于Mpro共价抑制剂的虚拟筛选与设计。
为了让大家快速了解Ligandscout进行共价抑制剂虚拟筛选的步骤与流程,我们还准备了教程[12]:以EGFR Kinase-Afatinia复合物为例来演示如何用Ligandscout基于结构的方法生成带共价键特征的药效团模型、以及如何用该药效团模型进行虚拟筛选。
4. 用SPARK设计共价抑制剂
SPARK进行骨架跃迁、侧链替换、大环分子设计、水分子替换进行结合亲和力优化、分子生长与分子链接已经提供了非常多的算例。SPARK还支持药效团约束,其中包括共价药效团特征,因此也可以用来设计共价抑制剂。其中的一个教程以EGFR激酶共价抑制剂Afatinib为例,描述了如何用SPARK的共价药效团特征进行共价抑制剂弹头的结构替换(跃迁)[13]。
5. 用AutoT&T设计共价抑制剂
AutoT&T是一款基于结构的从头药物分子设计软件,用于分子生长、分子链接、基团替换与从头分子生成。在共价抑制剂的设计里,您可以将感兴趣的化合物或片段对接到蛋白结合位点里,然后AutoT&T从您对接获得的pose识别可能结合亲和力好的片段、并将之裁剪下、按反合成分析移植到另一个分子片段上去(可以是指定的片段或程序自行拼合),这样就可能得到活性更好的化合物。AutoT&T可以与各种对接软件以及Ligandscout联合使用,更多的细节,请阅读我们的教程或案例。
靶向3CL Pro的抑制剂进展
辉瑞公司于2021年4月6日在美国化学会春季会议上透露了其口服新冠抗病毒临床候选药物PF-07321332的当前开发情况,共享了化合物结构和临床前数据。该化合物当前处于I期临床试验,是临床上首个口服给药的化合物,该化合物靶向新冠病毒SARS-CoV-2的3CL Pro。通过抑制主蛋白酶,PF-07321332可防止病毒将长蛋白链切割成其自我复制所需的部分。
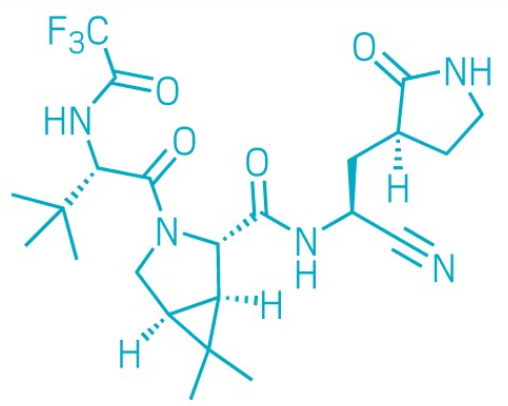
Figure 6. PF-07321332的化学结构式
辉瑞2020年曾报告了PF-07304814,这是一种不同的新冠病毒主蛋白酶小分子抑制剂,其开发工作始于 2002-2003 年非典爆发期间。但是该分子只能静脉给药,使得其使用局限于医院环境下。而PF-07321332可作为片剂或胶囊剂口服,因此如果被证明是安全有效的,则可以在医院外服用。那么经历新冠暴露的人就可以将该药作为预防措施使用。
文献
- 陈欢欢.新型肺炎病毒3CL水解酶高分辨率晶体结构公布.中国科学报. 2020-02-06 Retrieved from http://news.sciencenet.cn/htmlnews/2020/1/435186.shtm
- Xue X, Yang H, Shen W, et al. Production of Authentic SARS-CoV Mpro with Enhanced Activity: Application as a Novel Tag-cleavage Endopeptidase for Protein Overproduction. J Mol Biol. 2007;366(3):965-975. doi:10.1016/j.jmb.2006.11.073
- 选择性JAK3抑制剂PF-06651600的设计. 墨灵格的博客. http://blog.molcalx.com.cn/2019/11/10/jak3-inhibiotr-pf-06651600.html
- Thorarensen, A.; et al. Design of a Janus Kinase 3 (JAK3) Specific Inhibitor 1-((2 S ,5 R )-5-((7 H -Pyrrolo[2,3- d ]Pyrimidin-4-Yl)Amino)-2-Methylpiperidin-1-Yl)Prop-2-En-1-One (PF-06651600) Allowing for the Interrogation of JAK3 Signaling in Humans. J. Med. Chem. 2017, 60 (5), 1971–1993. https://doi.org/10.1021/acs.jmedchem.6b01694
- 可逆Michael受体硫醇加成反应的DFT理论计算. 墨灵格的博客.http://blog.molcalx.com.cn/2020/01/04/thiol-addition-to-michael-acceptor.html
- Krenske, E. H., Petter, R. C., & Houk, K. N. (2016). Kinetics and Thermodynamics of Reversible Thiol Additions to Mono- and Diactivated Michael Acceptors: Implications for the Design of Drugs That Bind Covalently to Cysteines. The Journal of Organic Chemistry, 81(23), 11726–11733. https://doi.org/10.1021/acs.joc.6b02188
- Townsend, P. A.; Grayson, M. N. Density Functional Theory Transition-State Modeling for the Prediction of Ames Mutagenicity in 1,4 Michael Acceptors. J. Chem. Inf. Model. 2019, acs.jcim.9b00966. doi:10.1021/acs.jcim.9b00966.
- 用DFT预测1,4-Michael受体的AMES致突变性. 墨灵格的博客.http://blog.molcalx.com.cn/2019/12/12/use-dft-to-predict-the-ames-mutagenicity-for-michael-acceptor.html
- Gaussian教程 | 用DFT预测Michael受体的遗传毒性(AMES test). 墨灵格的博客.http://blog.molcalx.com.cn/2020/01/08/dft-predict-ames-test.html
- Schulz, R., Atef, A., et al. (2018). Phenylthiomethyl Ketone-Based Fragments Show Selective and Irreversible Inhibition of Enteroviral 3C Proteases. Journal of Medicinal Chemistry, 61(3), 1218–1230. https://doi.org/10.1021/acs.jmedchem.7b01440
- Ligandscout案例 | 共价键结合药物的虚拟筛选.墨灵格的博客.http://blog.molcalx.com.cn/2018/05/15/ligandscout-case-study-covalent-feature.html
- Ligandscout教程 | 共价键药效团模型与虚拟筛选. 墨灵格的博客. http://blog.molcalx.com.cn/2018/05/15/ligandscout-tutorial-covalent-vs.html
- SPARK教程 | 共价结合抑制剂的骨架跃迁. 墨灵格的博客. http://blog.molcalx.com.cn/2018/07/24/spark-covalent-inhibitor.html
- Marra MA. The Genome Sequence of the SARS-Associated Coronavirus. Science (80- ). 2003;300(5624):1399-1404. doi:10.1126/science.1085953
- Anand K, Palm GJ, Mesters JR, Siddell SG, Ziebuhr J, Hilgenfeld R. Structure of coronavirus main proteinase reveals combination of a chymotrypsin fold with an extra α-helical domain. EMBO J. 2002. doi:10.1093/emboj/cdf327
- Yang H, Xie W, Xue X, et al. Design of Wide-Spectrum Inhibitors Targeting Coronavirus Main Proteases. Bjorkman P, ed. PLoS Biol. 2005;3(10):e324. doi:10.1371/journal.pbio.0030324



