摘要:为了对大型数据库进行搜索以发现潜在的活性化合物,需要对数据库进行合理的准备。本文系统论述了小分子数据库结构准备需要考虑的问题,并提供了OpenEye完整方案:FILTER,QUACPAC与OMEGA。该方案涵盖了结构准备所需的全部功能。
肖高铿/2020/09/09
化合物数据库准备的一般流程
为了对大型数据库进行搜索以发现潜在的活性化合物,需要进行合理的数据库准备工作。已经有很多文献介绍如何准备小分子数据库用于后续的虚拟筛选[1,2]。用于虚拟筛选的小分子数据建库,通常涉及到下面的准备过程:
- 标准化
- 脱盐
- 中和
- 过滤
- 去重复结构
- 互变异构体枚举
- 质子化状态枚举
- 立体化学枚举
- 3D结构生成
- 格式转化
不同来源的化合物结构,其SMILES编码、sdf、mol2文件对化合物的表示千差万别。以钠盐为例,一个常见的表达方式为钠与羧酸氧之间通过键连接,比如:
1 | [Na]OC(=O)c1ccc(C[S+2]([O-])([O-]))cc1 |
这不利于后续的脱盐处理(会被当成金属有机化合物而不是盐),需要标准化为非键连接:
1 | [Na+].O=C([O-])c1ccc(CS(=O)=O)cc1 |
酸、碱化合物通常有不同形式的盐(比如酸的钠盐、胺的盐酸盐等等),我们只需要保留酸根与碱基部分,其它的部分不参与计算而需要去除。比如上一步的分子,我们将Na+去除,只保留酸根部分:
1 | O=C([O-])c1ccc(CS(=O)=O)cc1 |
通常可选,因为接下来马上要进行“滴定”,取特定pH值下的质子化状态。但是中和之后的结构有利于下一步的去重复结构操作。
过滤掉不想要的化合物很重要,合理的过滤可以避免不必要的计算。比如过滤掉金属有机化合物,过滤掉含反应性基团的化合物,过滤掉某种子结构的化合物等等。
同一个活性成分的不同盐通常被当成不同的结构,脱盐之后就可能出现重复结构,因此有必要进行去重处理。
不同的互变异构体与蛋白发生相互作用的形式不同,分子对接结果也不同,因此包含对的互变异构体是很重要的。
根据研究靶标的结合位点特征,枚举特定pH值下的化合物质子化状态是很重要的。比如,对于含有金属的结合位点,该金属与化合物发生配位键相互作用。那么准备这样的配体的时候,不妨取其在高pH值时的质子化状态,以保证该化合物有机会与结合位点的金属发生配位相互作用。比如磺胺在高pH时N上带负电,可以与蛋白Zn发生配位键。但是质子化中性的磺胺,就不能与Zn发生配位作用了。
当SMILES或SDF文件含有双键、手性中心等立体化学因素时,有时存在立体化学没有限定清楚的问题,这时需要对立体化学不清楚的部分进行枚举以保证包含正确的结构。比如试剂公司提供的数据库化合物为消旋体,这是就需要对该消旋体进行立体化学枚举,列举其可能的异构体用于后续计算。
分子对接、药效团、分子形状等基于3D的虚拟筛选方法通常需要化合物为3D结构,高质量的3D结构至关重要。有的方法,比如vina、Dock等对接方法不对化合物的环进行构象搜索,这时需要枚举出化合物的环构象以备后续计算使用是非常重要的。
不同的软件可能需要使用不同格式化合物结构文件,通常需要进行一个格式转化。良好的格式转化能力也是不同格式数据进行整合的关键,因此选用合适的格式转化工具非常重要。
用OpenEye进行化合物数据准备
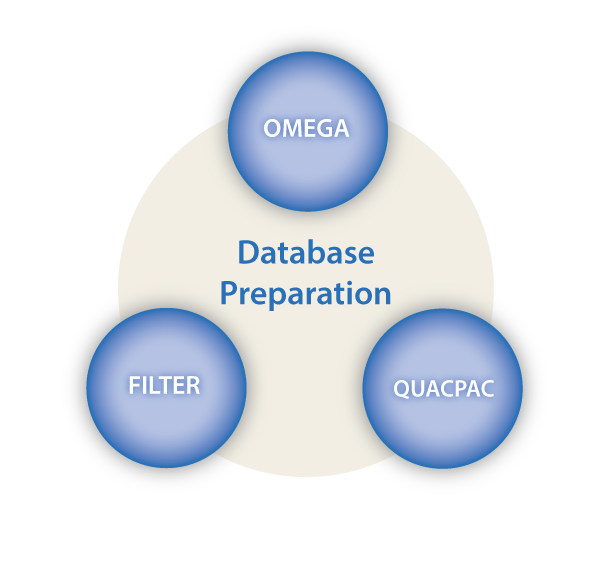
Figure 1. OpenEye提供了3个模块进行数据库的整合与准备
之前,在《云计算教程 | 在云端用VirtualFlow实现超高通量虚拟筛选》一文中也提出了一套结构准备的方案,但是很多用户反馈不同来源的软件的使用非常不方便。本文为大家介绍了OpenEye的一体化解决方案。为了准备化合物数据库,OpenEye提供了一系列工具:FILTER、QUACPAC、OMEGA几乎可以实现上述的全部功能。这三个工具相关的参数叙述如下:
FILTER:性质计算与不理想化合物的去除
FILTER与数据库准备的主要功能总结如下:
FILTER是一款非常快速的分子过滤和选择应用软件。它结合物理性质计算和官能团知识,在不需要的化合物进入实验或虚拟筛选之前除去它们。不良性质可能包括:毒性功能、与靶蛋白共价结合的高可能性、干扰实验测定和/或口服生物利用度低的可能性。在使用建模应用程序之前消除不需要的分子,反过来,将大大提高这些工具的积极预测值,并显著减少其处理时间。
1. 过滤
FILTER可以在进行耗时的任务前过滤掉理化性质不理想或图性质不理想的化合物。FILTER包含了内置过滤器与自定义文件过滤器。内置过滤器为预先准备好的过滤器,用来过滤出类药、类先导分子等等。用户还可以用文件按照自己的目的自定义过滤器。FILTER的内置过滤器包括:
- Blockbuster:推荐的默认过滤器
- Lead:类先导化合物过滤,用于准备HTS数据库
- Drug:类药化合物过滤,用于后期阶段的药物开发,很多时候这个时候这个过滤器太严格
- Macrocycle:类药化合物过滤,去除非大环分子
- Frag:用于从数据库中提取具有连接点的小分子
- Pains:过滤掉常见含有干扰生物活性测试子结构的化合物
过滤器用-filter选项来实现,比如内置过滤器Pains过滤:
1 | -filter Pains |
-filter的选项还可以是一个文件,该文件含有4种类型的声明:
- physical property limits
- rules
- New rules
- Selections
在过滤文件种,每行一种类型的声明。通过过滤文件,可以实现自定义的各种化合物过滤规则。下面列举少数几种来说明过滤文件的强大功能与使用风格(不是全部功能的演示),全部的过滤特性,请参见说明书:filter file
1.1 物理性质限制
FILTER支持各种各样的物理性质约束,以1行3列的方式出现,比如:
1 | MIN_HETEROATOMS 2 "Minimum number of heteroatoms" |
第1列是性质关键词,第2列为该关键词的值,第3列为该关键次的简要说明。在FILTER里包含了非常多的物理性质关键词,用户不能自己添加关键词。目前支持的关键词见说明书:filter file
1.2 原素组成过滤
FILTER支持元素组成过滤,比如ALLOWED_ELEMENTS关键词定义了什么样的元素是允许的,把含有该列表元素之外的元素的化合物去除掉。
1 | ALLOWED_ELEMENTS H,C,N,O,F,P,S,Cl,Br,I |
1.3 官能团过滤
比如限制某个基团出现的次数等。例如,去除酰卤,可以限制酰卤出现次数为0:
1 | RULE 0 acid_halide |
1.4 理化性质过滤
可以过滤多种理化性质,比如溶解度,油水分配系数,PSA等过滤。
2. 脱盐
FILTER的-salt选项可以用来脱盐。-salt后面的参数为一个文件。该文件里包含了一系列你认为是盐的化合物。此时,如果数据库的一个化合物由多个不连续片段组成,那么任何出现在参数文件中的片段就被认为是盐而被去除。如果-salt选项没有试用文件参数,那么由多个不连续片段组成的分子仅保留其第一个最大的片段,其它的片段被认为是盐而去除掉。-salt的默认选项为空,即保留最大片段的方式脱盐。
3. 固定pH值的质子化状态处理
FILTER的pkanormal是boolean型选项:如果为true,则将化合物设定为pH=7.4时的质子化状态。默认该选项为true,使用方法:
1 | -pkanormal true |
3. 去重复结构
FILTER的-unique选项可用来去除重复结构。
QUACPAC:质子化状态、互变异构体与电荷的计算
分子相互作用的化学本质是分子的形状与静电问题,糟糕的静电处理比不做任何处理还糟糕;精确的电荷是非常重要的。如果分子的质子化状态是错误的,精确的电荷计算模型也没有用。QUACPAC试图提供正确计算电荷的全套必要功能,包括pKa预测与互变异构体枚举以便获得正确的质子化状态;兼顾速度与精度的多种电荷计算模型;静电势图的构建与存储。
QUACPAC包含下面几个子程序可以实现不同的结构准备功能。
- FixpKa
- MolCharge
- pKaTyper
- Tautomers
用基于规则的系统设置输入分子的离子化状态。
为小分子、生物大分子计算合适的原子偏电荷
枚举化合物的质子化状态与评估化合物的pKa
枚举化合物的互变异构体。
OMEGA: 生成高质量的3D构象
OMEGA主要用于生成小分子3D构象以及包含生物活性构象的构象系综。OMEGA专门为大规模化合物数据库的构象搜索、分析而设计,它可以快速、可靠地产生多构象数据库。OMEGA生成的构象数据库可用于分子对接虚拟筛选(比如FRED)、形状比较虚拟筛选(比如ROCS)、以及药效团识别与虚拟筛选(比如Ligandscout)。
OMEGA除了用于生产化合物构象之外,还包含了FLIPPER模块。FLIPPER模块用于枚举化合物立体化学异构体以便进一步用于OMEGA生成3D结构。OMEGA里面也有flipper选项用来实现相关的功能,下列举几个与立体化学相关的选项。
3.1 立体化学枚举
-flipper true选项让OMEGA在生成3D构象前枚举立体化学不明的立体化学中心的立体异构体;
-flipper_maxcenters选项设置需要让OMEGA处理的立体化学中心的最大数量;
3.2 非平面N的翻转枚举
-enumNitrogen true选项将激活OMEGA对非平面N进行翻转枚举构象。一般分子对接软件不会翻转氮原子的构象,因此预先枚举N的构象是非常重要的。
3.3 环构象的枚举
-enumRing true选项将激活OMEGA对环的构象进行枚举,以确保输入分子的环包含了正确的构象。一般分子对接软件把环系视为刚性,在计算结合模式预测过程中不会改变环的构象,因此预先枚举环的构象是非常重要的。
3.4 大环分子的构象搜索
OMEGA支持对大环分子的构象搜索。
小结
总的来说,OpenEye的OMEGA,QUACPAC与FILTER涵盖了小分子化合物数据库准备所需的各种功能,为您的虚拟筛选提供高质量的起点。
文献
- Irwin, J. J.; Shoichet, B. K. ZINC – A Free Database of Commercially Available Compounds for Virtual Screening. J. Chem. Inf. Model. 2005, 45 (1), 177–182. https://doi.org/10.1021/ci049714+.
- Gally, J.-M.; Bourg, S.; Do, Q.-T.; Aci-Sèche, S.; Bonnet, P. VSPrep: A General KNIME Workflow for the Preparation of Molecules for Virtual Screening. Mol. Inform. 2017, 36 (10), 1700023. https://doi.org/10.1002/minf.201700023.



