摘要:本文以Merck FEP计算基准数据集TNKS2算例中的两个化合物7与5e为例,演示了Flare FEP在速石云计算平台上采用1张Tesla T4卡进行FEP计算的性能表现。其中TNKS2靶标全长210个氨基酸残基,FEP计算的每个转化方向使用了9个λ窗口(双向共18个9个λ窗口),每个λ窗口进行了4ns的模拟。完成整个FEP计算总共花了10.2小时,平均每个λ大约35分钟,这意味着如果用18个GPU并行加速计算有望在35分钟左右完成计算,实现总计算费用不变的情况下以最快的速度完成计算任务。该性能表现与我们之前在AWS T4 GPU计算卡上测试的性能基本一致,这进一步证明了本次性能测试的可靠性。因此,使用速石云平台搭载T4显卡的p1.c4t4.1机型以及核心的产品功能是非常值得推荐用于FEP计算的。
作者:肖高铿a,刘道龙b
日期:2022-11-11
a.广州市墨灵格信息科技有限公司
b.上海速石信息科技有限公司
前言
Flare FEP是一个高度集成且用户界面友好的自由能微扰(Free Energy Perturbation,FEP)计算工具,它由Cresset和英国爱丁堡大学的Julien Michel博士合作开发而来1。它集成了诸如AMBER tools,OpenMM,LOMAP,Sire和BioSimSpace等等最棒的开源工具、还进行了软件扩展和版本改进,与Cresset专业知识相结合集成在药物设计软件Flare里为用户提供了直观的使用体验。总的来说,Flare FEP健壮、用户友好、经过充分验证2,使用“炼金术”转化来预测同系物的相对结合亲和力变化。
FEP计算有两个步骤非常耗费计算资源:一个是小分子的力场参数化,另一个是炼金术法过程中每个λ窗口的模拟。
传统上,小分子的力场参数化是通过自定义扭转势来实现,通常用量子力学 (QM) 对两面角进行旋转扫描生成3,但这会带来巨大的计算成本,具体取决于分子的柔性(可旋转键的数量)。然而,我们利用了ANI-2X4,一种机器学习的QM近似,经过专门改进以更好地预测扭转曲线。有了这个,我们以很小的计算成本获得密度泛函理论 (ωB97X/6-31G(d)) 水平的准确性。这种深度学习加速FEP计算的特性已经在Flare V6.01开始可以使用5。
FEP计算过程需要大量的GPU资源,多个GPU并行计算加速该过程的原理是:在每对分子转化时的每个λ窗口都是独立的计算,因此可以用多个GPU分别同时进行。具体可以用多少张GPU卡加速取决于有多少对分子(linker)以及每对分子用了多少个λ窗口。比如,在之前的一个FEP计算演示里,就用上148张T4 GPU进行计算6。然而GPU硬件并不便宜,不少FEP用户并没有足够的GPU资源去完成计算。得益于云计算供应商的发展,目前GPU资源可以方便从供应商那里以按量付费或包月、年付费的方式获得。根据之前我们的测试,Flare FEP对典型的算例(蛋白大约300-400氨基酸长度,每个λ窗口进行4ns的模拟,每对分子双向转化含18个λ窗口)在AWS云上常见的GPU计算资源测试的性能表现如表1所示。
表1. Flare FEP在AWS常见GPU计算资源上的性能表现
| System | Time (mins) for 1 λ window | Time (hrs) per-2-way 9 λ calc |
|---|---|---|
| g4dn.xlarge(Tesla T4) | 37.4 | 11.2 |
| p3.2xlarge (Tesla V100-SXM2) | 23.4 | 7.0 |
| g5.xlarge (A10G OpenCL) | 19.7 | 5.92 |
典型的FEP计算,蛋白靶标通常含有300-400个氨基酸残基,每对分子一个方向转化用9个λ窗口,双向转化则为18个λ窗口;每个λ窗口进行4ns时长的模拟。如表1所示,每个λ窗口不同的计算资源所需的机时不同,较新的A10G显卡计算速度比T4快了一倍左右。但是,搞药化的朋友都精打细算,他们会发现:如果我用18张显卡进行并行计算,T4显卡需要37分钟,A10G显卡需要19.7分钟,A10G所需的墙时间(挂钟时间)在体验上仅比T4显卡快了17分钟左右,这个时间差对项目进度的影响完全可以忽略不计,但是A10G显卡的机时费可远远地比T4贵得多!也就是说T4卡比A10G具有更高的性价比。
鉴于T4显卡良好的性价比,本文的主要目的是测试Flare FEP在速石科技的T4显卡上的性能表现,为大家在选择计算资源上提供可靠的参考。
测试材料与方法
测试方法
本次计算的数据集来自德国默克公司Christina Schindler与Daniel Kuhn等人的TNKS2算例7。我们并不对所有可能的分子对都进行计算,我们仅选择其中的7与5e这对分子为算例进行测试,结构式与实验结合亲和力ΔG见图1。
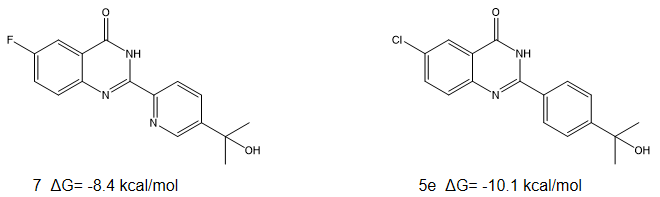
图1. 7与5e的结构结构式与实验结合亲和力ΔG(kcal/mol)
在Flare V6.01里下载PDB 5NVF,然后用Protein Prep进行结构准备,准备过程按默认的方式进行,并将配体从结合位点里提取出来,为了后期处理方便将Chain A Other里除了Zn离子之外的其它分子全部删除。然后将Merck基准数据集7TSNK2算例的蛋白与配体导入到Flare里,并用Flare | Sequences比对两个蛋白、将准备好的5NVF蛋白叠合到基准数据集的蛋白结构上。这么做的目的是让基准数据集里已经准备好的配体7与5e以正确结合模式出现在5NVF的结合口袋里(图2)。
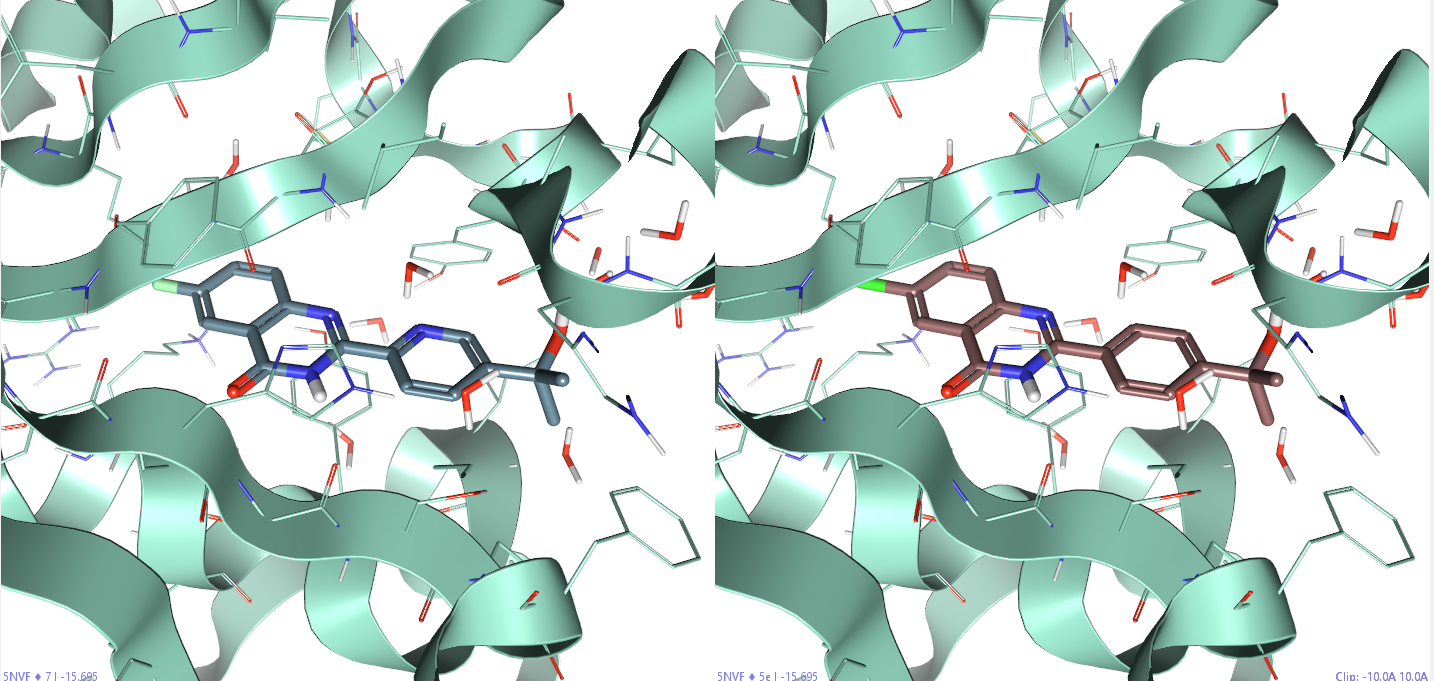
图2. 7与5e与PDB 5NVF结合口袋的3D结构
通过与5NVF共晶配体(未呈现)比较发现,7与5e的各原子与之在叠合非常一致,7与5e的各个原子也叠合地几乎完全一致。这确保了7与5e满足了FEP计算一个前提假设:用于计算的配体具有相似的结合模式。进一步用Check protein对蛋白结构进行检查,发现没有什么错误。用Home | Contacts检查蛋白与配体的相互作用,也与我们预先对该体系的理解一致。蛋白的配体的质子化状态、互变异构体等都符合我们的预期。此外,根据蛋白的序列可以知道A、H两个链总共包含了210个氨基酸残基。
现在可以开始FEP计算了,在Ligand表单选中7与5e,然后点击Flare | 3D pose| FEP | New FEP Project创建FEP计算项目,参数如下:
- Ligands: Selected 2 Ligands
- Activity column: exp dg(ΔG)
- Protein: 5NVF_P
- Chains: A Chain, A Other, A Water, H Chain, H Water
点击Create按钮,弹出FEP Generate Graph对话框,设置如下参数:
- Select the type of graph: Generate new normal graph
- Mode: Benchmark
其它参数均采用默认值,然后点击Generate Graph会弹出FEP计算界面。
在进行FEP计算之前先检查7与5e互相转化的原子映射关系是否对,先用鼠标单击连接在7与5e分子方框之间的双向箭头,再点击菜单栏上的Atom Mapping,检查两分子的原子对映关系(图3)。
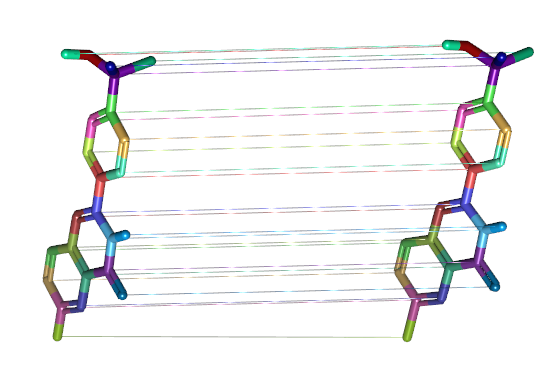
图3. 7与5e的Atom mapping检查
确认Atom mapping无误之后,点击Run FEP,弹出FEP Calculation菜单(图4)。
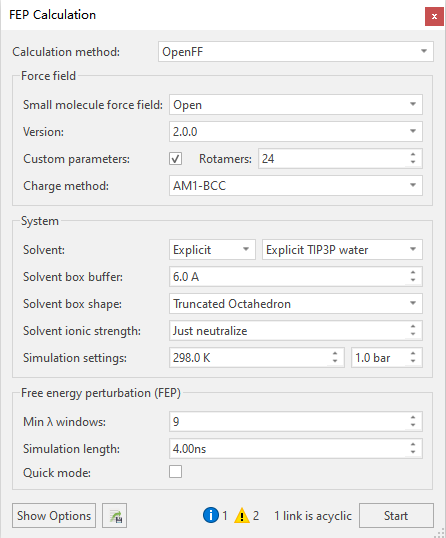
图4. FEP计算的参数设定
FEP计算参数采用默认值,如图4所示。其中自定义力场参数(Custom parameters)选项已经打钩且Rotamers设置为24,这意味着每个分子将分割成片段,每个片段柔性键进行24次旋转,然后用深度学习势能ANI-X2进行结构优化、计算势能,并进行力场拟合。这本次计算之前,我已经对这些分子进行过自定义力场拟合,新的力场参数被缓存起来待用。因此,本次FEP计算将会直接利用缓存里的力场参数而不重新计算。
Flare FEP会根据分子对之间的Link Score来调整FEP计算的关键参数λ窗口数。如图4所示,在本次计算中,λ窗口数为9,每个λ窗口的模拟时长为4ns。这正是大多数情况适用的参数。这本算例中进行了7到5e与5e到7e的双向计算,因此总λ窗口数为18。FEP计算所耗计算资源与Λ窗口数以及每Lambda窗口的模拟时长成正比。
鉴于力场参数的拟合是事先预计算过的,因此本次计算的主要机时消耗是FEP炼金术步骤。
计算环境
本次计算采用速石科技的云计算资源,登录节点与计算节点的关键硬件参数如下表2所示。
表2. FEP计算的硬件环境
| 机型名称 | 用途 | CPU与内存配置 | GPU配置 | 网络性能 |
|---|---|---|---|---|
| p1.c1.8 | 登录节点 | 8核心/16GB | 无 | 10Gb |
| p1.c4t4.1 | 计算节点 | 4核心(Lake)/16GB | 1张Tesla T4 | 25Gb |
本次计算采用的操作系统为Centos 7.9,显卡驱动为510.60.02,GUP计算采用CUDA平台(version 11.6),队列管理系统为SLURM。本次使用速石云计算平台的功能包括:速石云自动化技术、调度系统、灵活性配置、数据管理与审批、易用性接入、高性能存储等功能,实现一键构建集群、一键伸缩集群、一键销毁集群,并且很好的管理和维护、监控整个集群的生命周期等,与FEP高效对接,快速帮助FEP完成计算,输出结果。
结果
计算完毕,得到图5所示的计算结果。连接化合物7与5e方框的箭头为绿色,意味着7与e5相互转化的FEP计算得以成功完成。
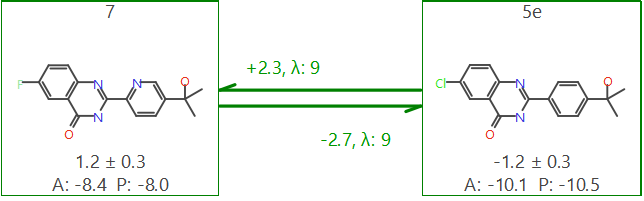
图5. 7与5e的FEP结合亲和力预测结果
箭头上下的数值为对应FEP计算转化的结合亲和力(ΔG)的差值(ΔΔG)。方框里面的A为ΔG实验值,P为ΔG预测值。其中预测值P是根据徐华锋8提出的数学方法计算而来。可以发现,计算值与实验值匹配地相当不错。
本次计算限制在1个计算节点,检查日志可以发现,从开始FEP计算到结束总共花了10.5小时,比之前报道11.2小时略快,这可能与本次计算的蛋白序列长度较短(210个AA)有关。虽然在计算中也包含了拓扑网络的生成、力场的参数化以及500ps的分子动力学模拟平衡体系等计算,但是这些计算在本次实验中所用机时非常少,可以认为10.5小时全部都花在FEP计算本身上,则平均每个λ窗口的计算时间为35分钟,总结见表3。这意味着,如果云上的GPU卡足够的话,不管对多少化合物进行FEP计算,有望在35分钟的挂钟时间内完成计算。
表3. 在TNKS2抑制剂算例中,Flare FEP在TESLA T4 GPU设备上的计算速度
| System | Time (mins) for 1 λ window | Time (hrs) per-2-way 9 λ calc |
|---|---|---|
| TESLA T4 | 35 | 10.5 |
结论
本文以Merck FEP计算基准数据集中TNKS2算例中的两个化合物7与5e为例,演示了Flare FEP在速石云计算平台上采用1张Tesla T4卡进行FEP计算的性能表现。其中TNKS2靶标全长210个氨基酸残基,FEP计算的每个转化方向使用了9个λ窗口(双向共18个9个λ窗口),每个λ窗口进行了4ns的模拟。完成整个FEP计算总共花了10.2小时,平均每个λ大约35分钟。这意味着如果用多个GPU并行加速计算,无论需要的分子数有多少,都有望在35分钟挂钟时间内完成计算,实现总计算费用不变的情况下以最快的速度完成计算任务。该性能表现与我们之前在AWS T4 GPU计算卡上测试的性能基本一致,这进一步证明了本次性能测试的可靠性。因此使用速石云平台搭载T4显卡的p1.c4t4.1机型以及核心的产品功能是非常值得推荐用于FEP计算的。
关于速石科技
速石科技(fastone)致力于构建为应用定义的云,让任何应用程序,始终以自动化、更优化和可扩展的方式,在任何基础架构上运行。
速石科技(fastone)为创新驱动型用户提供为应用优化的一站式研发云平台,满足半导体、新药研发、汽车/智能制造、人工智能、金融科技等企业及高校科研机构多种研发场景需求。基于本地+公有混合云环境的灵活部署及交付,帮助用户提升20倍研发效率,降低成本达到75%以上,加快市场响应速度,面向全球开展竞争。
速石科技(fastone)提供即开即用的一整套研发环境,以BIO/EDA/CAE/AI/Fintech等行业应用的分析与加速为核心,小时级交付用户能快速上手的可视化平台,海量云端异构资源(CPU/GPU/TPU/FPGA)智能化调度,全球多区域一体化协同管理和专业的R&D-IT服务,实现以业务为导向的IT自动化,助力研发驱动行业创新。
文献
- Flare FEP. https://www.cresset-group.com/software/desktop/flare/flare-fep
- Kuhn, M.; Firth-Clark, S.; Tosco, P.; Mey, A. S. J. S.; Mackey, M.; Michel, J. Assessment of Binding Affinity via Alchemical Free-Energy Calculations. J. Chem. Inf. Model. 2020, 60 (6), 3120–3130. https://doi.org/10.1021/acs.jcim.0c00165.
- 自定义力场参数. 墨灵格的博客. http://blog.molcalx.com.cn/2020/12/17/custom-force-field.html
- Devereux, Christian, et al. “Extending the applicability of the ANI deep learning molecular potential to sulfur and halogens.” Journal of Chemical Theory and Computation 16.7 (2020): 4192-4202
- 自定义力场进行分子动力学模拟与FEP计算. 墨灵格的博客. http://blog.molcalx.com.cn/2022/09/15/ff-md-and-fep.html
- 用FLARE FEP计算相对结合自由能. 墨灵格的博客. http://blog.molcalx.com.cn/2020/07/17/flare-fep.html
- Christina Schindler and Daniel Kuhn. Benchmark set for relative free energy calculations. https://github.com/MCompChem/fep-benchmark. DOI:10.5281/zenodo.3360435
- Xu, H. Optimal Measurement Network of Pairwise Differences. J. Chem. Inf. Model. 2019, 59 (11), 4720–4728. https://doi.org/10.1021/acs.jcim.9b00528.
联系我们,亲自测试FEP
下载算例项目文件,搭建测试环境,请联系我们。



