锁定生物活性构象增强活性
摘要:本文以HNE抑制剂为例来演示如何用Flare QM的Torsion Profile来评估化合物构象能偏好,复现了从化合物22到27的锁定生物活性构象、活性增强195倍的设计。Flare QM的Torsion profiling可以方便地进行化合物扭转角分析,进行锁定生物学构象的分子设计。
肖高铿/2024-02-15
前言
人类中性粒细胞弹性蛋白酶 (Human neutrophil elastase,HNE) 是基质降解的关键蛋白酶,在炎症性疾病中可观察到过高的HNE活性。因此,HNE是治疗慢性阻塞性肺病 (COPD)、急性肺损伤 (ALI)、急性呼吸窘迫综合征 (ARDS)、支气管扩张 (BE) 和肺动脉高压 (PH) 等肺部疾病的潜在靶点,HNE抑制剂有望重建蛋白酶-抗蛋白酶的平衡。
Vonnussbaum等人1报道了一种HNE的二氢嘧啶酮先导化合物,具有良好药代动力学性质的口服活性化合物,例如候选药物BAY-85-8501(化合物29,图1)在ALI啮齿动物模型上证明有效,并进入临床研究阶测试治疗肺部疾病的有效性。BAY-85-8501在保持出色的靶标选择性的同时,通过关键位置的甲基砜取代基将这些抑制剂的生物活性构象锁定来实现pM水平活性。
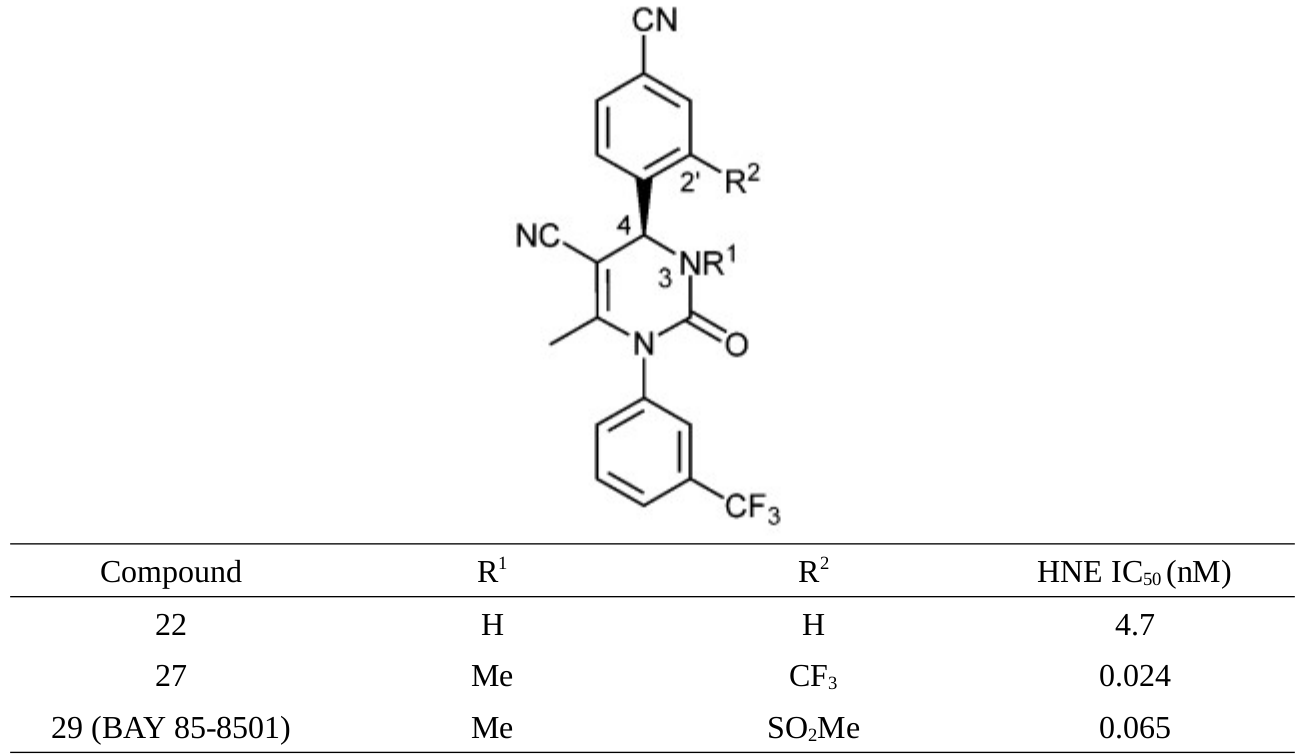
图1. 二氢嘧啶酮类HNE抑制剂22、27与291
本文地目的是以Vonnussbaum等人1报道的化合物22与27(图1)为例来演示如何用Flare QM3,4的Torsion Profile来评估化合物构象能偏好,以复现从化合物22到27锁定生物活性构象、活性增强195倍的设计。
结果与讨论
配体的生物活性构象
1,4-二氢嘧啶酮类化合物19与HNE共晶结构PDB 5A09揭示的结合模式如图2所示,化合物19折叠成钳子状,间-三氟甲基苯基与对-腈基苯基两个钳脚以垂直于1,4-二氢嘧啶酮母核的方式嵌入到S1与S2结合子口袋,19与HNE的结合由形状互补驱动。母核的酰胺羰基氧与Val216的酰胺-NH发生了强氢键相互作用;间-三氟甲基苯基完全占据了疏水S1子口袋,苯环与Phe192、Val216侧链发生疏水接触;对-腈基苯基嵌入到子S2口袋,苯环与Phe215、Leu99侧链发生疏水接触。

图2. 二氢嘧啶酮类HNE抑制剂19与HNE共晶结构PDB 5A09的结合模式
apo-HNE的结合位点比较浅,将apo-HNE(PDB 1HNE)叠合到holo-HNE(PDB 5A09)的共晶结构上,比较apo-HNE的分子表面与化合物19,如图3所示,可以发现间-三氟甲基苯基侧链嵌入到apo结构的S1子口袋;而对-腈基苯基侧链所在S2子口袋为闭合状态。因此可以推断S2子口袋是通过化合物19与HNE相互作用的诱导契合而产生的。
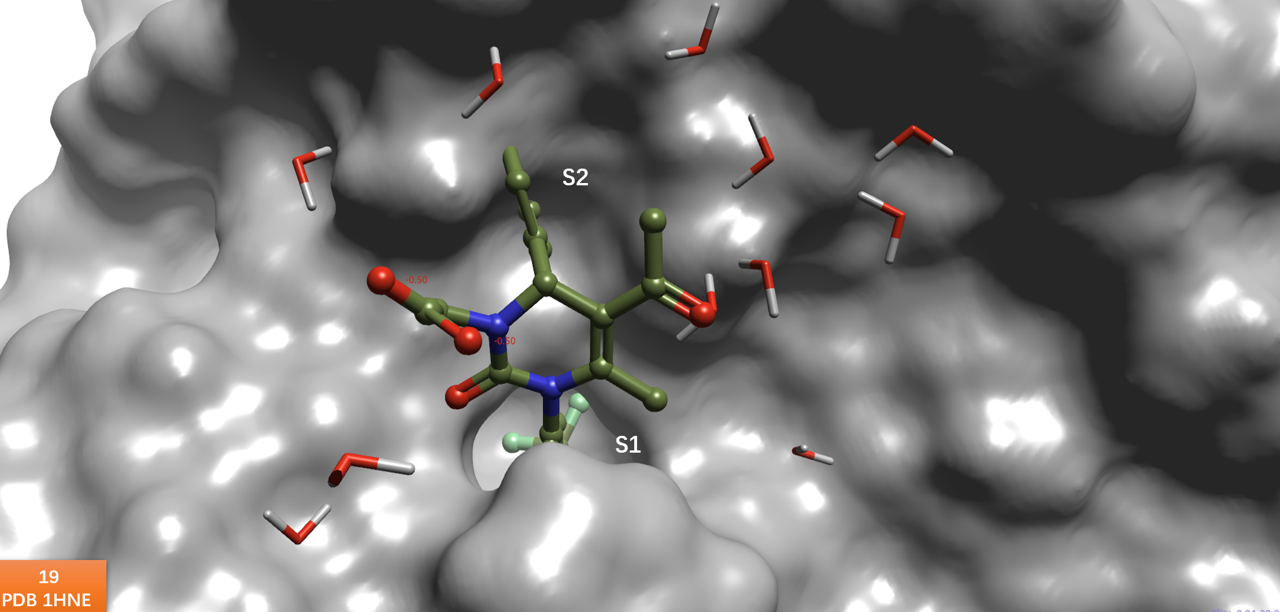
图3. 二氢嘧啶酮类HNE抑制剂19与HNE apo结构(PDB 1HNE)的分子表面比较
Guido Hansen等人2通过比较apo与holo-HNE结构发现Asp95-Leu99的重排而产生深的、典型结合口袋。如图4-右所示,将holo-HNE(PDB 5A09)叠合到apo-HNE(PDB 1HNE)之后,可以看到PDB 1HNE的Leu99侧链出现在19的对-腈基苯基处,导致S2口袋关闭而消失。当与化合物19结合时(PDB 5A09),如图4-左所示,S2附近的水被排除,Leu99侧链移动而暴露出S2口袋,19的对-腈基苯基占据S2子口袋。
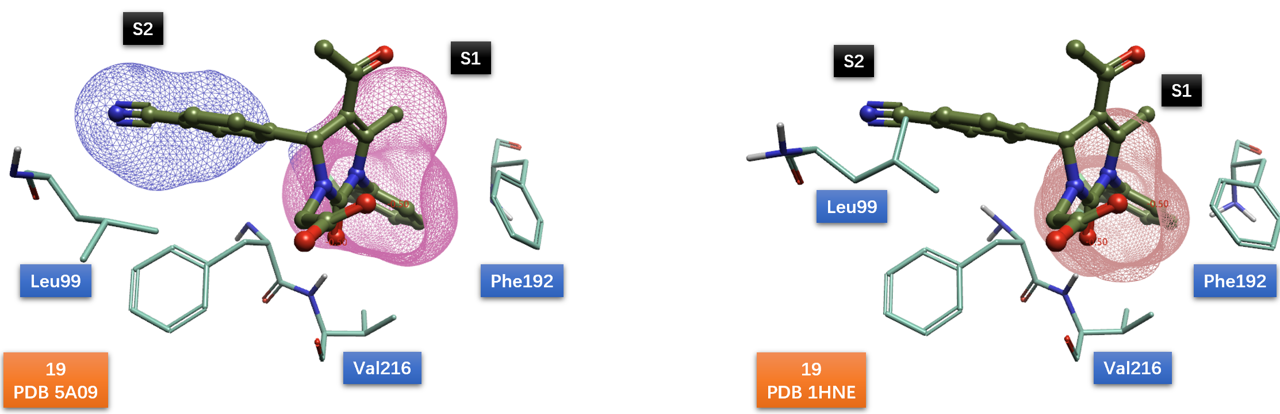
图4. apo-HNE(PDB 1HNE)与holo-HNE(PDB 5A09)的S1与S2子口袋
在二氢嘧啶酮全系列已解释的共晶结构中均可看到这个诱导契合,比如在化合物17(PDB 3Q77)、19(PDB 5A09)、20(PDB 5A0A)与24(PDB 5A0B)的共晶结构中,腈基取代的苯环或吡啶以几乎相同的取向嵌入到S2子口袋,如图5所示。
图5. 二氢嘧啶酮类HNE抑制剂17、19、20、24与HNE的共晶结构
比较化合物17、19、20与24的生物活性构象,如图6(a)所示,结合到S1子口袋的间-三氟甲基与母核1,4-二氢嘧啶酮的取向基本一致;结合到S2子口的对-腈基苯基/吡啶基与1,4-二氢嘧啶酮连接轴的两面角有所变化,大约分布在90-135°,如图6(a与b)所示。

图6. 二氢嘧啶酮类HNE抑制剂17、19、20与24的生物活性构象1
以化合物19为例,如图6(c)所示,根据共晶配体(PDB 5A09)测得的N3-C4-C1’-C2’两面角=106°,接近4个化合物的两面角均值为110°,并作为理想生物活性构象两面角。
化合物22与27的QM扭转角分析
对化合物22、27的两面角N3-C4-C1‘-C2’(编号见图6化合物19的示例)进行了基于QM的扭转角分析(见材料与方法部分),计算在B3LYP-D3BJ/6-311+G(d,p)理论水平下进行,步长为5°。计算时将结合于S1口袋的间-三氟甲基苯基删除,用氢替换。
虽然Flare QM/Torsion Profile默认的扫面范围是0-360°,但是我们关心的活性构象范围在90-135°之间且主要考察对-腈基芳基是否得到限制,出于方便、公平的比较两个化合物,仅对0-180°的两面角区间进行分析,结果如图7所示。

图7. 基于QM的扭转角分析结果。绿色区域:生物活性构象范围90-135°,理想值为~110°。红色分子:截短的22;蓝色:截短的27;高亮原子为进行扭转角分析的两面角。
一方面,化合物22最低能构象的两面角N3-C4-C1‘-C2’角度为155°,能量优势的两面角不落在活性构象范围内并与理想值110°相差45°。另一方面,当两面角为45°时能量最高,形成的能垒(ΔE)为2.456kCal/mol,这与Vonnussbaum等人1报道的ΔE~10kJ/mol基本一致,这个能垒在室温下是相当容易跨越的,也就是说化合物22的两面角N3-C4-C1‘-C2’可以轻易地发生旋转。
与化合物22相比,化合物27有两个优势:(1)27的最低能构象两面角N3-C4-C1‘-C2’角度为135°,虽然与Vonnussbaum等人1报道的120°不同,但是落在活性构象范围之内,依然支持22的生物活性构象是一个能量优势构象。(2)当两面角为45-50°时构象能最高,形成的能垒ΔE=9.849kCal/mol,这与Vonnussbaum等人1报道的ΔE~40kJ/mol基本一致,比之化合物22,跨越这个能垒的难度增加,起到了锁定生物学构象的作用。
结论
总地来说,基于QM的扭转角分析表明,在1,4-二氢嘧啶酮酰胺N(3位)引入甲基以及在与S2子口袋结合的对-腈基苯基的邻位(1‘位)引入三氟甲基,一方面使得生物活性构象转化为低能的优势构象,另一方面使得旋转能垒提高而锁定生物活性构象。这两个因素一起使得化合物的体外HNE抑制活性提高了195倍,半数抑制浓度(IC50)从22的4.7 nM降低到27的0.024 nM。Flare QM的Torsion profiling可以方便地进行化合物扭转角分析,进行锁定生物学构象的分子设计。
材料与方法
蛋白结构的准备
在化合物29优化地过程中,解释了同系物19与HNE的共晶结构PDB 5A09(图8)。将共晶结构PDB 5A09下载到Flare3中,并用Protein Prep工具进行结构准备以添加氢原子、优化氢键、消除原子冲突并给蛋白结构与配体分配最佳质子化状态,并对截短的蛋白质链继进行封端。

图8. 化合物19(左)与HNE的共晶结构PDB 5A09以及在结合位点里对19编辑得到的22(中)与27(右)。
配体结构的准备
从上一步准备好的复合物结构里将化合物19提取出来,并在结合位点里对19的副本进行编辑得到化合物22与27的初始结合构象,然后用XED力场进行优化得到22与27的结合构象,如图8所示。
基于QM的扭转角分析(Torsion Profiling)
用Flare QM对化合物22与27的两面角N2-C1-C7-C8(图9)在B3LYP-D3BJ/6-311+G(d,p)理论水平进行Torsion Profile分析。

图9. 化合物22(左)与27(右)进行Torsion Profile分析的两面角N2-C1-C7-C8。
QM Torsion Profile是CPU计算量很大的任务,为了加速计算,将22与27的二氢嘧啶酮酰胺氮上的3-三氟甲基苯基删除之后用氢饱和氮上的共价键,并设置如图10的计算参数。

图10. 化合物22与27的QM Torsion Profile参数
在Torsion Profile的参数中(图10),Number of Rotatomer=72,意味着两面角N2-C1-C7-C8在0-360°范围内划分72个构象,也就是每隔5°一个构象进行两面角约束的能量最小化计算。
文献
- Vonnussbaum, F.; Li, V. M. J.; Allerheiligen, S.; Anlauf, S.; Bärfacker, L.; Bechem, M.; Delbeck, M.; Fitzgerald, M. F.; Gerisch, M.; Gielen-Haertwig, H.; et al. Freezing the Bioactive Conformation to Boost Potency: The Identification of BAY85-8501, a Selective and Potent Inhibitor of Human Neutrophil Elastase for Pulmonary Diseases. ChemMedChem 2015, 10 (7), 1163–1173. https://doi.org/10.1002/CMDC.201500131.
- Guido Hansen, Heike Gielen-Haertwig, Peter Reinemer, Dietmar Schomburg, Axel Harrenga, Karsten Niefind,Unexpected Active-Site Flexibility in the Structure of Human Neutrophil Elastase in Complex with a New Dihydropyrimidone Inhibitor. Journal of Molecular Biology,2011,409(5):681-691. https://doi.org/10.1016/j.jmb.2011.04.047.
- Flare V8. https://www.cresset-group.com/software/flare
- Flare QM. https://www.cresset-group.com/software/flare-qm
联系我们,获取软件试用
想要亲自尝试,您可申请Flare试用版。



