摘要:本文预告了Flare V3的部分新功能与性能改进。自由能微扰(FEP)计算相对结合自由能是该版本的最大亮点,已经用多种数据完成了内部验证。与Schrodinger的FEP+相比性能相当,而优于AMBER/TI;此外,模板对接(Template Docking)改善了同系物分子对接的性能。
最新的Flare版本V3即将发布,这里预告一下部分新功能。
GPU加速的自由能微扰(FEP)
自由能微扰(Free Energy Perturbation,FEP)计算可用来预测同系物的相对结合亲和力变化。 在FEP计算中,分子通过非物理(“炼金术”)途径逐渐转变为结构上密切相关的类似物。通过对这些互相转化的终态之间的自由能差(ΔΔG)的计算评估,可以获得与实验值相比约1kcal/mol的预测精度。
Flare V3内置了FEP计算工作流,支持GPU加速计算。它将AMBER Tools,OpenMM,LOMAP,Sire和BioSimSpace等开源工具与Cresset专业、直观特性相结合,为完全自动化的FEP工作流程提供了用户友好的界面(图1)。
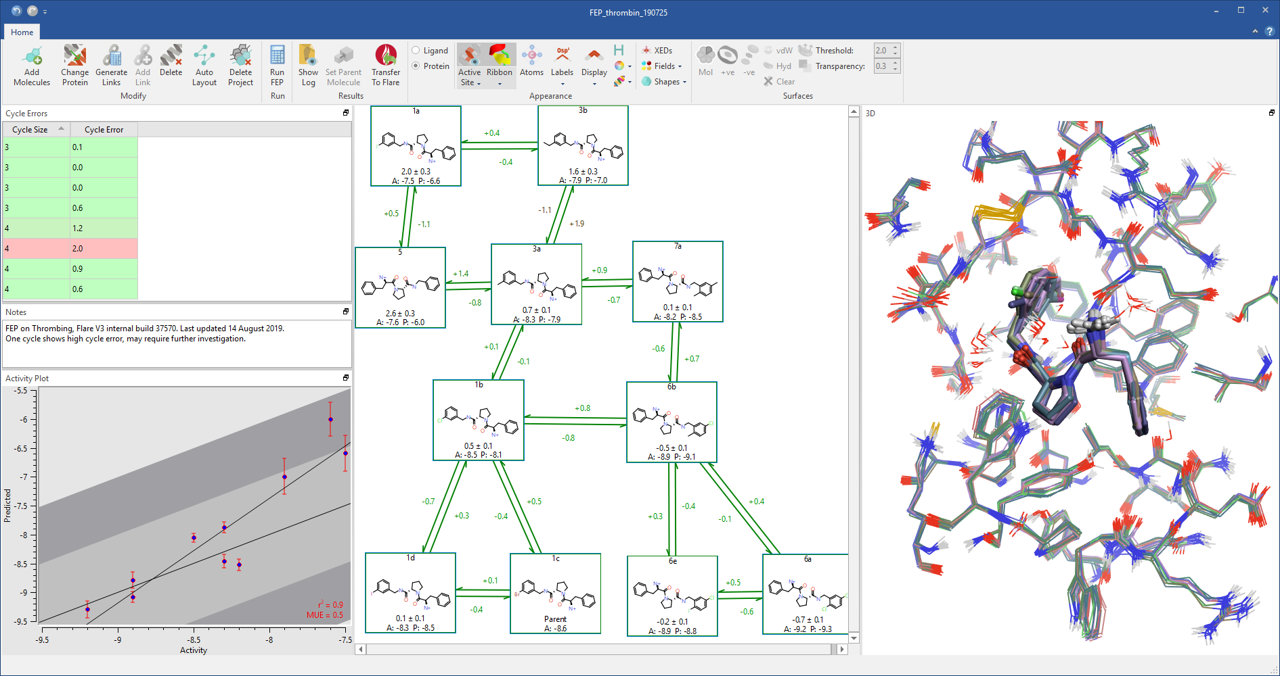
Figure 1. Flare V3全自动的FEP工作流
Flare V3中的FEP工作流程已用多种数据集完成内部验证,包括FEP +数据集作为参考基准。初步的结果见表1,FLARE V3的FEP计算结果与Schrodinger的FEP+(Wang et al.)相当,大大地优于AMBER的GPU-TI(Song et al.)。通过Flare图形用户界面和Flare Python API,可以完全控制模拟参数,使您能够探索和设定给定配体集合及其靶蛋白的理想条件。
Table 1. 初步的FEP基准数据测试结果
| Data Set | Flare V3/UoE | Wang et al. | Song et al. | |||
|---|---|---|---|---|---|---|
| R | MUE | R | MUE | R | MUE | |
| Thrombin | 0.88±0.04 | 0.35±0.04 | 0.71±0.24 | 0.76±0.13 | 0.76 | 0.46 |
| TYK2 | 0.87±0.02 | 0.60±0.04 | 0.89±0.07 | 0.75±0.11 | 0.57 | 1.07 |
| PTP1B | 0.83±0.04 | 0.84±0.06 | 0.80±0.09 | 0.89±0.12 | 0.71 | 1.06 |
| JNK1 | 0.81±0.02 | 0.85±0.04 | 0.85±0.07 | 0.78±0.12 | 0.47 | 1.07 |
| MCL1 | 0.79±0.02 | 1.30±0.06 | 0.77±0.05 | 1.16±0.10 | 0.65 | 1.52 |
| BACE | 0.78±0.03 | 1.08±0.05 | 0.78±0.07 | 0.84±0.08 | 0.43 | 1.20 |
| p38 | 0.72±0.04 | 1.44±0.05 | 0.78±0.07 | 0.84±0.08 | 0.38 | 1.20 |
| CDK2 | 0.69±0.09 | 1.02±0.08 | 0.48±0.19 | 0.91±0.12 | 0.47 | 0.97 |
- Wang, L.; Wu, Y.; Deng, Y.; Kim, B.; Pierce, L.; Krilov, G.; Lupyan, D.; Robinson, S.; Dahlgren, M. K.; Greenwood, J.; et al. Accurate and Reliable Prediction of Relative Ligand Binding Potency in Prospective Drug Discovery by Way of a Modern Free-Energy Calculation Protocol and Force Field. J. Am. Chem. Soc. 2015, 137 (7), 2695–2703. https://doi.org/10.1021/ja512751q.
- Song, L. F.; Lee, T.-S.; Zhu, C.; York, D. M.; Merz, K. M. Using AMBER18 for Relative Free Energy Calculations. J. Chem. Inf. Model. 2019, 59 (7), 3128–3135. https://doi.org/10.1021/acs.jcim.9b00105.
Flare V3/FEP采用GPU加速计算,1对分子用1张GTX 1080显卡计算大约需要1天时间。
模板对接(Template Docking)
Lead Finder™(Flare中的对接算法)仅探索配体的两面角空间,因此配体起始的构象可能强烈地影响对接计算的结果。然而,手动控制3D起始构象是一项繁琐的工作,尤其是当大量的配体需要进行对接计算的时侯。
Flare V3中的模板对接(Template Docking)为您提供了一个实际的解决方案,解决了您有已知“模板”配体结合模式时的对接实验,并希望使用此信息来偏置同系物的对接结果。 当使用模板对接时,待对接的分子通过子结构与模板配体叠合,并且用叠合过的构象用于对接计算,这通常会得到改善的结果,如图2所示。

Figure 2. 左: 25个分子对接到PERK活性位点(PDB: 4G31);右: 4G31配体(浅绿色)作为模板进行的Template docking结果
其他新增功能
Flare V3还将提供分子动力学模拟、共价分子对接、基于配体的叠合等功能,还将与Blaze虚拟筛选平台整合。
联系我们
更多的Flare V3以及Cresset产品特性介绍,请阅读Mark Mackey的演讲稿。



