摘要:通过计算生成虚拟化学库以便用于各种虚拟筛选方法,是增加找到新苗头化合物可能性的方法之一。基于反应枚举是一种常用的虚拟库生成方法。本文以JAK3共价抑制剂集中库的生成为例,演示了如何用Flare V6的Library Enumeration将用户提供的试剂根据预先定义的反应枚举生成一个虚拟的化合物库。
肖高铿/2023-01-14
前言
苗头化合物发现是小分子药物发现的起点,是最关键的阶段之一[1]。通过计算生成虚拟化学库以便用于各种虚拟筛选方法,是增加找到新苗头化合物可能性的方法之一。因此,许多研究人员正在通过不同的计算方法开发新的从头生成化合物库与”按需定制 “库[2]。例如,Reymond等人[3]用从头生成方式枚举原子数高达17的有机小分子,生成的GDB-17化合物库包含了1600多亿个化合物。另一种方式是基于反应枚举得到虚拟库,比如Humbeck等人[4]报道了用传统组合化学常用的反应来枚举生成了9500万分子的CHIPMUNK虚拟库。制药公司用商品化学试剂基于流行的或文献报道反应开发的虚拟库的例子也很多,比如勃林格殷格翰公司的BI-Claim[5],礼来公司的Proximal Collection[6]以及辉瑞公司的PGVL[7]。在学术界著名的ZINC数据库里除了包含实体库之外,还包含此类按需定制的虚拟库并成功用于虚拟筛选发现新骨架的活性化合物[8]。化学品供应商也用这种方法生成 “按需定制 “的虚拟库,如 “Readily Accessible”(REAL)数据库和REAL Space是迄今为止最大的基于合成可及性的虚拟化合物库[9]。
在实际研究中,我们通常针对特定靶标创建一个化学空间覆盖范围不是非常大的组合库,称之为集中库(Focused library)。比如之前演示过的靶向KRASG12C共价别构抑制剂[10]与JAK3共价抑制剂[11]的化合物库就是典型的集中库。
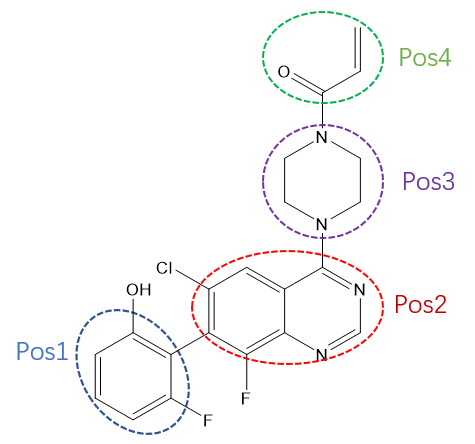
图1. KRASG12C共价别构抑制剂组合库设计策略。
在KRASG12C共价别构抑制剂组合库设计算例中[10],我们采用了基于片段的方法规划建库策略,如图1所示:以ARS-1620为模板并将其碎片化为4个片段,然后定义每个位置需要用什么样的片段来代替。
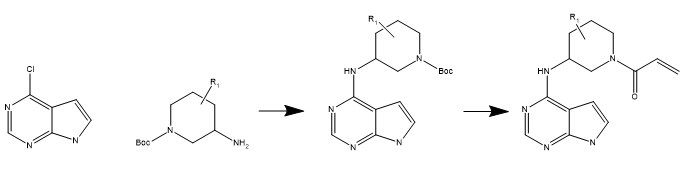
图2. JAK3共价抑制的合成路线
在JAK3共价抑制剂组合库设计中[11],以Thorarensen等人[12]报道的PF-06651600为模板,并根据其发现过程中系列化合物的合成路线(如图2所示),用KNIME调用RDKit组件构建工作流生成基于反应的组合库。
然而,有很多读者反馈,虽然有KNIME界面可以使用,但是用SMARTS写个反应式依然是创建虚拟组合库的挑战。正值Flare V6发布,其中一个新特性是提供了一个基于反应的组合库枚举工具Library enumeration[13],它内置了50多个常见的化学反应,极大地避免了反应式的撰写,让建库变得简单、高效。本文的主要目的是:以JAK3共价抑制剂组合库设计为例,描述如何用Flare V6的Library enumeration从试剂开始生成靶向JAK3的共价抑制剂集中库。
操作步骤
合成路线规划与试剂
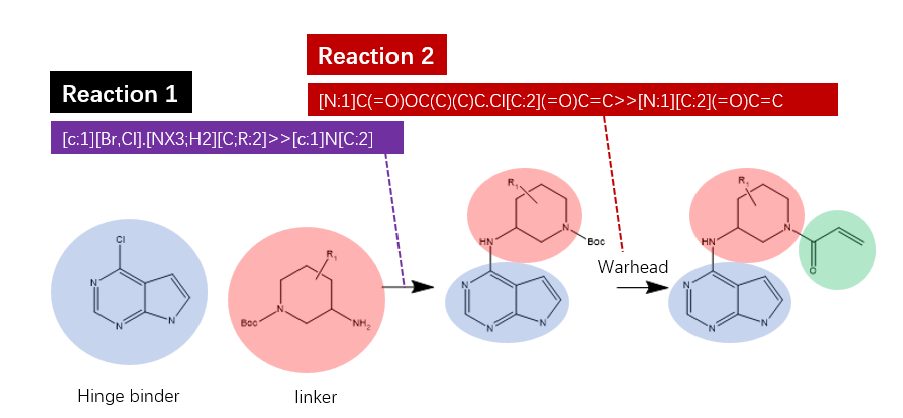
如图2所示,采用两步反应:1)4个铰链区结合片段(如图3所示)与185个Boc-保护的3-氨基哌啶连接臂发生N烷基化反应;2)上一步的产物脱去BOC保护基与丙烯酰氯反应获得终产物。
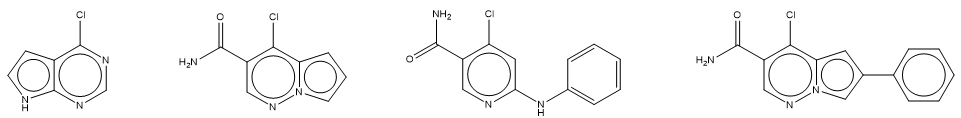
图3. 4个铰链区结合片段
每个试剂都保存为SDF文件,备用:
- 氯取代的芳香杂环铰链区结合片段:jak3_hinge_binder.sdf
- Boc-保护的连接臂:chem-space-jak3-linker.sdf
- 与Cys共价结合的弹头:warhead.sdf
仅是从BMS的文献里[14]里取4个片段用于演示,并没有查阅很多文献,因此并不全面。
连接臂片对段来源于Boc-保护的3-氨基哌啶衍生物是关键的中间体,是从试剂供应商chem-space经过子结构搜索而来,具体方法见前文[11]。
这里只有一个丙烯酰氯,当然你可以放很多不同的共价结合弹头。
用Library enumeration生成虚拟化合物库
首先,让铰链区结合片段与Boc-保护的3-氨基哌啶进行一个N-烷基化反应。点击Flare | Ligand | Library enumeration,弹出Library enumeration对话框。
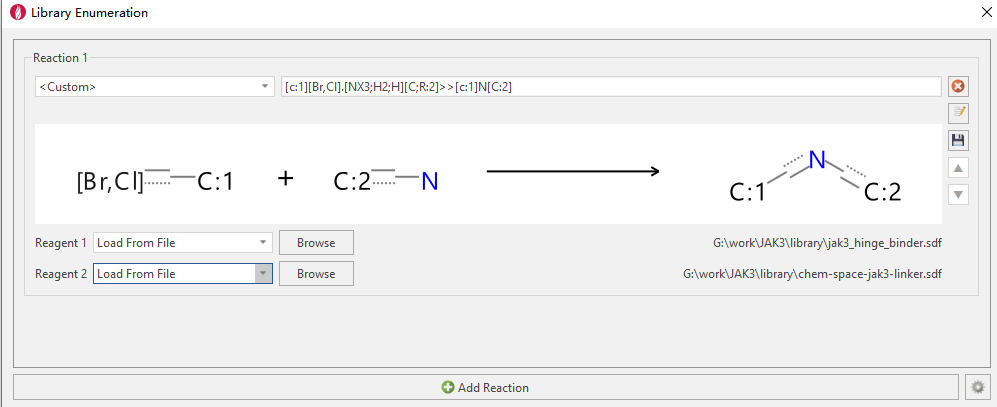
图4. 铰链区结合片段与Boc-保护的3-氨基哌啶的N-烷基化反应
如图4所示,在Reaction 1的下拉菜单里选择Custom,然后在旁边的对话框里输入:
[c:1][Br,Cl].[NX3;H2][C;R:2]>>[c:1]N[C:2] |
然后在Reagent 1里点击Load from file,读入铰链区结合片段hinge_binder.sdf;相似的,在Reagent 1选项框里点读入Boc-保护的3-氨基哌啶chem-space-jak3-linker.sdf。
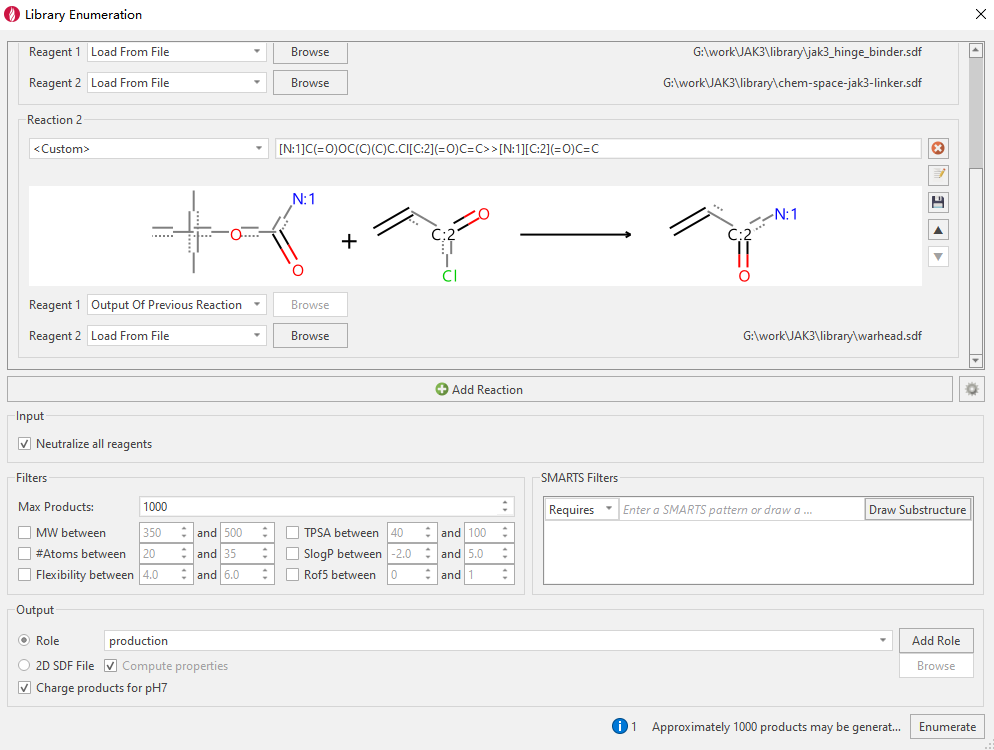
图5. Boc-哌啶与丙烯酰氯成酰胺反应
现在可以开始第二步反应,点击Add Reaction按钮,如图5所示,在Library Enumeration对话框的Reaction 1下方会出现新的反应设置窗口Reaction 2, 在下方的下拉菜单里选择Custom,然后在旁边的对话框里输入:
[N:1]C(=O)OC(C)(C)C.Cl[C:2](=O)C=C>>[N:1][C:2](=O)C=C |
这个反应意味着丙烯酰氯的碳基C与连接臂的Boc-N之间会形成一个N-C键,同时Boc片段与氯原子离去,得到对哌啶氮原子的丙烯酰化反应。
如图5所示,在Reagent 1处点击下拉菜单,选择Output of the Previous Reaction,以指示使用上一步的产物作为这一步的试剂1;在Reagent 2点击下拉菜单,选择Load from file,读入丙烯酰氯文件,即warhead.sdf
其它的选项设置如下:
- Input | Neutralize all regent: Yes (打钩)
- Filter | max production: 1000
- Filter 其它性质使用默认(不设置)
- Output | Role: production
- Output | Charge products for pH7: Yes
现在,点击enumerate按钮,开始按预设的反应枚举组合库,大约1钟即可完成,在配体表单区可以看到多了一个prodution的role,含有645个化合物。
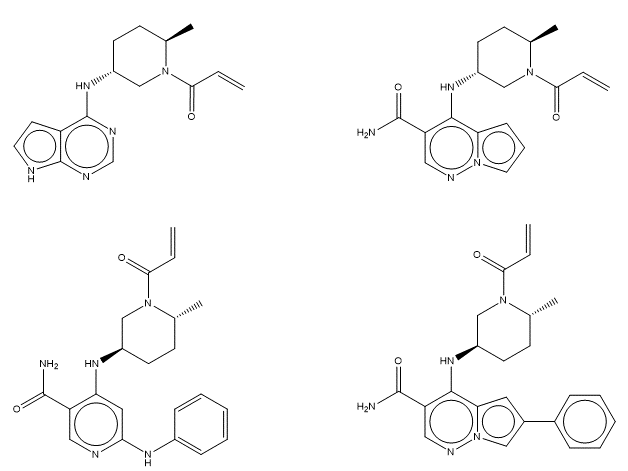
图6. 四种hinge binder与同一个连接臂反应,连接上共价结合弹头
随机对一个连接臂系列产物结构进行检查,如图6所示,可以发现4个不同的Hinge Binder都与之反应的产物都得到了,这说明我们预定义的反应确实遍历了全部的试剂。
接下来可以做什么
- 共价对接虚拟筛选
- 药效团虚拟筛选
- 用Ignite™进行3D虚拟筛选
如之前的博文所述[15],可以采用受体立体碰撞消除(SCAR)策略用常规分子对接工具对生成的数据库进行虚拟筛选。还可以用Flare的Covalent docking进行虚拟筛选。
Ligandscout是成熟的采用药效团技术进行共价抑制剂的虚拟筛选工具,比如Schulz等人[16]用该方法发现靶向鼻病毒3C Proteases的共价抑制剂。此外,我也提供了多个演示算例[10,17]。
利用现有的试剂与反应,我们经常可以枚举出超级大的数据库,比如包含100多亿的化合物库。常规的虚拟筛选不能在足够短、且成本足够低的进行虚拟筛选,而使用Ignite,正如在Bayer里的一个测试表明,可以用100个CPU在4个小时内完成虚拟筛选。目前Ignite不以软件方式提供本地安装,而仅以技术服务的方式间接使用。了解更多的:Ignite。
相关主题
SPARK在分子生长、片段连接、基团替换、水分子替换等策略设计新分子时,也可以采用基于反应的策略。具体的算例参见用ADMET Predictor与SPARK重现和记黄埔的PI3Kγ/δ双重抑制剂优化的过程。在这个算例中,我们用图7的环合反应来对Spark试剂数据库进行虚拟筛选,以确保骨架跃迁计算实验所得到的化合物是合成可行的。
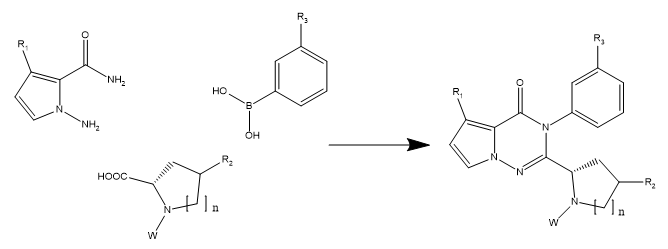
图7. 化合物6-26基于反应的集中库设计策略
文献
- Yan XC, Sanders JM, Gao Y-D, Tudor M, Haidle AM, Klein DJ et al (2020) Augmenting hit identification by virtual screening techniques in small molecule drug discovery. J Chem Inf Model. https://doi.org/10.1021/acs.jcim.0c00113
- Walters WP, Patrick WW (2019) Virtual chemical libraries. J Med Chem. https://doi.org/10.1021/acs.jmedchem.8b01048
- Ruddigkeit L, van Deursen R, Blum LC, Reymond J-L (2012) Enumeration of 166 billion organic small molecules in the chemical universe database GDB-17. J Chem Inf Model 52:2864–2875
- Humbeck L, Weigang S, Schäfer T, Mutzel P, Koch O (2018) CHIPMUNK: A virtual synthesizable small-molecule library for medicinal chemistry, exploitable for protein-protein interaction modulators. ChemMedChem 13:532–539
- Lessel U, Wellenzohn B, Lilienthal M, Claussen H (2009) Searching fragment spaces with feature trees. J Chem Inf Model 49:270–279
- Nicolaou CA, Watson IA, Hu H, Wang J (2016) The Proximal Lilly Collection: mapping, exploring and exploiting feasible chemical space. J Chem Inf Model 56:1253–1266
- Hu Q, Peng Z, Sutton SC, Na J, Kostrowicki J, Yang B et al (2012) Pfizer Global Virtual Library (PGVL): a chemistry design tool powered by experimentally validated parallel synthesis information. ACS Comb Sci 14:579–589
- Lyu J, Wang S, Balius TE, Singh I, Levit A, Moroz YS et al (2019) Ultra-large library docking for discovering new chemotypes. Nature 566:224–229
- REAL Database – Enamine. https://enamine.net/library-synthesis/real-compounds/real-database. Accessed 13 January 2022.
- 肖高铿. 计算赋能工作流识别全新KRASG12C共价别构抑制剂. 墨灵格的博客.http://blog.molcalx.com.cn/2021/05/25/bayer-covalent-binder-for-kras.html.
- 肖高铿. KNIME教程|基于反应的组合库生成. 墨灵格的博客. http://blog.molcalx.com.cn/2018/07/25/reaction-based-library-design.html
- Thorarensen, A.; et al. Design of a Janus Kinase 3 (JAK3) Specific Inhibitor 1-((2 S ,5 R )-5-((7 H -Pyrrolo[2,3- d ]Pyrimidin-4-Yl)Amino)-2-Methylpiperidin-1-Yl)Prop-2-En-1-One (PF-06651600) Allowing for the Interrogation of JAK3 Signaling in Humans. J. Med. Chem. 2017, 60 (5), 1971–1993.
- Flare V6预告——药化先睹为快. 墨灵格的博客. http://blog.molcalx.com.cn/2022/04/26/sneak-peek-flare-v6-medicinal-chemists.html
- Kempson, J.; Ovalle, D.; Guo, J.; Wrobleski, S. T.; Lin, S.; Spergel, S. H.; Duan, J. J. W.; Jiang, B.; Lu, Z.; Das, J.; et al. Discovery of Highly Potent, Selective, Covalent Inhibitors of JAK3. Bioorg. Med. Chem. Lett. 2017, 27 (20), 4622–4625. https://doi.org/10.1016/j.bmcl.2017.09.023.
- 肖高铿. 用常规分子对接进行共价抑制剂的虚拟筛选. 墨灵格的博客. http://blog.molcalx.com.cn/2020/03/22/covalent-inhibitor-docking-vs.html
- Schulz, R., Atef, A., et al. (2018). Phenylthiomethyl Ketone-Based Fragments Show Selective and Irreversible Inhibition of Enteroviral 3C Proteases. Journal of Medicinal Chemistry, 61(3), 1218–1230. https://doi.org/10.1021/acs.jmedchem.7b01440. 附中文介绍
- 肖高铿. 共价键药效团模型与虚拟筛选. 墨灵格的博客. http://blog.molcalx.com.cn/2018/05/15/ligandscout-tutorial-covalent-vs.html



