摘要:系综共价对接(Ensemble Covalent Docking)是Flare最近引入的一个新功能。本文以脯氨酰寡肽酶(POP或 PREP)抑制剂KYP-2047为例,演示了如何在Flare中实现将一个分子共价对接到多个蛋白构象里;并且还演示了两种蛋白构象的获取方法:修改蛋白侧链构象与导入同一蛋白的多个PDB结构。
作者:Jessica Plescia/14 July 2023
编译:肖高铿
前言
Docking是Flare™平台最受欢迎的功能之一,可为用户提供有关新分子设计的详细反馈、高度富集的虚拟筛选以及出色的结合模式预测。 目前,可用的对接实验包括标准(Normal)、共价(Covalent)和系综(Ensemble)等三种模式。标准模式对接的特征为配体在指定空间能量网格内与蛋白对接。共价对接增加了蛋白的亲核残基和配体中的反应弹头之间共价键的形成。最后,系综对接允许用户同时将标准对接应用于多个蛋白构象。
最近,我们新增了系综共价对接(Ensemble Covalent docking),该选项组合了共价与系综对接特性:用户可以在一次对接实验里共价对接分子于同一个蛋白的多个构象。
系综共价对接入门
在开始系综共价实验之前,需要先对蛋白结构进行准备、序列比对以及叠合。在导入蛋白质结构时可以方便地用“Protein Preparation”进行结构准备,然后使用“Sequences”选项卡下的“Align”功能进行序列比对(图 1)。 再然后用“Superpose”功能将蛋白进行3D叠合,以便它们适用于系综共价对接:
图1: 在Flare里选用“Superpose”
一旦在3D中完成蛋白结构准备、序列比对与叠合,请在“Proteins”表单中选中您想要将配体对接的所有蛋白。系综共价对接实验可以在“3D Pose”选项卡的“Dock”下拉菜单下找到:
图2: 在Flare的Dock下拉菜单中选择Ensemble Covalent对接工作流
在Ensemble Covalent Docking计算面板(图 3)中,您可以用共晶配体的位置或从活性位点选取原子来轻松设置配体对接的区域,以及从蛋白里选择代表性的催化亲核残基。在这里,我们选择了脯氨酰寡肽酶(prolyl oligopeptidase)的Ser554,Flare将通过3D邻近度识别其它蛋白结构里对等的催化丝氨酸残基。 当这些蛋白具有不同的催化残基时,您可以使用“残基”下拉菜单里选择每个蛋白质中的共价残基:
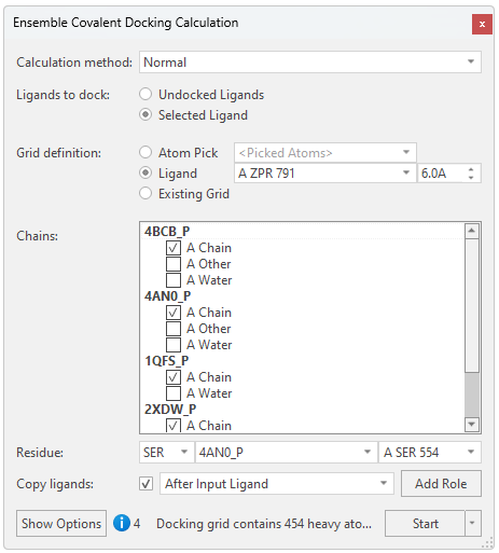
图3.在Flare里创建系综共价对接实验
现在,我们已经准备好开始系综共价对接实验了!
系综共价对接实验算例:KYP-2047
我们以一种脯氨酰寡肽酶(POP或 PREP)抑制剂KYP-2047为例来说明Flare这一新功能,目前人们已经开始研究POP在神经退行性疾病1、黄斑变性2和胶质母细胞瘤3中的作用。图4显示了该分子的2D和3D结构。
KYP-2047已获得与POP的共晶结构4,并且可从RCSB获得(pdb 4AN0)。图4(右)显示了POP活性位点的蛋白质表面和相互作用残基,其中丝氨酸554与KYP-2047共价结合。
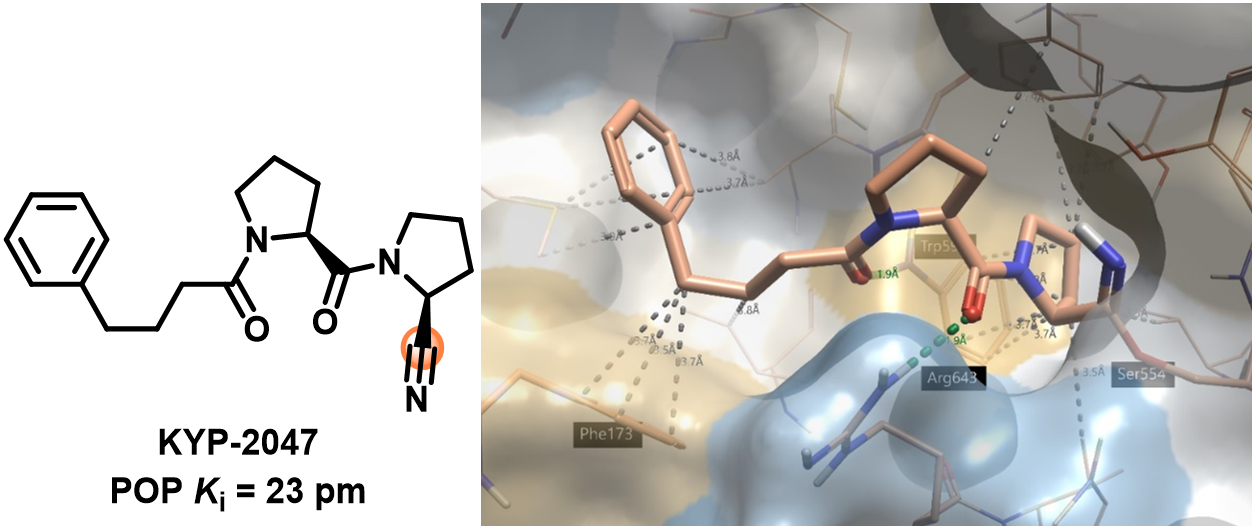
图4.左:具有POP活性的KYP-20475化学结构式,高亮部分为共价弹头。右:KYP-2047共价结合到POP(pdb 4AN0)。蛋白-配体相互作用采用Flare默认的着色方案:绿色 = 强氢键, 灰色 = 疏水;蛋白表面用疏水性进行着色6:蓝色 = 亲水, 浅褐色 = 疏水。
对接到不同的蛋白构象
系综共价对接作用之一是模拟蛋白“柔性”的对接。实现这一目标的方法之一是最小化或修改单个残基的3D构象,并观察对结合模式的影响:一次改变一个残基可以进行更受控的实验。对于该蛋白酶,在活性位点中稳定底物和抑制剂的关键残基之一是ARG643。在这里,我们稍微旋转一下其侧链,以允许活性位点构象发生变化(图 5,左)。然后将KYP-2047与POP的五种不同构象进行对接,这5个POP结构仅精氨酸侧链的位置有所不同(图 5,右)。在配体表单中列出了得分最高的结合模式,并按 LeadFinder™ Rank Score进行排序。与每个结合模式相关联的蛋白构象列在表单的蛋白列中(图 6)。

图5. 左:POP(pdb 4AN0)活性位点, 包括旋转精氨酸侧链得到的几个构象。右:用共价系综对接计算将KYP-2047对接到全部蛋白构象,打分最高的两个结合模式,紫色化合物为共晶配体;蛋白-配体相互作用采用Flare默认的着色方案:绿色 = 强氢键, 灰色 = 疏水;蛋白表面用疏水性进行着色6:蓝色 = 亲水, 浅褐色 = 疏水。
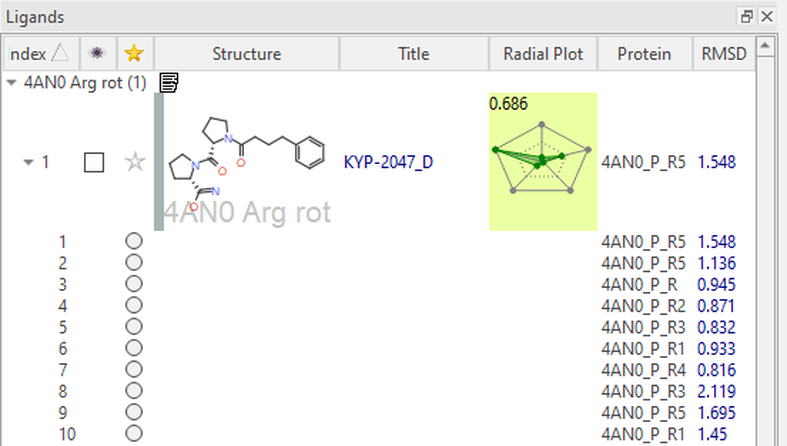
图6: 系综共价对接实验结果,呈现在Flare配体表单中的蛋白列
在图5中,我们可以看到KYP-2047两个打分最高的结合模式始终与共结晶配体对齐 (RMSD = 1.5 Å, 1.1 Å),并在活性位点中发生了必要的关键相互作用。1,4-二羰基子结构与Trp595和Arg643形成氢键,尽管后者残基的构象不同。我们还可以看到,与大部分疏水性结合口袋形成有许多疏水性接触。除了此处所示的示例之外,还可以生成不同亲核残基侧链构象的蛋白质结构以供探索。
另一种实现“柔性”对接的方法是简单地同时对接到同一蛋白的多个X-衍射结构。每个3D模型都略有不同,从而导致活性位点的3D结构存在一定变化。例如,PDB中有很多POP的结构。 仅比对其中四个结构(图 7、4AN0、2XDW、1QFS、4BCB)后,我们可以看到关键活性位点残基的 3D坐标存在轻微变化。
图7. 4个POP晶体结构(pdb 4AN0、2XDW、1QFS、4BCB)的关键活性位点残基
在这些准备好的蛋白上运行KYP-2047的系综共价对接,产生与所有晶体结构对接的KYP-2047结合模式。图8显示了与共结晶KYP-2047 相比的打分最佳结合模式的3D视图。
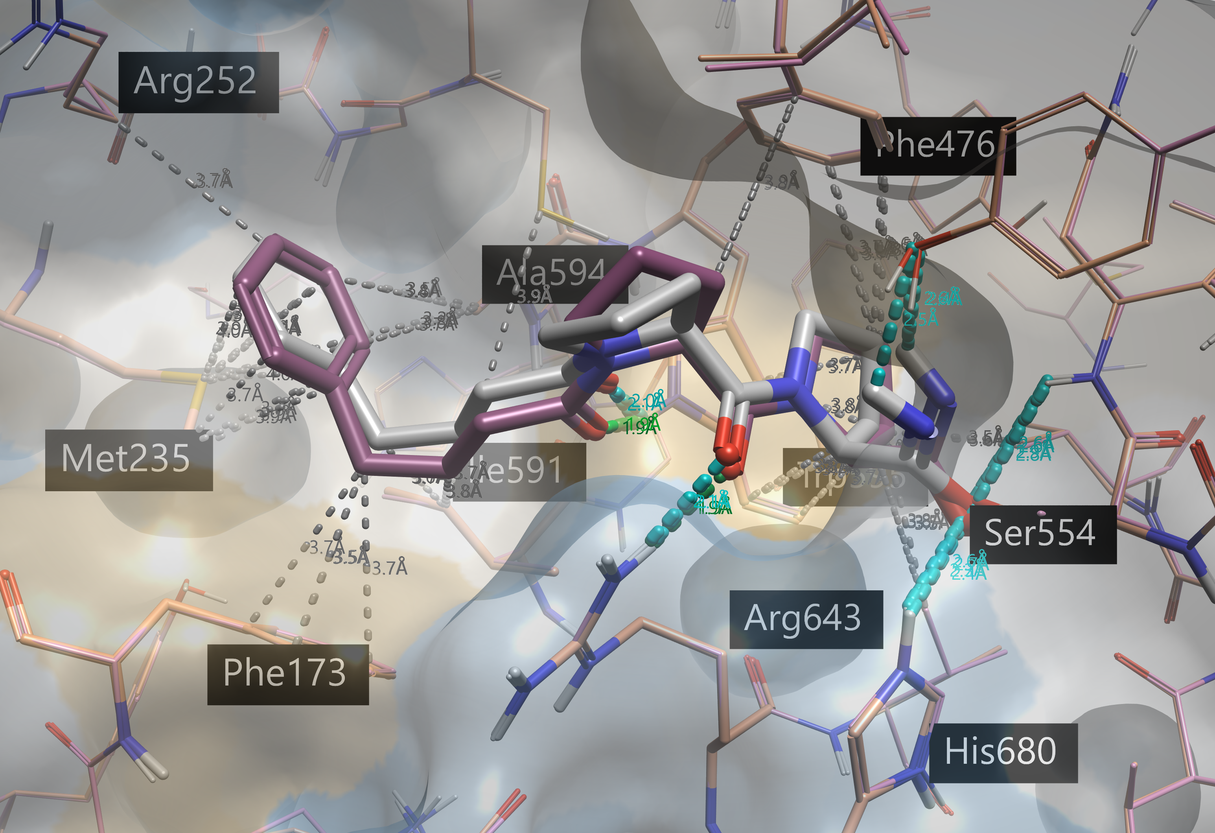
图8.与四个POP结构进行系综共价对接得到KYP-2047打分最高的结合模式(灰色),相关联的PDB 4BCB 以粉色显示。共晶 KYP-2047(PDB 4AN0,橙色)显示为紫色。蛋白-配体相互作用采用Flare默认的着色方案:绿色 = 强氢键, 灰色 = 疏水;蛋白表面用疏水性进行着色6:蓝色 = 亲水, 浅褐色 = 疏水。
打分最佳的前六种对接结合模式(每个蛋白结构中至少生成一种结合模式)与共晶配体非常相似(RMSD小于1.5 Å)。与共晶KYP-2047一样,1,4-二羰基与色氨酸和精氨酸残基形成氢键,而苄基参与疏水相互作用和芳香相互作用。
系综共价对接的高级选项
在上述任一情况下,我们都可以使用系综共价对接将我们感兴趣的配体与每个感兴趣的蛋白对接,并要求Flare输出每个蛋白的最佳结合模式(而不是最佳的系综结合模式)。要使用此选项,请单击“Ensemble Covalent Docking Calculation”面板中的“Show Options”:
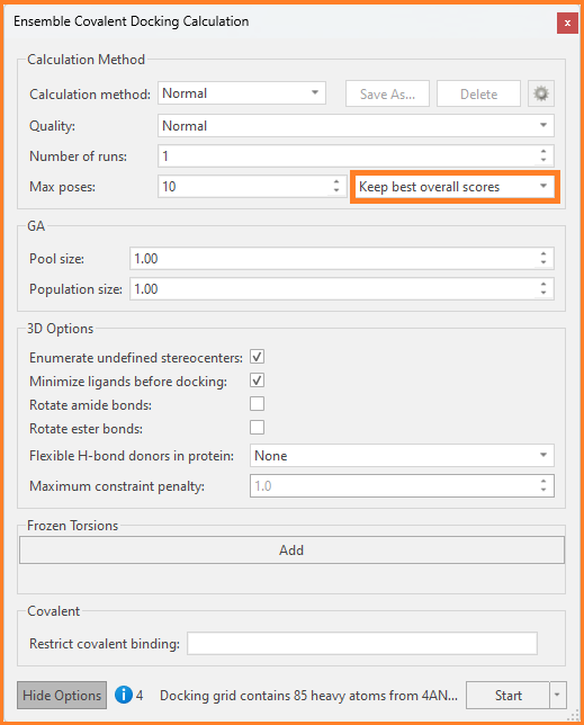
图9: Flare的系综共价对接计算面板
在您自己的项目中尝试使用系综共价对接
如KYP-2047算例所示,Flare的Ensemble Covalent docking可以通过对接到一种蛋白质的几种不同构象或对接到同一蛋白的不同pdb来使用。
如果您想在您的项目中测试系综共价对接功能,同时可以访问Flare中完整的分子建模功能组合,您可以立即申请免费试用。作为试用过程的一部分,您将获得安装平台和访问其广泛功能的全面支持,同时可以自由发布生成的任何结果并将其用于进一步的研究。如果您在自己的系综共价对接实验中遇到任何技术问题,请随时联系Cresset技术支持。
文献
- Eteläinen, T. S., Silva, M. C., Uhari-Väänänen, J. K. et al. A prolyl oligopeptidase inhibitor reduces tau pathology in cellular models and in mice with tauopathy. Sci Transl Med 2023, 15(691) https://doi.org/10.1126/scitranslmed.abq2915
- Toppila, M., Hytti, M., Korhonen, E. et al. The Prolyl Oligopeptidase Inhibitor KYP-2047 Is Cytoprotective and Anti-Inflammatory in Human Retinal Pigment Epithelial Cells with Defective Proteasomal Clearance. Antioxidants 2023, 12(6), 1279. https://doi.org/10.3390/antiox12061279
- Scuderi, A.S., Casili, G., Ardizzone, A. et al. settingsOrder KYP-2047, an Inhibitor of Prolyl-Oligopeptidase, Reduces GlioBlastoma Proliferation through Angiogenesis and Apoptosis Modulation. Cancers 2021, 13(14), 3444; https://doi.org/10.3390/cancers13143444
- Kaszuba, K., Rog, T., Danne, R., et al. Molecular dynamics, crystallography and mutagenesis studies on the substrate gating mechanism of prolyl oligopeptidase. Biochimie 2012, 94: 1398-1411.
- Jalkanen, A. J., Juuso V. L., Forsberg, M. M. KYP-2047 Penetrates Mouse Brain and Effectively Inhibits Mouse Prolyl Oligopeptidase. Basic Clin Pharmacol Toxicol 2014, 114(6), 460-463. https://doi.org/10.1111/bcpt.12184
- Wimley, W. C., White, S. H. Experimentally determined hydrophobicity scale for proteins at membrane interfaces. Nature Struct. Biol. 1996, 3, 842-848.




