摘要:主-客体构型搜索(Host-Ligand Coordination Search)与分子团簇(molecular cluster)搜索的帮助遍历主客体复合物或分子团簇分子间相对位置与取向。在这个功能里,一个分子被定义为主体(host),另一个分子或离子被定义为配/客体(ligand),客体围绕在主体周围。该功能可以用于研究分子团簇以及诸如分子识别、主-客体化学之类的超分子化学研究。
作者:肖高铿/2016-04-13
1. 前言
从CONFLEX 7.C开始,增加了主-客体构型搜索(Host-Ligand Coordination Search)与分子团簇(molecular cluster)搜索的功能:帮助遍历主客体复合物或分子团簇分子间相对位置与取向。在这个功能里,一个分子被定义为主体(host),另一个分子或离子被定义为配/客体(ligand),客体围绕在主体周围。因此该功能可以用于研究分子团簇以及诸如分子识别、主-客体化学之类的超分子化学研究。
2. 醋酸二聚体分子的最小能量结构搜索
2.1 准备工作
用GaussView5、ChemAxon或ChemDraw先画好两个醋酸:
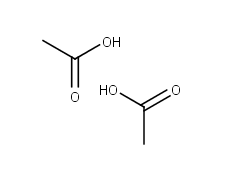
2.2 生成3D结构,保存
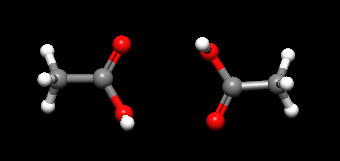
将上述画好的结构生成3D构象,保存为mol文件:demo.mol
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 | demo Mrv1611104131620373D 16 14 0 0 0 0 999 V2000 -3.9055 -0.6319 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -2.6428 0.1008 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 -1.4733 -0.5732 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -2.6437 1.3509 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 -4.2198 -0.8142 1.0277 H 0 0 0 0 0 0 0 0 0 0 0 0 -4.6665 -0.0444 -0.5138 H 0 0 0 0 0 0 0 0 0 0 0 0 -3.7732 -1.5840 -0.5138 H 0 0 0 0 0 0 0 0 0 0 0 0 -1.1960 -0.7329 0.9051 H 0 0 0 0 0 0 0 0 0 0 0 0 2.3671 2.7260 -0.1131 C 0 0 0 0 0 0 0 0 0 0 0 0 1.1444 1.9283 -0.1131 C 0 0 0 0 0 0 0 0 0 0 0 0 -0.0588 2.5401 -0.1131 O 0 0 0 0 0 0 0 0 0 0 0 0 1.2107 0.6800 -0.1131 O 0 0 0 0 0 0 0 0 0 0 0 0 2.6713 2.9246 0.9145 H 0 0 0 0 0 0 0 0 0 0 0 0 3.1577 2.1792 -0.6270 H 0 0 0 0 0 0 0 0 0 0 0 0 2.1850 3.6699 -0.6270 H 0 0 0 0 0 0 0 0 0 0 0 0 -0.3440 2.6852 0.7920 H 0 0 0 0 0 0 0 0 0 0 0 0 1 2 1 0 0 0 0 2 3 1 0 0 0 0 2 4 2 0 0 0 0 1 5 1 0 0 0 0 1 6 1 0 0 0 0 1 7 1 0 0 0 0 3 8 1 0 0 0 0 9 10 1 0 0 0 0 9 13 1 0 0 0 0 9 14 1 0 0 0 0 9 15 1 0 0 0 0 10 11 1 0 0 0 0 10 12 2 0 0 0 0 11 16 1 0 0 0 0 M END |
2.3 二聚体最小能量构象搜索
2.3.1 导入初始构象
将demo.mol拖放到Molecule Box,则在视窗区会看到一个二聚体分子:
2.3.2 二聚体构象搜索
点击 CONFLEX | Calculation 弹出CONFLEX Settings对话框。
将Calculation type设置为Conformation Search; 点击Detail settings,再点击Edit & Submit按钮,弹出CONFLEX对话框,输入:MMFF94S HLSEARCH
点击Submit按钮,开始执行构象搜索。
2.3.3 结果分析
计算完毕,重点关注demo.ls4, demo-F.mol, demo.sdf这三个文件。
- demo.ls4: 列出的构象信息,按能量从低到高排序,并给出玻尔兹曼分布。
- demo-F.mol: 最低能量的二聚体。
- demo.sdf: 包含了全部的低能构象集。
将demo.sdf拖放到Molecule Box,并用鼠标邮件点击demo.sdf,在Property Box会展示出搜索到的全部构象、构象能与玻尔兹曼分布。在Property box点击任意一个构象,在视窗就会展示对应的3D构象模型。图一展示最低能的那个二聚体构象, 图二展示了构象能最低的4个二聚体及其成氢键的O-O之间的距离。
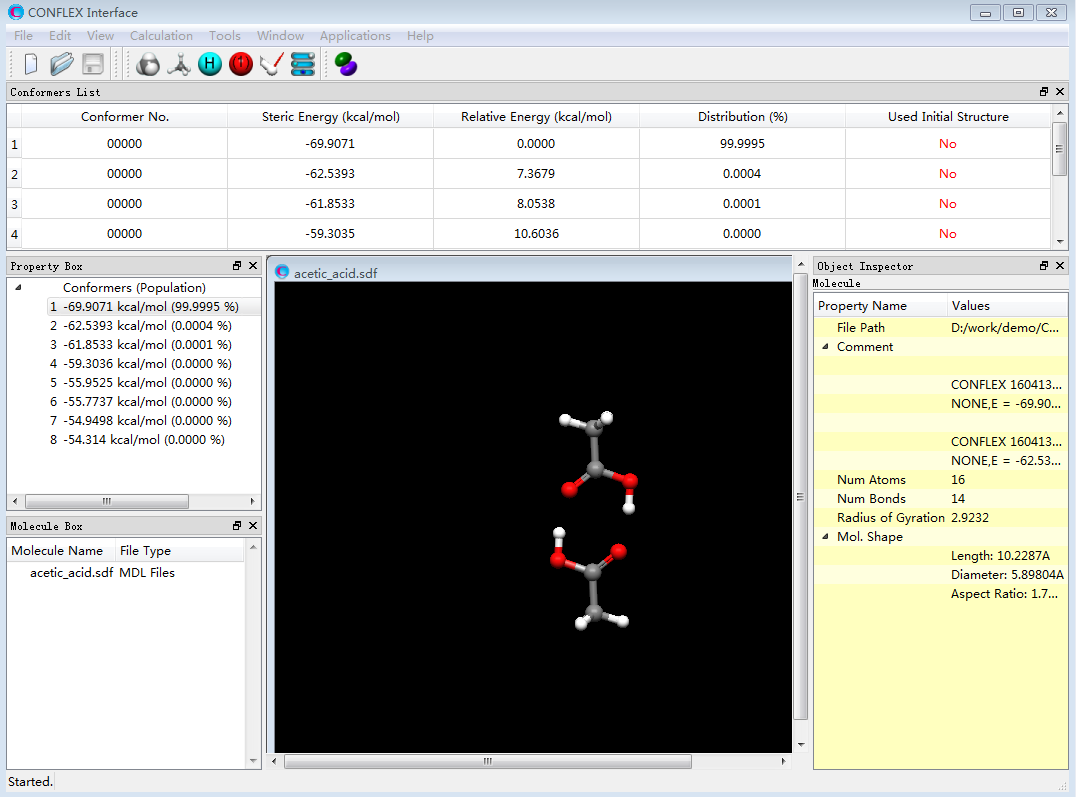
图一 最低能的二聚体构象
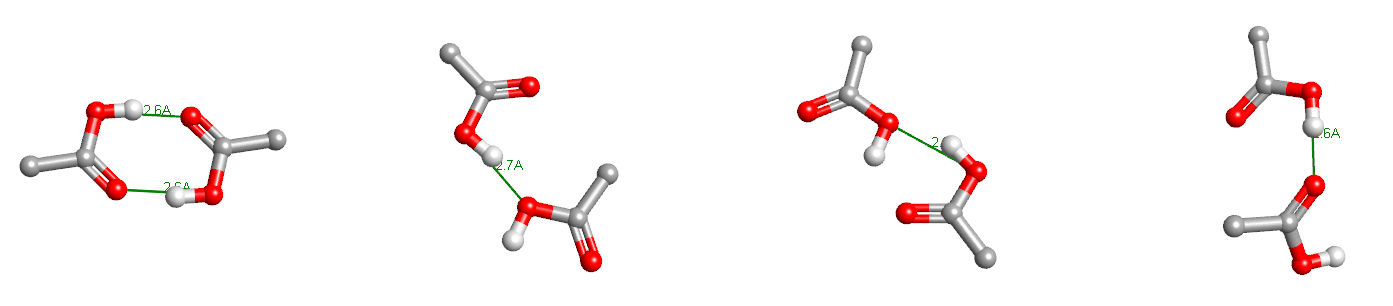
图二 四个最低能的二聚体构象及其成H-Hond相互作用的O-O距离
3. 软件试用
软件申请与下载:http://www.conflex.net
同时请提交试用license申请的中文信并提交:



