摘要:本文说明了pyFlare环境如何兼容、并轻松地与各种强大科学Python库进行集成。这种适应性强的脚本环境提供了对高级数据可视化和计算功能的访问,从而进一步扩展了现有的Flare功能。这使用户能够为其药物发现项目创建定制的自动化工作流,并与Flare GUI无缝集成。
作者:Oliver Hills/2024-05-02
编译:肖高铿
PyFlare是一个先进而强大的Python®环境,可从Cresset的CADD工作台FlareTM获取使用,支持开源科学Python库和代码库。PyFlare通过Flare Python API与原生Flare功能互操作,进一步扩展了Flare分子建模功能的庞大组合。PyFlare提供了开发自定义功能和自动化工作流的自由,以进一步加速分子设计项目。
在本文中,我们将展示药物发现环境中的一些示例,其中pyFlare环境通过高级数据可视化和计算功能得到进一步扩展。具体而言,我们将介绍化学空间可视化、化合物聚类、系综PCA和从PubChem中按名称检索化合物的工作流。
本文演示了pyFlare环境的适应性,以托管各种独特的Python库来支持您的药物发现工作。
化学空间可视化
能够绘制虚拟筛选实验探索的化学空间是识别重复的子结构和化学型的关键,确保优先考虑的化合物涵盖足够的化学多样性。与Pandas和Plotly一起使用,RDKit1和scikit-learn2功能(均在pyFlare环境中可用),可以在Flare中基于2D描述符相似性生成信息丰富化学空间可视化。
图1显示了存储在Flare配体表单中约1800个配体数据集的t-分布随机邻域(t-SNE)3降维示例。在Flare Python解释器窗口直接写入Python脚本,构建了一个由RDKit的2D Morgan/Circular指纹描述符构建的all-by-all Tanimoto相似性矩阵,并将其投影到二维图中。根据t-SNE投影,很容易在2D相似性空间中可视化不同的化学类别,对应于不同的化学系列。数据点之间的距离反映了它们在2D结构上的差异性。
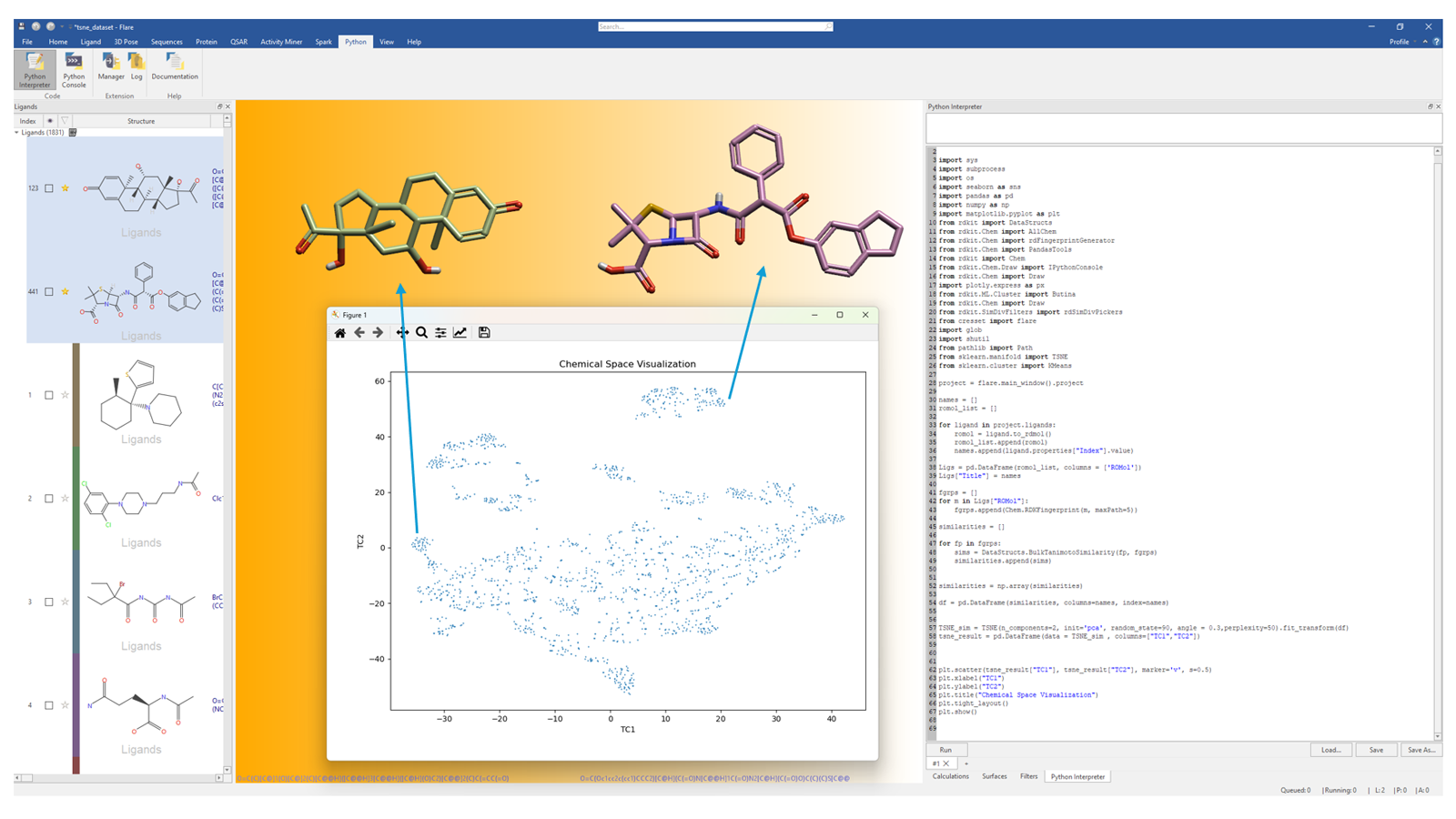
图1. 导入Flare化学结构多样的配体数据集的t-SNE投影到2D化学相似性空间。t-SNE图是通过直接写入Flare Python解释器小部件的Python脚本生成的。
化学系列聚类
在上一个示例中使用的相同Python库(即RDKit、Plotly和Pandas)也可用于使用Butina算法4对配体进行聚类,以便将采样的化学空间组织成离散的类。更具体地说,当使用Butina聚类算法时,类的质心与其成员表现出高度相似性,类内相似性反映了为聚类选择的Tanimoto阈值。 这为Flare基于3D/2D相似性的分级聚类方法提供了替代方法。在图2中,Python解释器中的Flare Python API脚本用于将配体表单中存储的配体处理进行“Butina聚类”。
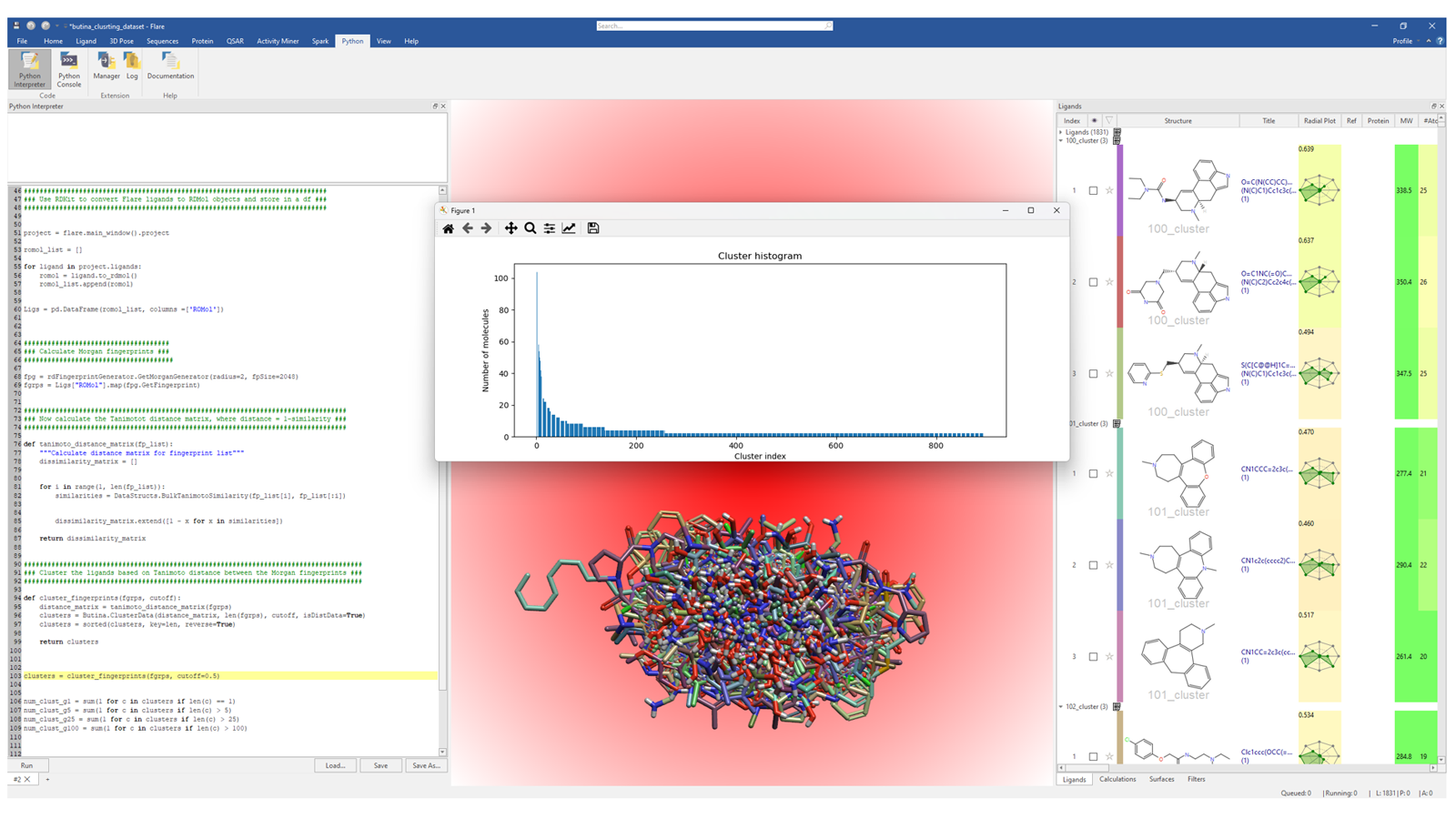
图2. 此Flare Python API脚本使用Butina聚类将Flare项目中的配体分组为单独的、结构紧凑的类。每个类中的分子数量显示在直方图中。
探索分子动力学模拟重复计算时的构象空间
对单个MD轨迹执行主成分分析(PCA)是一种在MD模拟过程中可视化分子体系采样的构象空间的常用方法。为了改进MD模拟数据的生物物理解释,通常进行重复模拟,以查看分子体系采样的构象空间在MD运行中是否一致。
PyFlare可以与外部库MDAnalysis5集成,编写Flare Python API脚本,进一步扩展Flare综合的MD轨迹分析功能,使Flare能够对Flare蛋白质表单中保存的同一分子体系的多个构象系综进行PCA分析。该计算的输出是“构象空间热图”(图3),显示了MD重复计算时蛋白质进入的所有相空间。这使得可以区分和比较在每次MD模拟中访问到的相空间。
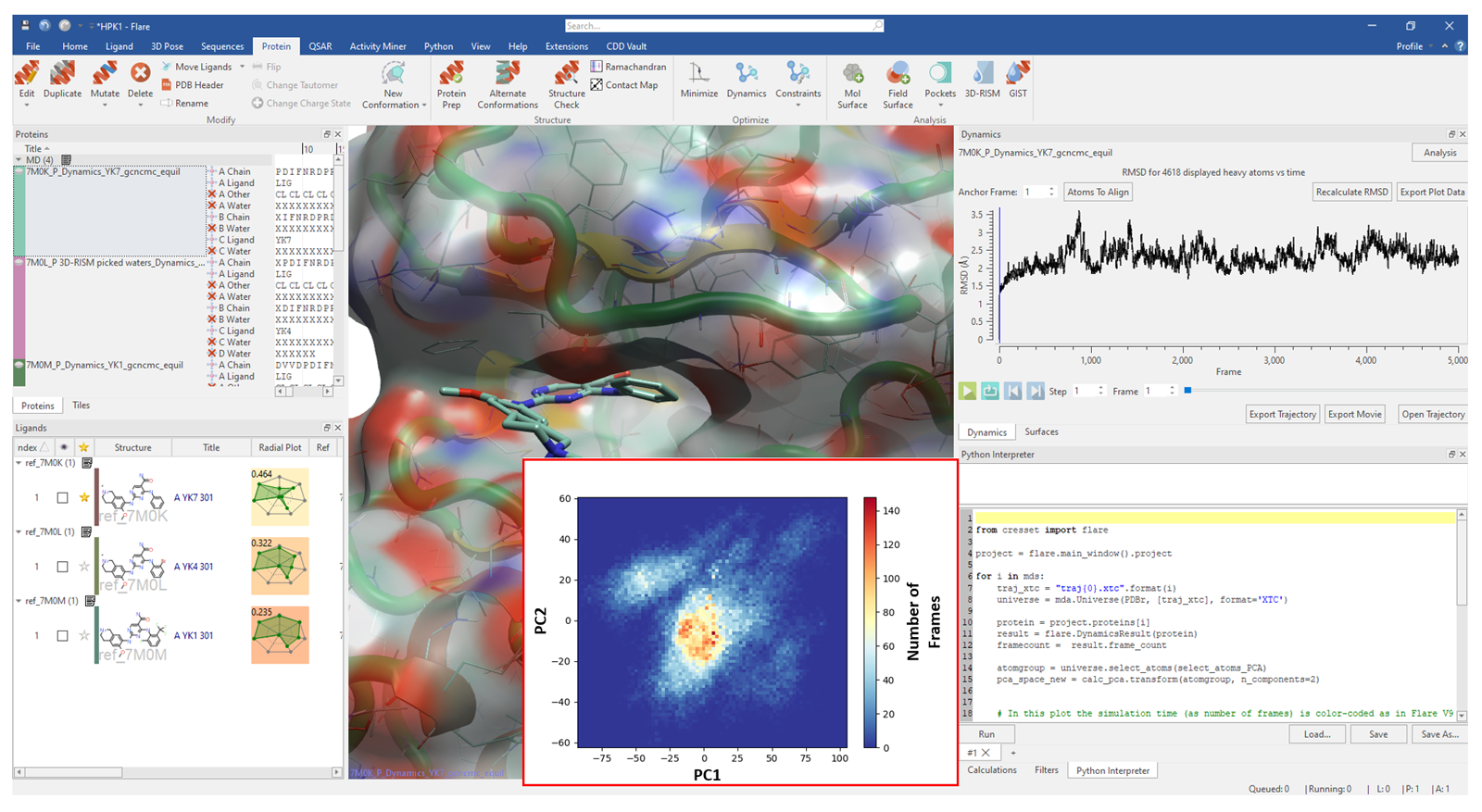
图3. 通过对存储在Flare蛋白质表单中的相同分子体系的动力学蛋白质系综的PCA分析生成的构象空间热图。
直接从PubChem下载配体到Flare
Flare提供了多种将配体添加到项目中的方法。它们可以从各种标准文件格式导入,使用编辑工具在3D视窗中手动绘制,或者通过键入SMILES字符串来创建。然而,它不会根据化学名称创建分子。令人鼓舞的是,这个问题可以通过 Flare Python API来克服,利用 PubChemPy6,一个开源的Python库,提供了一种方式在Flare中通过Python与PubChem交互的方法。
从存储在CSV文件的化合物名称列表开始,可以在实时Flare会话中启动Flare Python API脚本,读取化合物文件,并在PubChem数据库中搜索按名称匹配的条目,如图4所示。例如,当您希望导入到Flare GUI的化合物目录不包含SMILES字符串或2D/3D坐标时,非常有用。

图4. 仅根据提供的化合物名称将化合物从PubChem下载到Flare的工作流。
结论
本博文的示例说明了pyFlare环境如何兼容并可以轻松地与各种强大的科学Python库集成。这种适应性强的脚本环境提供了对高级数据可视化和计算功能的访问,从而进一步扩展了现有的Flare功能。这使用户能够为其药物发现项目创建定制的自动化工作流,并与Flare GUI无缝集成。
参考文献
- RDKit. https://www.rdkit.org/ (accessed 2024-04-16)
- scikit-learn: machine learning in Python — scikit-learn 1.4.2 documentation. https://scikit-learn.org/stable/ (accessed 2024-04-16)
- Maaten, L. van der; Hinton, G. Visualizing Data Using T-SNE. J. Mach. Learning Res 2008, 9 (86), 2579–2605
- Butina, D. Unsupervised Data Base Clustering Based on Daylight’s Fingerprint and Tanimoto Similarity: A Fast and Automated Way To Cluster Small and Large Data Sets. J. Chem. Inf. Comput. Sci. 1999, 39 (4), 747–750. https://doi.org/10.1021/ci9803381
- Michaud-Agrawal, N.; Denning, E. J.; Woolf, T. B.; Beckstein, O. MDAnalysis: A Toolkit for the Analysis of Molecular Dynamics Simulations. Journal of Computational Chemistry 2011, 32 (10), 2319–2327. https://doi.org/10.1002/jcc.21787
- Introduction — PubChemPy 1.0.4 documentation. https://pubchempy.readthedocs.io/en/latest/guide/introduction.html#pubchempy-license (accessed 2024-03-13)



