摘要:本文用CASF-2013数据集测试了Lead Finder的采样性能与打分性能。结果表明:重复进行3次计算,取打分最高前20、10、3个结果,dG Score的对接成功率分别为78.1%、68.2%、58.3%;而打分最高pose的成功率43.2%。本文还考察了Lead Finder三种打分函数的打分性能,发现dG Score预测值与实验值的Pearson系数为0.66,具有强的相关。dG Score打分函数的排序能力成功率为56.9%,仅次于并列第一的X-Score与ChemPLP@GOLD(成功率为58.5%)。
肖高铿 2018-05-24
更新:2018-06-02,添加了CASF-2013打分函数的排序能力
一. 测试目的
Lead Finder是一个比较新的软件,本文用CASF-2013数据集测试了Lead Finder的采样性能与打分性能。
二. 测试方法
1. 数据集
CASF2013是中国科学院上海有机研究所王仁小课题组开发的基准数据集1,2,共含195个复合物结构、覆盖65种药物靶标,广泛应用于分子对接软件的采样、打分以及虚拟筛选性能评估。
2. 软件
Lead Finder version 1804 build 1, 24 April 2018
3. 结构准备
蛋白结构:直接用CASF2013提供的protein.mol2文件。
配体3D结构: 用Openbabel将CASF2013数据集中的ligand.mol2转化为SMILES格式,再用CORINA从SMILES计算生成3D结构,未经处理直接给Lead Finder对接计算。
4. 格点文件(grid)生成
Lead Finder对接计算需要用能量的格点文件,直接从CASF2013的蛋白mol2文件与配体mol2文件用Lead Finder计算而得。典型的grid文件用生成命令如下:
1 2 | leadfinder --grid-only --protein=$10gs_protein.mol2 --ligand-reference=10gs_ligand.mol2 --save-grid=protein.grid |
其中10gs_protein.mol2与10gs_ligand.mol2是SCAF2013准备现成的蛋白、配体结构。
5. 分子对接计算
Lead Finder采用遗传算法,每次计算结果都不一样,因此重复计算三次,每次最多输出20个pose,合并三次计算结果。每个计算采用xp模式,并打开rta、rte、rtc选项,典型的算例如下:
1 2 | leadfinder -g protein.grid --ligand-reference=10gs_ligand.mol2 -li ligand.sdf -o lf_pose_1.sdf -l lf_report_1.log -rta -rtc all -rte -mp 20 --output-tabular=lf_score_1.csv --verbose |
其中protein.grid是上一步生成的格点文件,用来打分用;10gs_ligand.mol2是参比分子,用来计算对接pose的RMS;ligand.sdf是由CORINA生成的3D结构文件;输出文件lf_score_1.csv包含了每个pose的打分值、各个分值的成分、RMS值等等,这些值可以用于性能评估。
三. 结果
1. 对接计算引擎的采样性能(Sampleing power)评估
采样性能评估对接计算引擎能否产生正确的pose,将计算引擎产生的pose与蛋白-配体复合物结构中的那个pose比较并计算RMSD(root mean square deviation),如果RMSD小于2.0(Å),那么就认为对接计算成功。因为打分最高的那个pose(the top score pose)不一定是对接最佳的那个(the best pose),因此采样性能评估分别考察了打分最高pose的对接成功率与最佳pose的对接成功率。
将打分最高20、10、3、1个pose与复合物晶体结构里的配体相比,考察最小的RMS是否小于等于2Å:是则认为对接计算成功、否则认为失败。
因为1zea、3ag9与3uri等三个复合物结构配体的柔性键数量超出Lead Finder允许的最大值而没有被Lead Finder进行对接计算,所以对其它的192个配体进行性能评估。
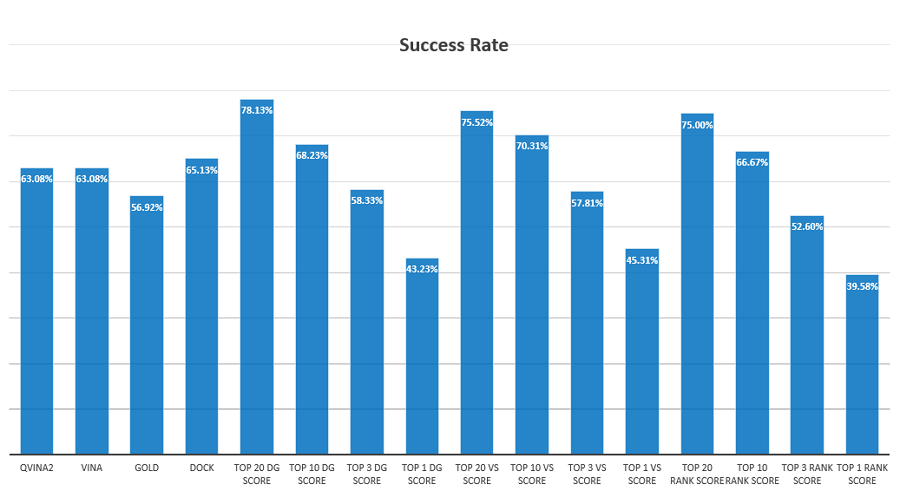
图1. Lead Finder的对接计算成功率
结果如图1所示,在78.1%算例中,dG Score可以在打分最高的20个pose里包含了一个正确的pose;VS score与Rank score也表现出相似的性能,成功率分别为75.5%与75.0%。如果仅考察打分最佳的一个pose,则成功率大大降低:dG score、VS score与Rank score的成功率分别为43.2%、45.3%以及39.5%。所有的对接算法都存在打分最高的pose不是对的pose这个问题,在《十种分子对接软件的性能评估》第3.1节已经有讨论。因此,如何将正确pose从打分最高前的20个里挑出是我们面临的主要挑战。
Alhossary A等人3在开发分子对接软件QuickVina2的时候,用CASF2013研究了QuickVina2、AutoDock Vina、GOLD 5.2、DOCK 6的性能, 结果见图1。显而易见,采用打分靠前的20或10个POSE时,Lead Finder的成功率优于QuickVina,VINA,DOCK6与GOLD 5.2。
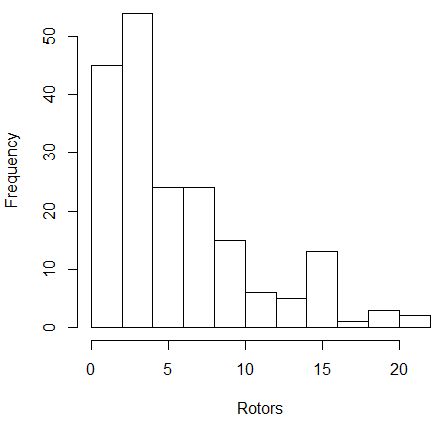
同时,我们考察了柔性键数量与成功率之间的关系:图2(右)是dG Score打分最优20个pose里,最低rms与柔性键数量(Rotors,OpenBabel 2.4计算)的关系。
 |
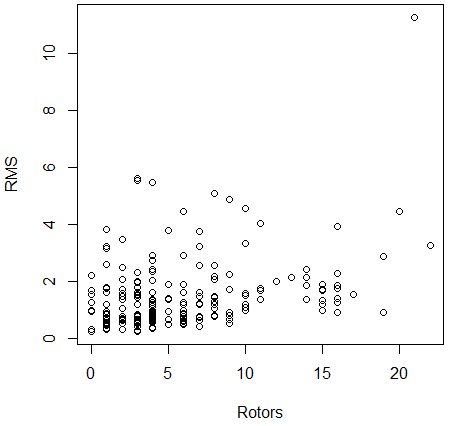 |
图2. 数据集柔性键数量分布以及dG Score打分前20最佳pose的RMS分布
用第三方打分函数提高打分Top 3的成功率
通过对CASF-2013基准测试集的验证,我们发现Lead Finder的对接引擎具有非常强的采样性能:在打分最高的20、10个pose里,富集到正确pose的算例分别占78%与70%。也就是采集打分最高的20个、10个pose,best pose的成功率分别为78%与70%。在Lead Finder采样的基础上,用ChemPLP 打分函数可以提高Lead Finder在Top 1,2,3时识别正确pose的性能,对接性能测试也证实ChemPLP优异的性能(参见:打分函数的对接性能评估部分)。下图S1展示了ChemPLP对Lead Finder生成的pose重新打分后的对接性能。可以发现Top 1,2与3的对接性能大幅提高了。
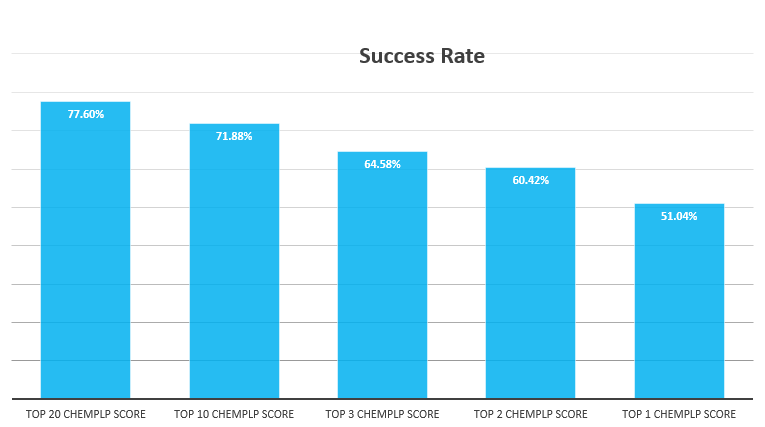
图S1. Lead Finder用ChemPLP打分函数重新打分后的Top 1,2,3,10,20的成功率
从图S1可知,ChemPLP对Lead Finder对接结果重新打分,有51%算例的打分最佳pose是正确的pose。
2. 打分函数的打分性能(Scoring power)评估
打分性能(Scoring power)是指打分函数预测的结合打分与实验值的线性相关性2。采用score only模式,计算蛋白与共晶配体的结合自由能,将Lead Finder三种打分函数dG Score、VS Score与Rank Score的计算值与实验值进行比较,评估线性相关性。
Lead Finder完成了对195个复合物的计算,194个复合物获得计算结果并用于统计处理。Lead Finder的三种打分函数dG Score,VS Score与Rank Score的打分值散点图分别见图3、4、5,可以让我们有个直观的印象:直觉是dG Score的打分值与实验值有明显的相关性,而VS Score与Rank Score的散点图更加离散。
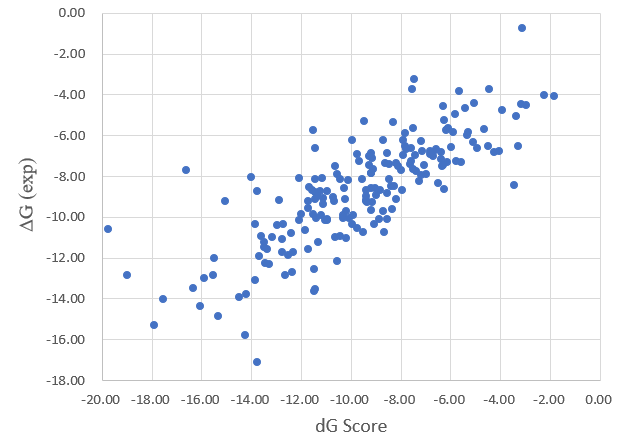
图3. dG Score VS 实验值(R2=0.4334)
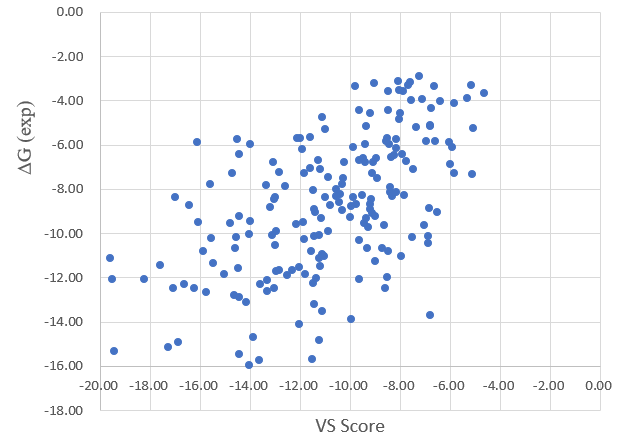
图4. VS Score VS 实验值(R2=0.3146)
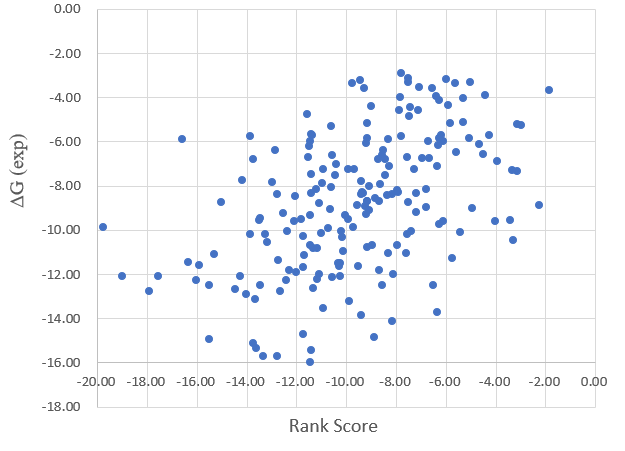
图5. Rank Score VS 实验值(R2=0.23)
Lead Finder三种打分函数dG、VS与Rank Score打分值与实验值的线性回归系数R2与Pearson相关性系数分别见下表1。
Table 1. Lead Finder不同打分函数计算值与实验值的线性回归系数与Pearson相关性系数
| Items | N | R2 | Pearson相关性系数 |
|---|---|---|---|
| dG Score | 194 | 0.4334 | 0.660 |
| VS Score | 194 | 0.3146 | 0.561 |
| Rank Score | 194 | 0.23 | 0.480 |
如表1所示,三个打分函数的预测值与实验值的线性回归系数R2都不高,但是从图3的dG score VS 实验值散点图看着有相关性,实际上Pearson相关性系数为0.66,证实确实具有强相关。Lead Finder的dG Score打分函数为预测结合亲和力而设计,其Pearson系数为0.66也要优于用样数据集测试的其它软件2,见表2。
Table 2. 不同打分函数计算值与实验值的Pearson相关性系数比较,除了dG、VS与Rank score外,均引用自Li Y(2014)2
| Docking Score | N | Pearson相关性系数 |
|---|---|---|
| dG Score | 194 | 0.660 |
| X-Score | 195 | 0.614 |
| ΔSAS | 195 | 0.606 |
| ChemScore@SYBYL | 195 | 0.592 |
| ChemPLP@GOLD | 195 | 0.579 |
| PLP1@DS | 195 | 0.568 |
| VS Score | 194 | 0.561 |
| G-Score@SYBYL | 195 | 0.558 |
| ASP@GOLD | 195 | 0.556 |
| ASE@MOE | 195 | 0.544 |
| ChemScore@GOLD | 189 | 0.536 |
| D-Score@SYBYL | 195 | 0.526 |
| Alpha-HB@MOE | 195 | 0.511 |
| LUDI3@DS | 195 | 0.487 |
| GOLDScore@GOLD | 189 | 0.483 |
| Affinity-dG@MOE | 195 | 0.482 |
| Rank Score | 194 | 0.480 |
| LigScore2@DS | 190 | 0.456 |
| GlideScore-SP | 169 | 0.452 |
| Jain@DS | 114 | 0.408 |
| PMF@DS | 194 | 0.364 |
| GlideScore-XP | 164 | 0.277 |
| London-dG@MOE | 195 | 0.242 |
| PMF@SYBYL | 191 | 0.221 |
由表2可知,就CASF2013的测试集而言,Lead Finder的dG Score表现出最好的实验值与计算值相关性,下图6会看着更清楚一点。
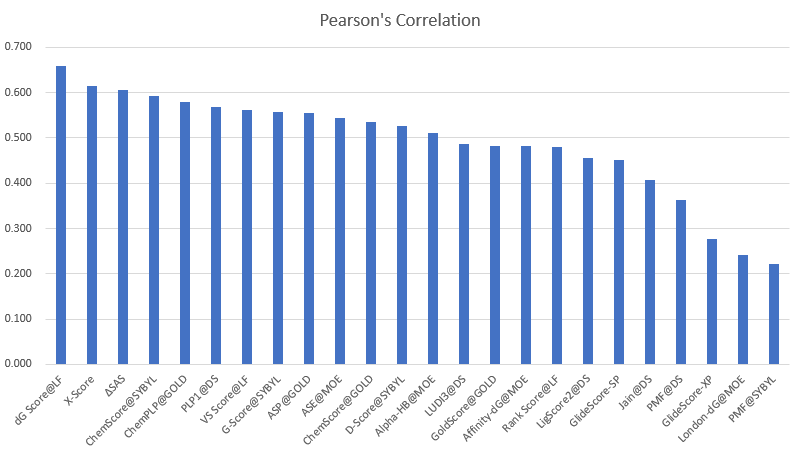
图6. 不同打分函数预测值与实验值Pearson系数比较
在Wang Z(2016)4等人对10种分子对接的比较研究中也考察了打分性能(见:十种分子对接软件的性能评估)。不同于CASF2013方法的是,Wang Z等人用最优的打分值(而不是实验pose的打分值)与实验数据比较。结果表明,10种对接软件的打分值与实验值Pearson系数均小于0.6,这与Li Y(2014)的研究结果2基本一致:计算值与实验值具有强相关的打分函数非常少。X-Score与Lead Finder的dG Score是少有的、具有强相关的打分函数。
3. 打分函数的排序性能(Ranking power)评估
在CASF-2013数据集里,覆盖了65个靶标,每个靶标有3个蛋白-配体复合物结构,这三个配体的结合亲合力覆盖了高中低三个档次。如果打分函数可以正确地区分高中低三档次,则计1分,65个靶标则满分65分。成功率为得分除以65。
Table 3. 不同打分函数的排序能力,除了dG、VS与Rank score外,均引用自Li Y(2014)2
| Docking Score | Success Rate%(High-level) |
|---|---|
| X-Score | 58.5 |
| ChemPLP@GOLD | 58.5 |
| dG Score | 56.9 |
| PLP2@DS | 55.4 |
| GOLDScore@GOLD | 55.4 |
| ΔVINARF206 | 55 |
| VS Score | 53.8 |
| ChemScore@SYBYL | 53.8 |
| Affinity-dG@MOE | 53.8 |
| G-Score@SYBYL | 52.3 |
| Alpha-HB@MOE | 52.3 |
| LUDI1@DS | 52.3 |
| LigScore1@DS | 52.3 |
| ΔSAS | 49.2 |
| D-Score@SYBYL | 49.2 |
| AutoDock VINA6 | 49 |
| ASP@GOLD | 47.7 |
| ChemScore@GOLD | 46.2 |
| Rank Score | 43.1 |
| GlideScore-SP | 43.1 |
| PMF@DS | 43.1 |
| London-dG@MOE | 43.1 |
| PMF@SYBYL | 43.1 |
| Jain@DS | 41.5 |
| ASE@MOE | 40.0 |
| GlideScore-XP | 35.4 |
如表3所示,高级排序能力X-Score与ChemPLP@GOLD并列第一(成功率58.5%);dG Score排名第二(成功率56.9%); 接着是PLP2@DS与GOLDScore@GOLD并列第三(成功率55.4%)。
4. 打分函数的对接性能(Docking power)评估
打分函数的对接性能(Docking power)是指打分函数将正确pose从decoy pose里识别出来的能力,结果见下表4。在CASF-2013的基础上,我们还计算了Lead Finder的dG score,VS score与rank score以及PLANTS的ChemPLP score5。我们发现,Lead Finder的打分函数识别正确pose的性能较差,而ChemPLP对接性能优于绝大部分打分函数。将ChemPLP与Lead Finder组合,可以大幅提高Lead Finder在top 1、2、3 score的虚拟筛选性能。
Table 4. 不同分子对接软件打分函数的对接性能(点击表头可排序)
四. 文献
- Li, Y.; Liu, Z.; Li, J.; Han, L.; Liu, J.; Zhao, Z.; Wang, R. Comparative Assessment of Scoring Functions on an Updated Benchmark: 1. Compilation of the Test Set. J. Chem. Inf. Model. 2014, 54 (6), 1700–1716.
- Li, Y.; Han, L.; Liu, Z.; Wang, R. Comparative Assessment of Scoring Functions on an Updated Benchmark: 2. Evaluation Methods and General Results. J. Chem. Inf. Model. 2014, 54 (6), 1717–1736.
- Alhossary, A.; Handoko, S. D.; Mu, Y.; Kwoh, C. K. Fast, Accurate, and Reliable Molecular Docking with QuickVina 2. Bioinformatics 2015, 31 (13), 2214–2216.
- Wang, Z.; Sun, H.; Yao, X.; Li, D.; Xu, L.; Li, Y.; Tian, S.; Hou, T. Comprehensive Evaluation of Ten Docking Programs on a Diverse Set of Protein–ligand Complexes: The Prediction Accuracy of Sampling Power and Scoring Power. Phys. Chem. Chem. Phys. 2016, 18 (18), 12964–12975.
- Korb, O.; Stützle, T.; Exner, T. E. Empirical Scoring Functions for Advanced Protein-Ligand Docking with PLANTS. J. Chem. Inf. Model. 2009, 49 (1), 84–96.
- Wang C, Zhang Y. Improving scoring-docking-screening powers of protein–ligand scoring functions using random forest. J Comput Chem. 2017;38(3):169-177. doi:10.1002/jcc.24667.



