用系综虚拟筛选策略提高基于药效团模型的虚拟筛选性能——PDE5A抑制剂虚拟筛选案例分析
摘要:人们已经证明系综虚拟筛选策略可以提高基于配体与基于分子对接虚拟筛选方法的性能,但能否提高基于药效团虚拟筛选方法的性却没有文献报道。本文以DUDE+的PDE5A数据集为例,比较了使用单一药效团模型的虚拟筛选与使用五个药效团模型的系综虚拟筛选在性能上的差异。结果表明,系综虚拟筛选不仅以AUC为代表的总体性能上,而且在以ROC AUC、BEDROC以及富集因子(Eenrichment Factor,EF)、富集比(Enrichment ratio 1%, ER1%)为代表的早期富集能力上都优于单一药效团模型虚拟筛选。药效团系综虚拟筛选与流行分子对接方法进行的单一蛋白结构虚拟筛选相比,在代表总体性能的AUC指标上优于AutoDock Vina,与DOCK相当,而位于Glide与Surflex之后;但是在代表早期富集能力的BEDROC、EF上远超GOLD、GLIDE、Surflex-Dock、FlexX、AutoDock与Dock等流行的分子对接软件。总的来说,系综药效团虚拟筛选不仅在AUC上与单一蛋白的对接虚拟筛选方法上具有可比性,而且在早期富集能力方面远优于分子对接方法。
1. 前言
用同一靶标多不同形状结合位点的代表性蛋白结构对数据库进行虚拟筛选(virtual Screen,VS)是提高虚拟筛选性能的常用策略。这一策略也称为ensemble VS。Jain课题组[1]在DUDE数据集基础上建立了DUDE+数据集,每个靶标至少5个蛋白-配体复合物结构,证明了无论是基于结构还是基于配体的方法,使用多个模型进行虚拟筛选要比使用单一模型进行虚拟筛选具有更好的性能表现。
2. 方法与材料
2.1 药效团模型的准备
使用DUDE+的5个PBD结构1UDT、1UDU、3JWR、3SHZ、3TGE用Ligandscout基于结构的方法忽略水分子进行创建。
2.2 化合物数据库的准备
用Ligandscout的idbgen在Best模式下对从DUDE下载的PDE5A的actives与decoys数据集(SDF格式)进行构象搜索。
2.3 药效团虚拟筛选
采用Ligandscout的iscreen在Best模式下进行默认参数的虚拟筛选,虚拟筛选用绝对拟合值作为打分函数。
2.4 性能评估
鉴于DUDE下载的actives与decoys数据集因为互变异构体与质子化状态的枚举,导致一个化合物会以不同的形式多次出现、并可能被虚拟筛选多次命中,因此对虚拟筛选结果按化合物名称进行去重,同名化合物仅以其中打分最高那个来代表。所有的性能评估指标都在去重的基础上进行计算。
需要注意的是药效团虚拟筛选的打分不同于分子对接与分子形状,药效团虚拟筛选通常要么与药效团匹配、要么不匹配。不匹配的化合物通常不被保存下来,为了进行性能评估,将不匹配的化合物(没有命中的化合物)人为地给予一个很低的打分值0。
用AUC、logAUC、三种α参数的BEDROC、富集因子等指标来评估虚拟筛选的性能。它们的计算方法见前文《如何进行虚拟筛选的方法学验证》[3]。
3. 结果
3.1 药效团的准备
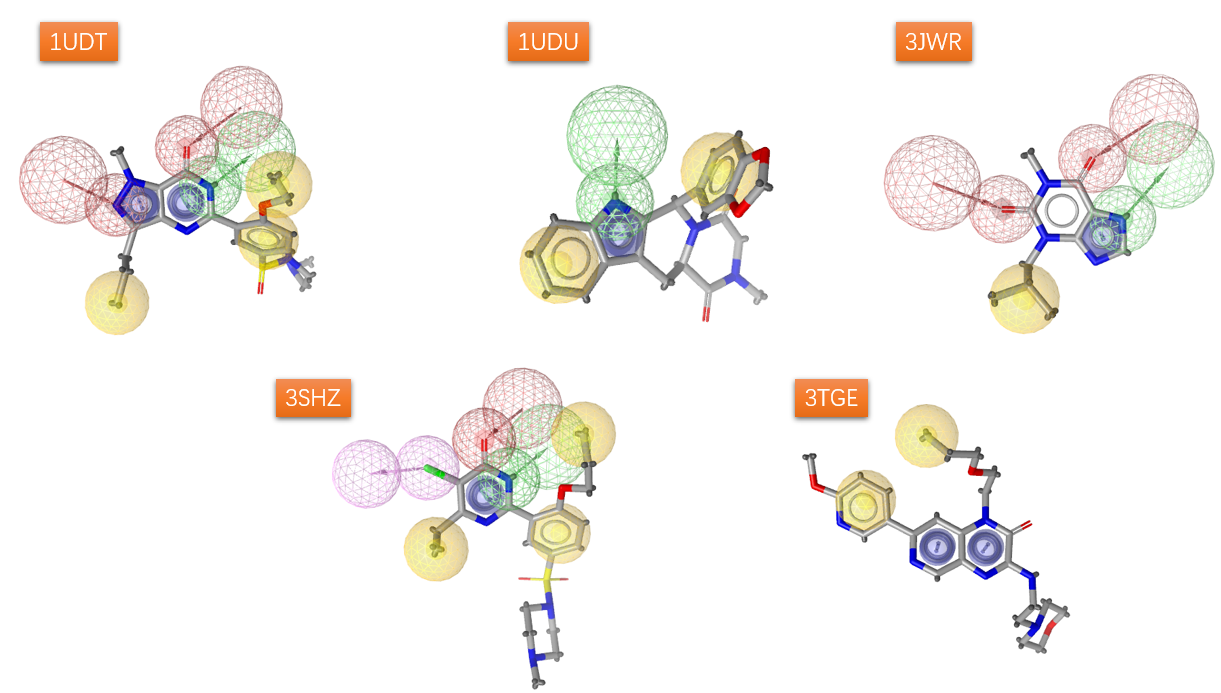
根据PDB代码下载复合物结构,去除水分子,采用默认的参数生成5个药效团模型,如图1所示。
图1. 5个初始的药效团模型
很显然5个模型各不相同。虽然默认参数生成的模型很多时候相当好,但是经常总有很大的优化空间。观察1UDT与3HSZ的模型,他们之间有很多的共同点,用Ligandscout的alignment模块,很容易对这两个模型进行叠合比较,并获得他们的公共特征,加以人工分析可能就得到更加贴切的药效团模型,如图2所示。

图2. 1UDT与3HSZ的公共药效团模型
显然1UDT与3HSZ公共药效团特征更能说明结合特征,在实践中也经常这么用,比如De Luca等人[5]就在碳酸酐酶VII型抑制剂的发现中使用了该策略,我们对该文进行了编译与分享[6]。为了兼顾1UDT与3HSZ各自的特点,可以将两个非公共特征设为可选的药效团特征。比如将3HSZ的卤键设为可选的特征,将1UDT的五员环所在芳香环与氢键受体设为可选的特征,如图3所示。
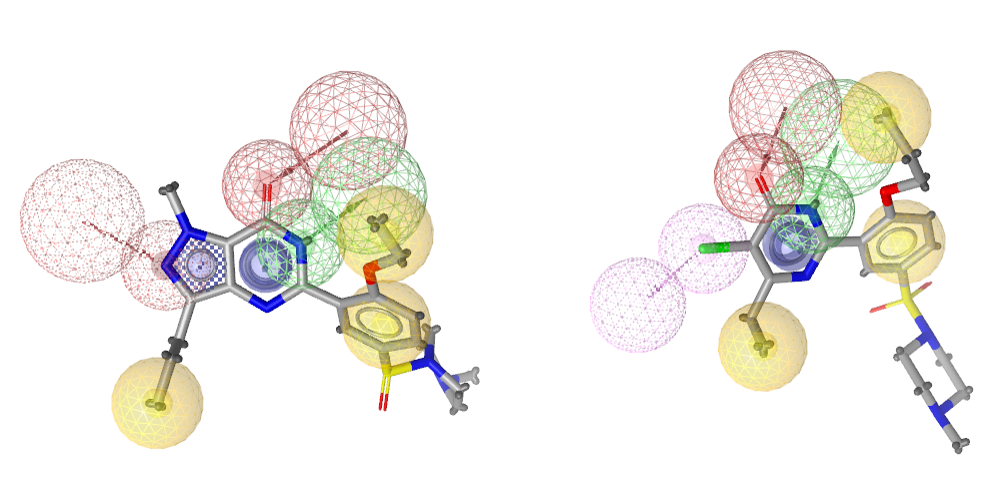
图3. 在1UDT与3HSZ药效团模型中使用可选特征
比之单纯是用公共特征的药效团模型,加了可选特征的模型更具灵活性。如果一个特征不匹配不会影响活性,但是会增强活性,那么将这个特征设为可选特征既有机会筛选到满足模型的化合物,还可能筛选到活性增强的化合物。图3给出了以1UDT与3HSZ公共药效团特征为基础的增强药效团模型。
此外,ligandscout默认的相互作用模式非常严格,比如氢键、芳香堆积、卤键相互作用等很多相互作用具有距离与角度约束,为了不同方法之间的比较,有时候有必要对相互作用参数进行调整以便能够得到相同的药效团模型。如图4所示,偏好设置非常容易实现参数调整以改变ligandscout自动生成药效团的行为。除了相互作用参数调整之外,还可以通过ligandscout药效团编辑在现有的药效团基础上进行特征添加、删减、权重的加减等操作。以1UDT与3HSZ的苯环为例,默认条件下分析得到疏水中心,但是应要求设为芳香性中心,就是通过添加特征来实现。我在图1中将该苯环显示为疏水中心,而在图2中将其显示为芳香环,当然也可以两者都是,如图4所示。

图4. 芳香堆积参数设置面板与苯环的特征设置。Ligandscout支持一个官能团具有多种特征,比如高亮部分的苯环兼具芳香性与疏水性特征。
最终用于虚拟筛选性能评估的5个模型如图5所示。其中在1UDT五员环部分的氢键受体与芳香环、以及3HSZ卤键被设为可选的药效团特征。此外,1UDT与3HSZ的苯环对应的部分仅使用了芳香环特征(是芳环还是疏水中心,在该例子中影响不大)。
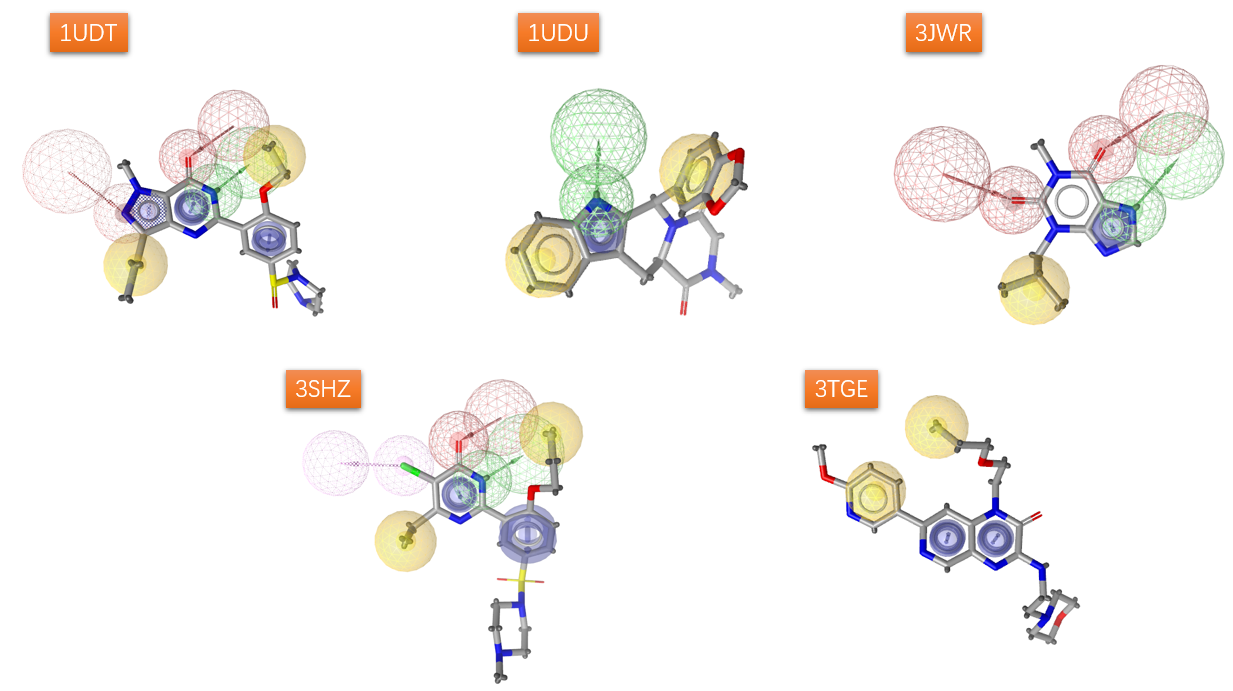
图5. 最终用于虚拟筛选性能评估的模型(Ligandscout药效团模型文件见附件)
在后续的虚拟筛选过程中,如果是ensemble虚拟筛选,则使用or布林运算符,对5个药效团模型同时进行虚拟筛选,将匹配最佳的那个打分作为该化合物的最终打分。
3.2 化合物数据库的准备
首先计算DUDE PDE5A的actives与decoys数据集4的化合物数量:将化合物名称提取出来,去掉重名的,计算数量。
1 2 | (rdkit2020) [gkxiao@master pde5a]$ gettitles.py actives_final.sdf|sort|uniq|wc -l 398 |
由此可见,actives数据集的化合物数为398,这与DUDE给出的数据[4]完全一致。
1 2 | (rdkit2020) [gkxiao@master pde5a]$ gettitles.py decoys_final.sdf|sort|uniq|wc -l 27521 |
下载到的decoys数据集含有不重复的化合物数为27521,比起DUDE所述的27550个[4]少了29个。
接下来用idbgen在best模式下对化合物进行构象搜索,得到ldb格式的多构象数据库。
actives数据集的准备:
1 2 3 4 | idbgen -i actives_final.sdf \ -o actives.ldb \ -t icon-best \ -C 32 -A -l actives_idbgen.log |
decoys数据集的准备:
1 2 3 4 | idbgen -i decoys_final.sdf \ -o decoys.ldb \ -t icon-best \ -C 32 -A -l decoys_idbgen.log |
3.3 虚拟筛选
下面以5个药效团的ensemble VS为例,来说明虚拟筛选流程;单一药效团模型虚拟筛选流程与此一致。首先,我们准备个文件ensemble_vs.list,内容如下:
1 2 3 4 5 | 1udt.pml 1udu.pml 3jwr.pml 3shz.pml 3tge.pml |
每行一个药效团模型,因此,这个文件包含了5个之前准备好的药效团模型的名称(包含路径)。默认情况下,它们之间的关系是or,当然还可以and与not。因为我们要进行ensemble VS,所以用or。
对阳性数据集进行虚拟筛选:
1 | iscreen -q ensemble_vs.list -d actives.ldb -o actives_vs.sdf -C 24 -l actives_vs.log -s BEST |
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 | 13:50:15 Using database 'actives.ldb' with 706 compounds.
13:50:15 Configuration:
Database: /public/gkxiao/work/dude/pde5a/actives.ldb (706
molecules)
Query file (1): ensemble_vs.list
Hit file: actives_vs.sdf
Check exclusion volumes: true
Max. omitted features: 0
Use first matching conf: false
Distribution mode: Use 24 CPUs on this system
Heap size: 42968 MB
13:53:29 299 virtual hits found.
13:53:29 Overall execution time: 193.55 seconds.
13:53:29
13:53:29 iscreen finished successfully. |
总共筛选了706个化合物,命中299个未去重的阳性化合物。
对阴性数据集进行虚拟筛选:
1 | iscreen -q ensemble_vs.list -d decoys.ldb -o decoys_vs.sdf -C 24 -l decoys_vs.log -s BEST |
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 | 13:53:36 Using database 'decoys.ldb' with 27826 compounds.
13:53:36 Configuration:
Database: /public/gkxiao/work/dude/pde5a/decoys.ldb (27826
molecules)
Query file (1): ensemble_vs.list
Hit file: decoys_vs.sdf
Check exclusion volumes: true
Max. omitted features: 0
Use first matching conf: false
Distribution mode: Use 24 CPUs on this system
Heap size: 42968 MB
15:59:38 651 virtual hits found.
15:59:38 Overall execution time: 7562.53 seconds.
15:59:38
15:59:38 iscreen finished successfully. |
总共筛选了27826个化合物,命中651个未去重的阴性化合物。
ensemble_vs.list包含了5个药效团模型,阳性与阴性数据集具有相似的虚拟筛选速度,分别为平均3.65与3.68个化合物/秒。
3.4 合并虚拟筛选结果
首先对命中化合物SDF文件的Active/Decoy标签进行修改,确保阳性化合物Active/Decoy标记为active,阴性化合物标记为decoy:
1 2 3 4 5 6 | sed s/unknown/active/g actives_vs.sdf >> act_tmp.sdf mv act_tmp.sdf actives_vs.sdf sed s/unknown/decoy/g decoys_vs.sdf >> decoy_tmp.sdf mv decoy_tmp.sdf decoys_vs.sdf #合并 cat actives_vs.sdf decoys_vs.sdf >> hits.sdf |
3.5 去重复结构
因为DUDE数据集在准备时进行了异构体枚举(包括质子化状态与互变异构体枚举),在进行性能评估前需要将重复出化合物进行去重复。用python的聚合函数对进行去重:同名化合物仅保留打分最高的一个。
1 | sdf2uniqsdf.py hits.sdf hits_nodup.sdf |
将去重、聚合后的文件保存为hits_nodup.sdf。
3.6 用irocplotter绘制ROC曲线
用irocplotter绘制ROC曲线,键入如下命令:
1 2 3 4 5 | (rdkit2020) [gkxiao@master pde5a]$ /public/gkxiao/software/ligandscout4/irocplotter \ -a 398\ -d 27521\ -i hits_nodup.sdf \ -o multi_vs_roc.png |
其中-a参数为actives数据集的化合物数;其中-d参数为decoy数据集的化合物数;-i参数为虚拟筛选命中的化合物,由actives与decoys命中化合物合并、去重而来;-o参数为输出文件名称,默认生成png格式图片。键入命令后出现如下提示:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 | _ _ _
_ __ ___ ___ _ __ | | ___ | |_ | |_ ___ _ __
| '__| / _ \ / __| _____ | '_ \ | | / _ \ | __|| __| / _ \| '__|
| | | (_) || (__ |_____|| |_) || || (_) || |_ | |_ | __/| |
|_| \___/ \___| | .__/ |_| \___/ \__| \__| \___||_|
|_|
roc-plotter - LigandScout V4.4.8
Licensed to: Gaokeng Xiao, Guangzhou Molcalx Ltd (until 2023-01-01).
Actives: 398
Decoys: 27521
Hitlist: hits_nodup.sdf
Output: multi_vs_roc.png
Image-size: 600x600
Reading data (2/4) [=========================================] 100.000%
Reading data (4/4) [=========================================] 100.000%
Creating ROC-curve [=========================================] 100.000%
roc-plotter finished successfully. |
irocplotter默认保存的图片分辨率为600×600,结果如图6所示。
图6. irocplotter绘制的PDE5A系综虚拟筛选ROC曲线
3.7 用sklearn来绘制ROC曲线
为了与irocplotter进行比较,用前文[3]描述的方法通过python的sklearn库来绘制ROC曲线,结果如图7所示。
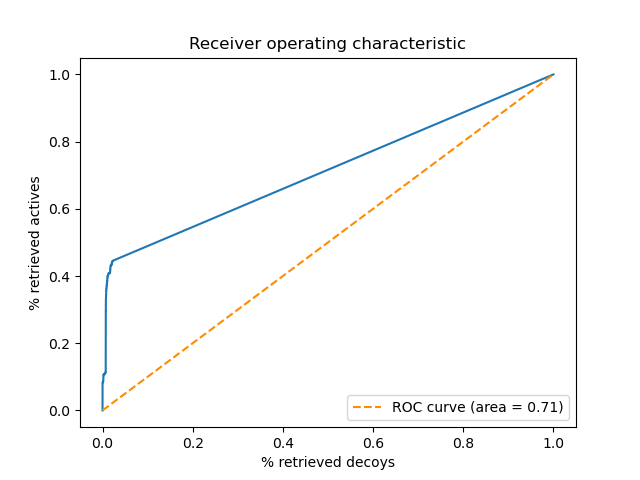
图7. python sklearn库绘制的PDE5A系综虚拟筛选ROC曲线
比较python sklearn与irocplotter绘制的ROC曲线,两者基本一致。无论哪一张曲线,都可以看到在假阳性很低的时候,曲线就非常靠近纵坐标,这是早期富集能力好的表现。
3.8 主要的虚拟筛选性能指标
用前文[3]描述的方法进一步计算AUC,logAUC[0.001,1],BEDROC,EF等虚拟筛选性能参数,结果如表1所示。
表1、单一药效团模型与ensemble药效团模型虚拟筛选性能比较
| 指标 | 1udt | ensemble |
|---|---|---|
| 命中阳性化合物数 | 25 | 178 |
| 命中阴性化合物数 | 1 | 649 |
| AUC | 0.53 | 0.71 |
| CI 95%(1000 resample) | 0.5189 – 0.5428 | 0.6902 – 0.7385 |
| ER1% | 100 | 38 |
| logAUCλ=0.1% | 53.6 | 49.0 |
| adjusted-logAUCλ=0.1% | 38.1 | 34.5 |
| BEDROC α=321.9 | 0.25 | 0.40 |
| BEDROC α=80.5 | 0.10 | 0.38 |
| BEDROC α=20.0 | 0.07 | 0.42 |
| EF0.5% | 12.5 | 21.5 |
| EF1% | 6.3 | 21.0 |
| EF5% | 1.3 | 8.9 |
| EF10% | 0.6 | 4.5 |
从虚拟筛选的总体性能上看,很明显,比之单一药效团模型的虚拟筛选、ensemble药效团模型虚拟筛选的ROC AUC提高了0.18:即从0.53提高到0.71。而后者的性能可以与单一蛋白分子对接虚拟筛选的性能相当。如图8所示,ensemble VS(LS4-ensemble)的AUC优于流行的分子对接软件AutoDock Vina 1.2(AUC=0.68)[7],与DOCK(AUC=0.72)[1]性能相当,但落后于GLIDE(AUC=0.86)与Surflex(AUC=0.76)[1]。AUC的改善得益于不同结合模式的化合物被多样的药效团模型所覆盖,这也验证了ensemble策略的有效性。
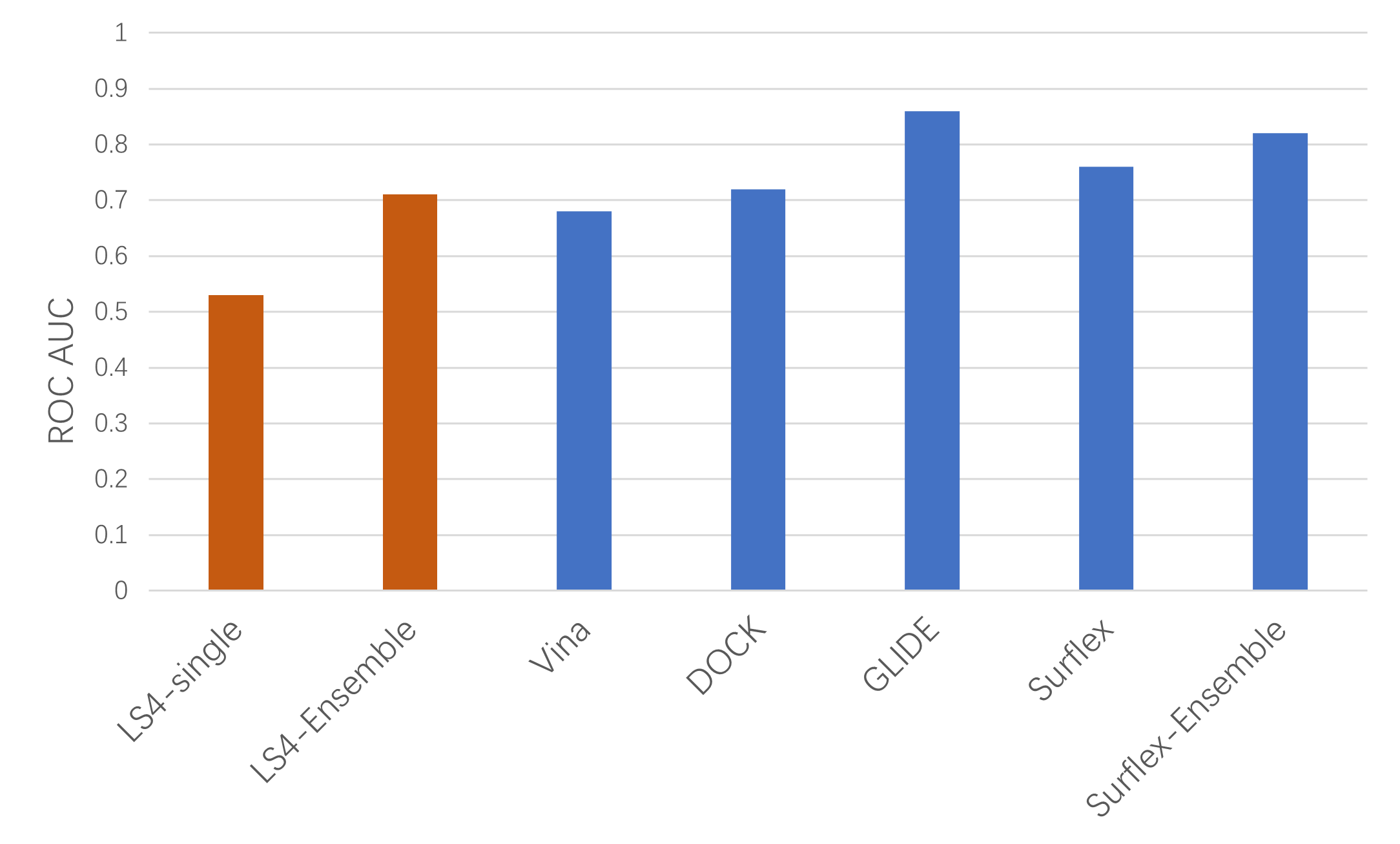
图8. 药效团虚拟筛选与分子对接虚拟筛选总体性能ROC AUC的比较
从早期富集能力上看,比之单一药效团模型的虚拟筛选,ensemble虚拟筛选也表现出更好的性能。除了logAUC之外,ensemble虚拟筛选在不同α参数(321.9,80.5,20.0)的BEDROC或EF指标上均大幅优于单一药效团模型的虚拟筛选。
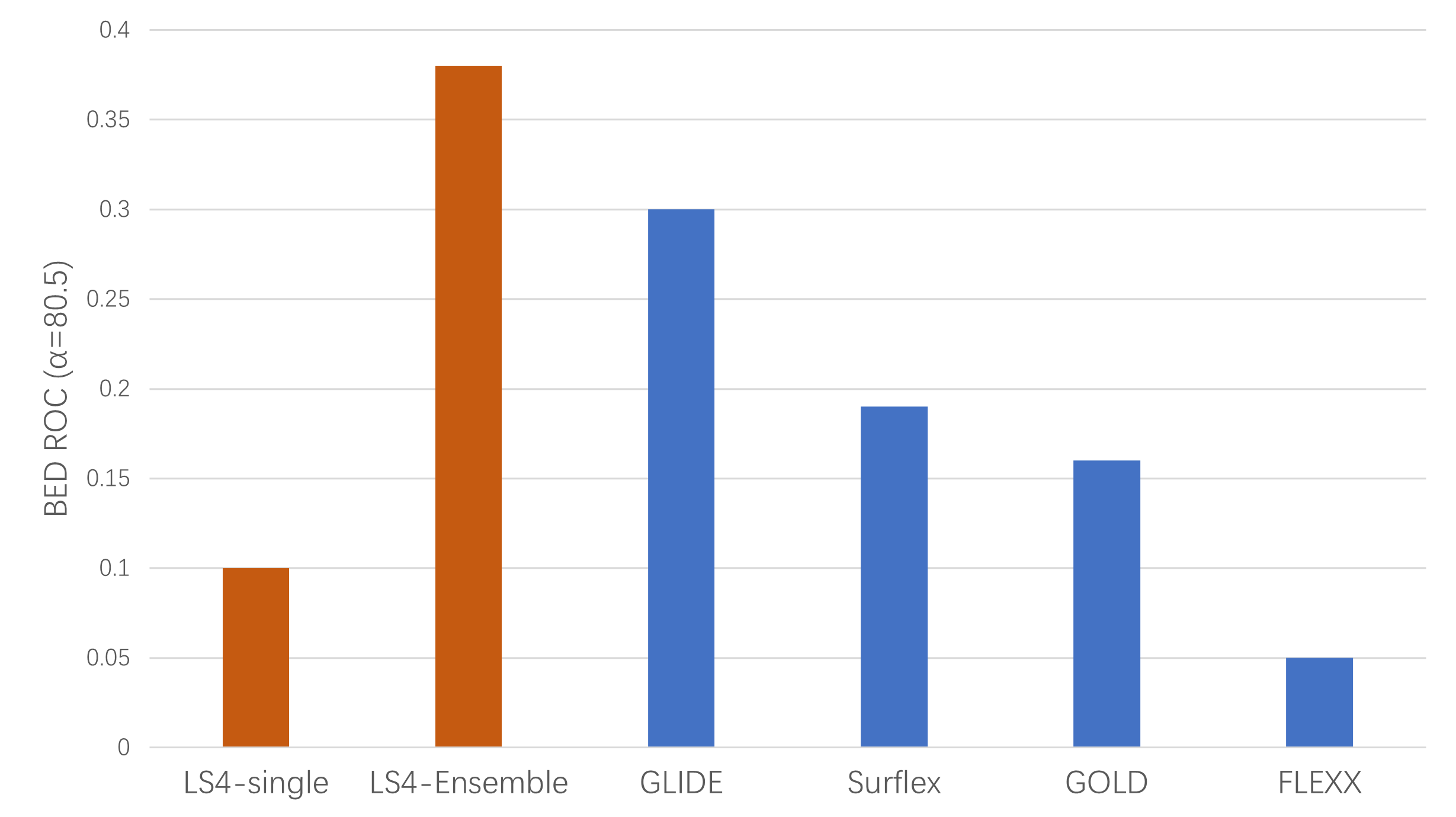
图9. 药效团虚拟筛选与分子对接虚拟筛选以BEDROC(α=80.5)表征的早期富集能力比较
如图9所示,从BEDROC表征的早期富集能力上看,药效团系综虚拟筛选的BEDROCα=80.5=0.38,优于采用单一蛋白进行的分子对接虚拟筛选。根据Chaput等人[9]的研究,GOLD、GLIDE、Surflex-Dock与FlexX等方法的BEDROCα=80.5分别为0.16、0.3、0.19与0.05。这说明在早期富集率上,Ligandscout基于药效团的系综虚拟筛选性能要远远的优于这些最流行的单一蛋白的分子对接虚拟筛选方法。

图10. 药效团虚拟筛选与分子对接Vina 1.2虚拟筛选表征早期虚拟筛选富集性能的EF1%,EF5%以及EF10%值比较
根据Eberhardt等人[9]的研究,AutoDock Vina 1.2在PDE5A数据集上的EF1%、EF5%与EF10%值分别为12.25、5.16与3.43,优于单一药效团模型的虚拟筛选性能,但远远落后于5个药效团模型的系综虚拟筛选性能,如图10所示。
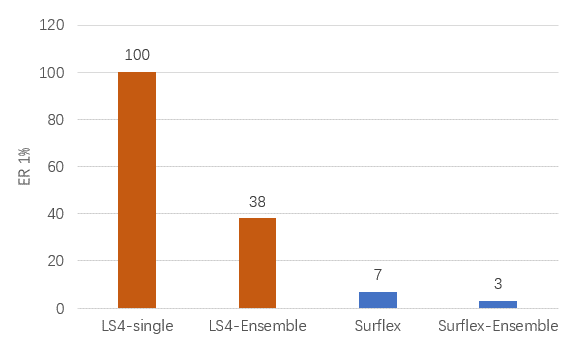
图11. 药效团虚拟筛选与Surflex-Dock分子对接虚拟筛选的ER1%比较
在研究系综对接与单一蛋白对接虚拟筛选性能的时候,Jain课题组[1]使用Enrichment Ratio 1%(ER1%)来评估虚拟筛选方法的早期富集能力。根据作者的研究,该指标与BEDROCα=80.5具有极强的相关性。根据文章提出的计算方法,ER1%可以视为在假阳性率为1%时的真阳性率与假阳性率的百分比值。使用单一药效团模型进行虚拟筛选时,假阳性率为0,因此单一药效团模型虚拟筛选的ER1%为100。如图11所示,无论是单一蛋白结构虚拟筛选,还是ensemble虚拟筛选,基于药效团的方法比Surflex-Dock具有更好的早期富集能力,体现在药效团方法具有更高的ER1%。
4. 总结
人们已经证明系综虚拟筛选策略可以提高基于配体与基于分子对接虚拟筛选方法的性能,但能否提高基于药效团虚拟筛选方法的性能却没有文献报道。本文以基准数据集DUDE+的PDE5A靶标为例,测试了以单一药效模型虚拟筛选与使用5个药效团模型的系综虚拟筛选的性能,并与文献报道的性能数据进行了比较。
以ROC AUC为代表的整体性能而言,系综虚拟筛选比单一模型虚拟筛选的AUC有大幅提高,从即从0.53提高到0.71。系综虚拟筛选还在EF与BEDROC为代表的早期富集能力上全面优于单一模型虚拟筛选。logAUC为评价指标的早期富集能力是个例外,在PDB5A这个例子里,单一模型虚拟筛选的早期富集能力要优于系综虚拟筛选。
在总体的虚拟筛选性能上,与流行的分子对接软件AutoDock Vina、DOCK、Glide与Surllex-Dock相比,单一模型的药效团虚拟筛选方法在ROC AUC上不如分子对接方法;使用了5个模型的系综药效团虚拟筛选方法的性能优于AutoDock Vina,与Dock相当,落后于Glide与Surlfex。
在以BEDROCα=80.5为指标的虚拟筛选早期富集能力上,系综药效团模型虚拟筛选方法的性能大幅领先于Gold、Glide、Surflex与FlexX;在以EF1%、EF5%与EF10%为指标的虚拟筛选早期富集能力上,药效团系综虚拟筛选的性能远超AutoDock Vina。同样,在以ER1%为指标的虚拟筛选早期富集能力上,药效团系综虚拟筛选比Surflex-Dock系综虚拟筛选具有更好的性能。
此外,Ligandscout药效团虚拟筛选具有非常快的计算速度,包含5个药效团的系综虚拟筛选,采用24核心可实现平均每秒3.6个化合物的计算速度,这意味着非常低的计算成本。
总的来说,系综药效团虚拟筛选与主流的分子对接虚拟筛选(单一结合位点)相比,具有相似的总体虚拟筛选性能,但是具有更好的早期富集性能;同时考虑到药效团虚拟筛选的便利与快速,因此强烈推荐尝试使用基于药效团的系综虚拟筛选。
5.附件
百度网盘下载链接:pde5a_materials.tar.gz, 提取码:w65e。目录如下:
1 2 3 4 5 6 7 8 9 10 11 | ├── 1udt.pml ├── 1udu.pml ├── 3jwr.pml ├── 3shz.pml ├── 3tge.pml ├── actives.ldb ├── decoys.ldb ├── ensemble_vs.list ├── single_vs.list ├── to_cleanup.sh ├── to_run_vs.sh |
其中5个pml文件为药效团模型;2个ldb文件为已经准备好可以直接用于Ligandscout虚拟筛选的化合物库。
6.文献
- Cleves, A. E.; Jain, A. N. Structure- and Ligand-Based Virtual Screening on DUD-E + : Performance Dependence on Approximations to the Binding Pocket. J. Chem. Inf. Model. 2020, 60 (9), 4296–4310. https://doi.org/10.1021/acs.jcim.0c00115.
- 肖高铿. Ligandscout教程–布尔运算符(Boolean operator). 2016-08-16. http://blog.molcalx.com.cn/2016/08/06/ligandscout-tutorial-boolean-operator.html
- 肖高铿. 如何进行虚拟筛选的方法学验证.墨灵格的博客. 2016-09-22. http://blog.molcalx.com.cn/2016/09/22/virtual-screening-methodology-validation.html
- DUDE的PDE5a数据集(2022-01-25访问):http://dude.docking.org/targets/pde5a
- De Luca, L.; Ferro, S.; Damiano, F. M.; Supuran, C. T.; Vullo, D.; Chimirri, A.; Gitto, R. Structure-Based Screening for the Discovery of New Carbonic Anhydrase VII Inhibitors. Eur J Med Chem 2013, 71C, 105–111.
- 基于结构的虚拟筛选发现新的碳酸酐酶VII型抑制剂.墨灵格的博客.2019-05-10. http://blog.molcalx.com.cn/2019/05/10/ls-sbdd-hca.html
- Mao, Y.; Gong, B. EPharmer : An Integrated and Graphical Software for Pharmacophore-Based Virtual Screening. J. Comput. Chem. 2021, No. May, 1–15. https://doi.org/10.1002/jcc.26743.
- Eberhardt, J.; Santos-Martins, D.; Tillack, A. F.; Forli, S. AutoDock Vina 1.2.0: New Docking Methods, Expanded Force Field, and Python Bindings. J. Chem. Inf. Model. 2021, acs.jcim.1c00203. https://doi.org/10.1021/acs.jcim.1c00203.
- Chaput, L.; Martinez-Sanz, J.; Saettel, N.; Mouawad, L. Benchmark of Four Popular Virtual Screening Programs: Construction of the Active/Decoy Dataset Remains a Major Determinant of Measured Performance. J. Cheminform. 2016, 8 (1), 56. https://doi.org/10.1186/s13321-016-0167-x