ORION云计算——OEDocking与ROCS超高通量虚拟筛选计算服务
摘要:ORION是OpenEye基于AWS的云计算计算,部署了OpenEye的全部软件工具并整合多种开源软件。ORION以专有技术服务的方式,为国内客户提供基于FRED、HYBRID分子对接与ROCS分子形状技术对药明康德WUXI GALAXI库、Enamine REAL库、Mcule Ultimate库进行超高通量虚拟筛选的计算服务,让大家以最低的成本、最快的速度、最高的精获得非常有价值的结果。
ORION云计算

ORION是OpenEye基于AWS的云计算计算,部署了OpenEye的全部软件工具并整合多种开源软件。ORION以专有技术服务的方式,为国内客户提供基于FRED、HYBRID分子对接与ROCS分子形状技术对药明康德WUXI GALAXI库、Enamine REAL库、Mcule Ultimate库进行超高通量虚拟筛选的计算服务,让大家以最低的成本、最快的速度、最高的精获得非常有价值的结果。
可供选择的计算方法
- 基于配体的虚拟筛选:ROCS/FastROCS(每次筛选可以使用多达10个query)
- 就有结构的虚拟筛选:FRED、HYBRID
可供选择的数据库
- 药明康德化合物库(WUXI GALAXI):11.6亿化合物(包含枚举的立体化学异构体)
- Enamine化合物库(Real):36亿化合物(包含枚举的立体化学异构体)
- Mcule化合物库(Ultimate):1.58亿化合物(包含枚举的立体化学异构体)
服务方式
委托计算,不需要采购软件,不需要在搭建计算环境,让乙方经验丰富的专家帮您完成一切。
交付
交付两个命中化合物列表:
- 打分最高的10000个化合物
- 打分最高的500个骨架(Bemis-Murcko clustering)
更多的化合物数,请与我们另行商议。
成功案例
Beacon Discovery案例
Beacon Discovery利用Orion的GigaDock(FRED)分子对接筛选Enamine Real数据库:
- 受体用已知的活性化合物进行验证
- 定制56个化合物
- 总共合成出56个化合物
- 命中22个化合物,作为苗头化合物
- 化合物最佳的活性高达223nM
- 找到了新的系列化合物
发现全新化学类型的HSP90抑制剂
这是OpenEye内部进行的一个测试。为了验证OpenEye的GigaDock工作流的可靠性,OpenEye的科学家们了个超高通量的虚拟筛选计算实验。在ORION上,用GigaDock工作流的分子对接方法FRED调用AWS spot instances共25000个核心对Enamine Real的14亿化合物(立体化学枚举后共36亿个不同的立体异构体)进行了虚拟筛选。Docking的受体结构是从PDB下载的人类HSP90与配体共晶结构(PDB 1UYG)经Spruce准备而来,用活性化合物进行验证之后,直接用于GigaDock的FRED虚拟筛选。结果:
- 下单定制打分最高的120个化学类型新颖的化合物
- 总共合成出105个化合物
- 活性测试后,命中37个化合物
- 化合物最佳的IC50=4μM
- 找到了新的系列化合物
其中1UYG共晶配体与虚拟筛选命中活性最高的化合物结构如下图1所示。
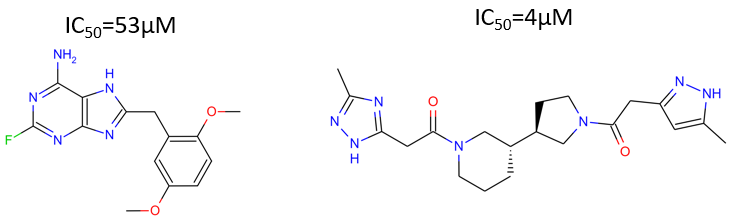
图1. PDB 1UYG共晶结构的配体(左)与FRED虚拟筛选打分最高的化合物之一(右)的化学结构
其中1UYG共晶配体的结合模式如下图2所示。
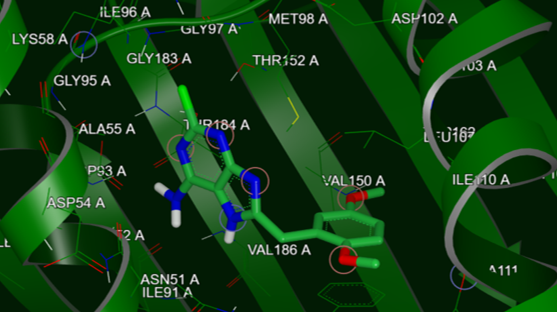
图2. PDB 1UYG共晶结构的配体的结合模式
其中虚拟筛选打分最高化合物的结合模式如下图3所示。

图3. 打分最高化合物的结合模式
总的来说,超高通量分子对接虚拟筛选可以仅根据受体结构发现化学结构新颖的苗头化合物。
相关主题
- 基于配体的虚拟筛选方法ROCS介绍
- 基于结构的虚拟筛选方法FRED与HYBRID介绍
- HYBRID:用多个蛋白信息来提高用单一蛋白结构分子对接虚拟筛选的性能
- Virtual Screening in the Cloud: How Big Is Big Enough?
- Comprehensive Assessment of Torsional Strain in Crystal Structures of Small Molecules and Protein–Ligand Complexes using ab Initio Calculations
Grebner, C.; Malmerberg, E.; Shewmaker, A.; Batista, J.; Nicholls, A.; Sadowski, J. J. Chem. Inf. Model. 2020, 60 (9), 4274–4282.
Rai, B. K.; Sresht, V.; Yang, Q.; Unwalla, R.; Tu, M.; Mathiowetz, A. M.; Bakken, G. A.; K. Rai, B.; Sresht, V.; Yang, Q.; et al. J. Chem. Inf. Model. 2019, 59 (10), 4195–4208.



