摘要:在本算例中,我们以6TAM的共晶配体为先导化合物,用LigandScout基于结构的方法识别药效团并搜索ZINC双环片段数据库获得参考分子库,然后用AutoT&T的分子连接策略对先导化合物的双环母核进行骨架跃迁设计新的分子,结果获得172个新的分子。这些新分子具有新颖的结构、更好的打分,为先导化合物的优化提供了很多新思路,从而有望促进项目的进度。
肖高铿/2021-06-21
1. 前言
之前,我们用Ilib Diverse重现了Bayer公司Mortier等人的KRAS共价抑制剂集中库设计过程,还用Ligandscout对集中库进行了虚拟筛选,发现虚拟筛选可以命中具有新骨架、令人感兴趣的化合物[1]。
实际上,我们还可以用药效团技术进行基于片段的虚拟筛选以发现新的骨架。其要点是,仅用需要被替换的骨架或侧链部分对应的药效团去搜索片段数据库,然后从命中的片段中发现感兴趣的生物等排体。比如,欧莱雅的Deyon-Jung L等人(2016)就用该策略发现新的化妆品苗头化合物,然后经过优化后发现了先导化合物[2]。
基于结构的药效团片段虚拟筛选可以与AutoT&T联合使用:将虚拟筛选命中的片段用AutoT&T的分子优化、生长、连接策略自动设计出新的分子。比如,我们展示过如何联用片段虚拟筛选与AutoT&T进行CDK2抑制剂的设计[3]。
本文将以Bayer公司的一个具有共晶结构(PDB 6TAM)的KRASG12C共价抑制剂(图1)为例,演示如何用片段虚拟筛选联用裁剪与移植策略对其双环母核(图1高亮部分)进行骨架跃迁。
图1.KRASG12C共价抑制剂(PDB:6TAM), 高亮显示的部分为需要被替换的部分
2. 虚拟筛选与新化合物设计流程
2.1 药效团模型
如前文[1]所述,用Ligandscout基于结构的方法从PDB 6TAM生成了药效团模型。除了与图1被替换部分对应的药效团特征保留不变之外,其它的与head group对应的芳环中心(最左边)、与弹头对应的氢键受体以及共价特征都被编辑为可选的(option),以便在虚拟筛选过程中这些特征是可匹配或可不匹配。最终的药效团如图2所示。
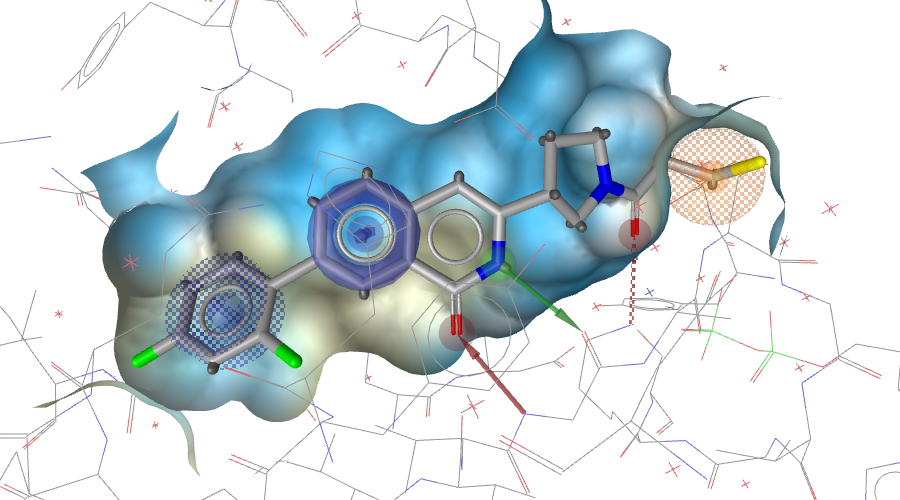
图2.用于片段虚拟筛选的KRASG12C共价抑制剂(PDB:6TAM)药效团模型。其中虚的特征为可选的,实的特征(与被替换双环片段对应)在虚拟筛选过程中是必须匹配的。
2.2 片段化合物库的准备
本次虚拟筛选片段化合物从ZINC15下载,我们仅对其中的双环化合物感兴趣[4]。在ZINC15中,包含了35473个双环化合物。下载保存为SMILES格式,用LigandScout4.4.7/idbgen的iCon以Best模式进行数据库准备,结果保存为LigandScout的ldb格式。
2.3 虚拟筛选
在LigandScout 4.4.7的Virtual Screen里,读入2.1的药效团模型与2.2准备的数据库,执行药效团虚拟筛选(使用get the best matching conformation模式),结果命中3216个片段。
对命中的片段进一步用Affinity score(Hyde-score风格)打分,仅保留打分值(Binding affinity score)小于-15kJ/mol的化合物,总共得到246个化合物。进行该打分主要考虑到两个方面。一方面是我们不需要那么多片段进行测试;另一方面,LigandScout的Binding affinity score是Hyde score的一种实现,根据Reulecke等人(2008)研究[5],对于片段binder来说,打分值应该要优于-15~-25kJ/mol。因此取优于-15kJ/mol的化合物片段具有更高的概率为真binder。
浏览命中的片段样化合物,我们会发现很多有意思的片段。以其中的Ring_103为例(结构见图3上右),它重现了共晶配体双环母核与蛋白的结合模式(图3上左)。
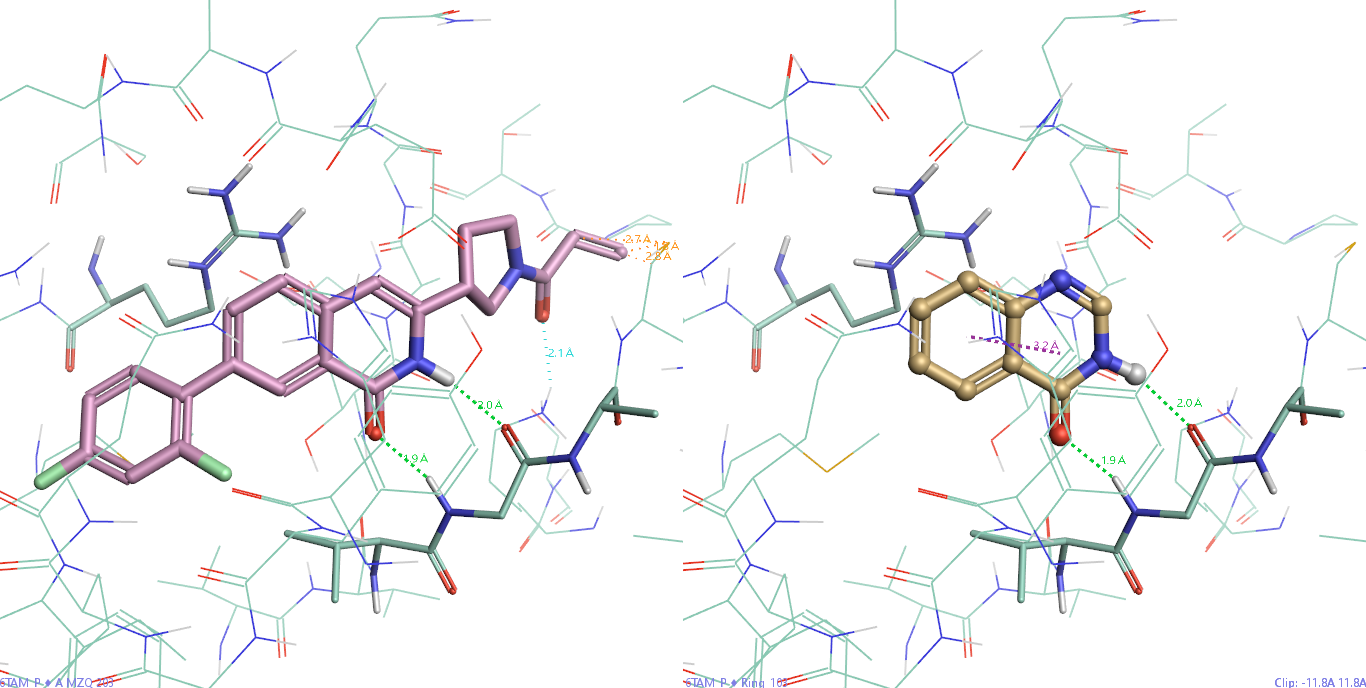
图3. PDB 6TAM共晶配体与命中片段的结合模式
进一步会发现,命中的片段Ring_103在结合位点里与共晶配体的待替换部分完美的叠合,如图4所示。
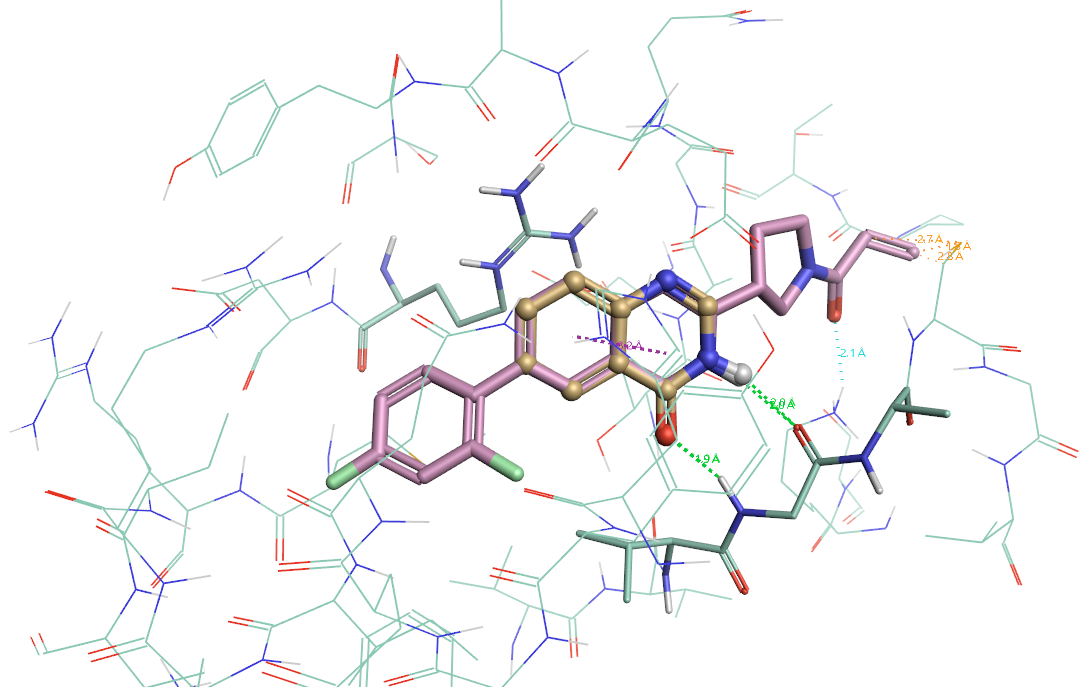
图4. PDB 6TAM共晶配体与命中片段样化合物的叠合。棍棒状配体:共晶配体;球棍状配体:命中的Ring_103片段样化合物。
2.4 人工检视结果,进行裁剪与移植设计新化合物
如图3、4所示,对片段虚拟筛选结果的人工浏览,会自然而然产生新的设计思路:将共晶配体的4,6-二氟苯基与作为连接臂五元环-丙烯酰共价弹头裁剪下来、再移植到感兴趣的新片段上。
手工检查结合模式、然后进行裁剪、移植构建新化合物并不难,比如用LigandScout或Flare在蛋白结合位点里很容易进行此类分子设计[6]。大量的这类操作过程却很繁琐、费时且效率低下,因此自动实现裁剪、移植是非常重要且必要的。
2.5 AutoT&T:自动裁剪与移植生成新化合物
AutoT&T软件是高效的药物分子从头设计软件[7,8,9],主要由剪裁和移植模块以及后续处理模块组成(见图 5)。其中剪裁和移植模块用于分析靶标蛋白、先导化合物和参考分子库。通过化学键匹配的方法找出参考分子库中可以用于移植的结构片段,将其中亲合性预测值高于先导化合物上相应子结构的片段嫁接到先导化合物上,从而形成新的分子。在剪裁和移植过程中,用户可以指定先导化合物上的一根或多根化学键作为优化位点,此时AutoT&T软件将根据用户指定的优化位点在参考分子库中查找可以与之相互匹配的化学键,然后根据这些化学键对相应的分子进行结构剪裁,最后将结构剪裁得到的合适的结构片段移植到先导化合物上,从而得到新的结构优化的分子。此外,AutoT&T软件也可以根据参考分子库与先导化合物上化学键的匹配情况自动选择合适的优化位点,从而将参考分子库中与靶标蛋白结合较好的部分移植到先导化合物结构中。
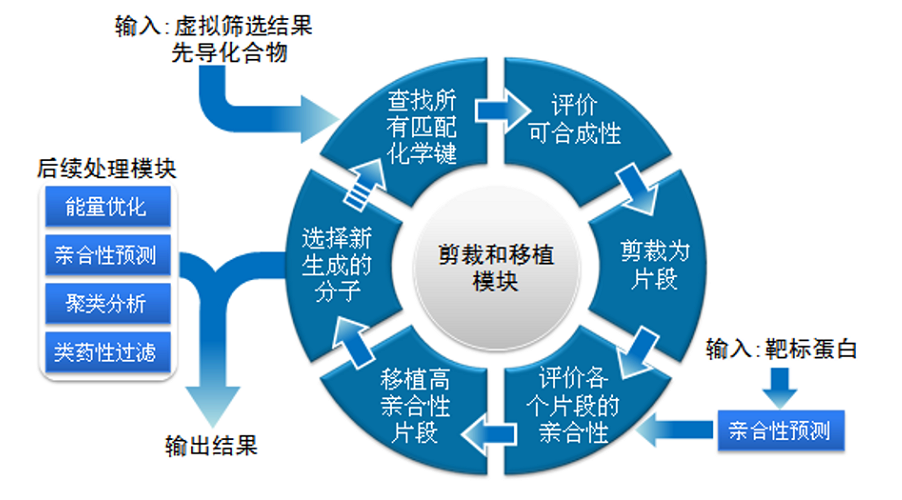
图5. AutoT&T的整体结构设计
如图6所示,在算例中需要进行骨架跃迁化合物为PDB 6TAM的共晶配体,在母核与两个侧链之间的(11,10)与(18,3)两个化学键定义为优化位点,其中10号与3号原子所在那一侧的片段将被替换。AutoT&T的LinkLeadOpt程序用来实现这个母核替换过程。

图6. PDB 6TAM共晶配体及其优化位点
LinkLeadOpt是AutoT&T实现片段连接策略的工具,命令如下:
1 2 3 4 5 6 | LinkLeadOpt -l reference.sdf \ -vs aromatic-ring-hits.sdf \ -p 6tam_C12G.pdb \ -out att-new-design.sdf \ -c '(11,10) (18,3)' \ -a 20 |
其中,参数-l读入先导化合物(本例中为PDB 6TAM的共晶配体,被保存为reference.sdf); 参数-vs读入参考分子库,也就是在2.3节命中的片段(aromatic-ring-hits.sdf); 参数-p读入共晶结构的蛋白(PDB 6TAM);参数-c指定了优化位点,若剪切下来的片段在打分上优于第10与3序号原子所在的片段,则进行片段替换;参数-out用来指定输出文件的名称;参数-a控制键匹配的角度范围,默认为15°。
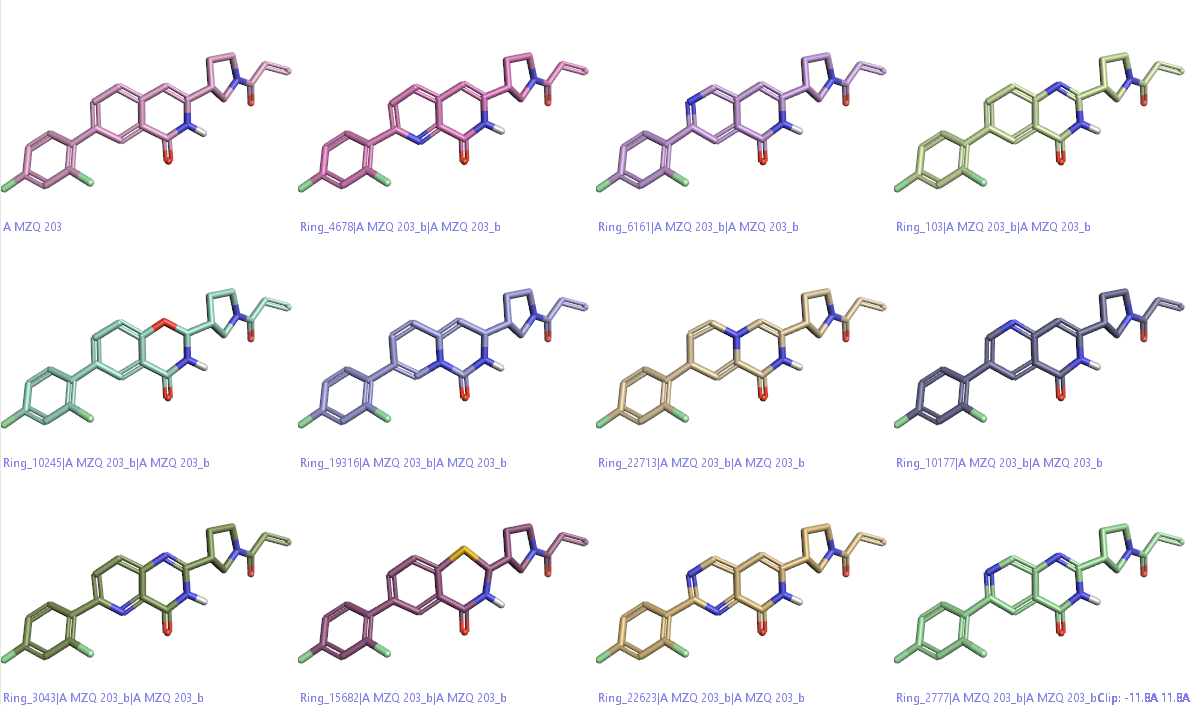
在本算例中,LinkLeadOpt生成了11个新母核分子,结构如下图7所示。
图7. LinkLeadOpt在本次计算中给出的11个新分子,其中第一个(左上角)为先到化合物(6TAM的共晶配体)
AutoT&T用片段的来源来给新分子命名,图7第1行第2个分子的名称为Ring_25291|A MZQ 203_b|A MZQ 203_b,这说明新母核来自名称为Ring_25292的片段,其结构如下图8。
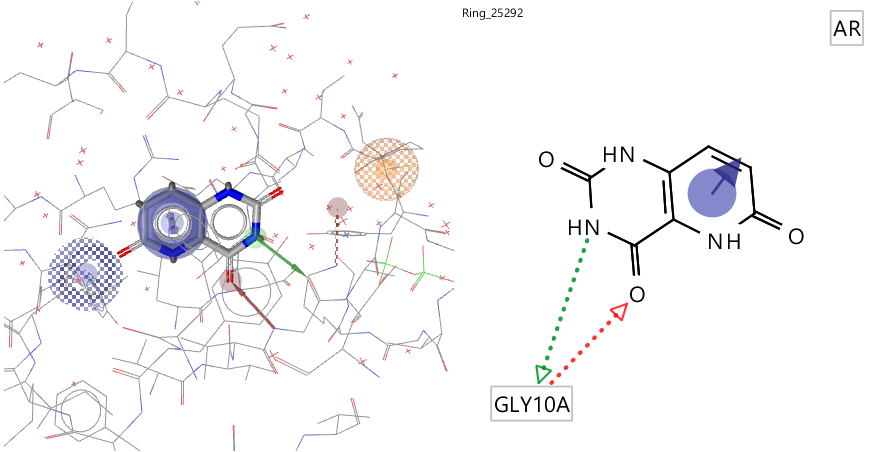
图8. Ring_25292的2D结构及其与药效团的叠合
Ring_25292的两个羰基取向与先导化合物的(11,10) (18,3)键取向一致(实际上是重合的),Ring_25292的双环被剪切、并移植到先导化合物上。
在新设计的11个化合物里,并没有见到Ring_103之类匹配很好的片段(见图3、4),这可能有两个原因:键不匹配或打分不如先导化合物里原有的双环片段。我们用-ih参数,以确保参考数据库中化合物的C-H键也参与键的匹配:
1 2 3 4 5 6 | LinkLeadOpt -l reference.sdf \ -vs aromatic-ring-hits.sdf \ -p 6tam_C12G.pdb \ -out att-new-design.sdf \ -c '(11,10) (18,3)' \ -ih |
现在,LinkLeadOpt生成了172个新化合物。部分新设计的分子如下图9所示,其中有含氧、硫的双环分子。
图9. 使用-ih参数后得到的部分新分子结构
相比于先导化合物,LinkLeadOpt设计出了很多新颖的结构,这些化合物在打分上优于先导化合物,为先导化合物的优化提供了很多新的思路,从而促进项目进度。
3. 总结
在本算例中,我们以6TAM的共晶配体为先导化合物,用LigandScout基于结构的方法识别药效团并搜索ZINC双环片段数据库获得参考分子库,然后用AutoT&T的分子连接策略对先导化合物的双环母核进行骨架跃迁设计新的分子,结果获得172个新的分子。这些新分子相对与先导化合物来说,具有新颖的结构、更好的打分,为先导化合物的优化提供了很多新思路,从而有望促进项目的进度。
在本算例中,采用的Ligandscout与AutoT&T都是成熟的软件,用户界面友好且操作简单,对计算与实验药物化学专家均具有非常实用的价值。
4.相关数据下载
为了方便重复本算例结果,提供了关键的数据:
- 用于LigandScout虚拟筛选用的ZINC15双环化合物数据库:zinc15_bicyclics.ldb.zip
- 用于片段虚拟筛选用的药效团模型:6tam_fragment_vs.pmz
- PDB 6TAM的蛋白结构(用于AutoT&T计算用):6tam_C12G.pdb
- PDB 6TAM的共晶配体(用于AutoT&T计算用):reference.sdf
- 片段虚拟筛选命中的化合物:aromatic-ring-hits.sdf
- 部分LinkLeadOpt骨架跃迁生成的结构:att-core-link-sample.sdf
5. 加下来可以做什么
- 对设计的结构进行力场优化
- 再打分进行优先性排序
- 评估反应速率
- 评估遗传毒性
- 评估构象稳定性
- 不止于片段筛选,对类药分子虚拟筛选结果照样有效
AutoT&T提供了Optimize可以对设计出来的化合物在蛋白的结合位点里进行力场优化,支持AMBER力场与Tripso力场。
如Mortier等人研究[1],可以进一步用分子对接、药效团模型筛选、MMGBSA、FEP等方法对化合物进行打分排序,对化合物进行充分排序。
共价抑制剂与蛋白结合过程分两步:(1)首先形成可逆的初始非共价复合物EI;(2)初始的复合物EI进一步过渡为共价键结合的复合物E-I:
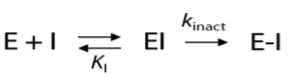
共价抑制剂的酶学活性用二级速率常数kinact/Ki来表示。kinact反映了共价键的形成速率,而Ki反映了通过非共价相互作用形成初始复合物EI的稳定性。第一步过程可以用基于结构的方法(比如分子对接)进行模拟;第二步涉及键的生成与断裂,需要用QM的方法进行模拟。
因此,分子对接、MMGBSA/MMPBSA、FEP等方法预测的结合亲和力(如果不涉及成键反应的话)不能完全决定化合物的酶学活性,还取决于反应速率常数kinact。而共价抑制剂的kinact预测我们已经有详细的描述,可参见:Gaussian教程 | Michael受体与巯基模型的加成反应和反应速率常数的计算
本文的共价抑制剂是个Michael受体,具有潜在遗传毒性风险,我们已经介绍过一个可靠的预测方法:Gaussian教程 | 用DFT预测Michael受体的遗传毒性(AMES test)
虽然一个分子pose的打分很高,但是该构象可能不是能量上合理的。分子对接方法、MMGBSA/MMPBSA、FEP等方法通常对分子的构象采样不全面或者对构象熵估计不足,因此需要额外评估构象的稳定性。我们推荐过三种方法:
基于QM的torsion profile分析:势能面扫描在Torsion Profile中的应用
基于力场的方法:FreeForm–构象稳定性评估及其在先导化合物优化中的应用
基于知识的方法:用Forge或Flare里内置的构象探索工具(Conformation explorer)来评估两面角在CSD数据库里的出现频率,低频的两面角极可能是高风险的构象。
《计算赋能工作流识别全新KRAS G12C共价别构抑制剂》一文所述的虚拟筛选结果也适合于AutoT&T来生成新的化合物。
6. 补充
有读者想看一看有没有更新颖的设计,还真有不少,比如图10所示的4个化合物。

图10. 4个更新颖的设计
7. 文献
- 肖高铿(2021-05-24).墨灵格的博客.计算赋能工作流识别全新KRASG12C共价别构抑制剂
- 肖高铿(2016-06-08).墨灵格的博客.Ligandscout案例–基于药效团片断的虚拟筛选
- 肖高铿(2018-08-31).墨灵格的博客.Ligandscout教程 | 基于片段的虚拟筛选联用AutoT&T进行从头设计
- Ring systems with two internal rings.(2021-06-21访问).Retrieved from http://zinc.docking.org/rings/subsets/double
- Reulecke, I.; Lange, G.; Albrecht, J.; Klein, R.; Rarey, M. Towards an Integrated Description of Hydrogen Bonding and Dehydration: Decreasing False Positives in Virtual Screening with the HYDE Scoring Function. ChemMedChem 2008, 3 (6), 885–897.
- 用Flare在蛋白结合位点里设计分子. https://www.bilibili.com/video/BV11a4y1W7PD
- Li, Y.; Zhao, Y.; Liu, Z.; Wang, R. Automatic Tailoring and Transplanting: A Practical Method That Makes Virtual Screening More Useful. J. Chem. Inf. Model. 2011, 51 (6), 1474–1491. DOI:10.1021/ci200036m
- Li, Y.; Zhao, Z.; Liu, Z.; Su, M.; Wang, R. AutoT&T v.2: An Efficient and Versatile Tool for Lead Structure Generation and Optimization. J. Chem. Inf. Model. 2016, 56 (2), 435–453.DOI:10.1021/acs.jcim.5b00691
- 中科院上海有机化学研究所,王任小等: (a) FWCluster基于骨架的分子结构聚类软件V1.0,登记号:2014SR135139,获批时间:2014.09.09;(b) AT&T先导化合物发现及结构优化软件 v2.0,登记号:2013SR160292,获批时间:2013.12.27; (c) 先导化合物的发现及结构优化软件AT&T v1.0,登记号:2009SR061028,获批时间:2009.12.31.